1
IDENTIFICACIÓN DE
Helicobacter pylori
EN AGUAS
RESIDUALES DEL RÍO ARZOBISPO
LIZETH VIVIANA ALVARADO HUERTAS SANDRA MILENA BUSTOS FUENTES
TRABAJO DE GRADO MICROBIOLOGÍA INDUSTRIAL
PONTIFICIA UNIVERSIDAD JAVERIANA FACULTAD DE CIENCIAS
2
NOTA DE ADVERTENCIA
Artículo 23, Resolución Nº 13 de Julio de 1946
3
IDENTIFICACIÓN DE
Helicobacter pylori
EN AGUAS
RESIDUALES DEL RÍO ARZOBISPO
Lizeth Viviana Alvarado Huertas
Sandra Milena Bustos Fuentes
PONTIFICIA UNIVERSIDAD JAVERIANA FACULTAD DE CIENCIAS
4
IDENTIFICACIÓN DE
Helicobacter pylori
EN AGUAS
RESIDUALES DEL RÍO ARZOBISPO
Lizeth Viviana Alvarado Huertas
Sandra Milena Bustos Fuentes
PONTIFICIA UNIVERSIDAD JAVERIANA FACULTAD DE CIENCIAS
5
IDENTIFICACIÓN DE
Helicobacter pylori
EN AGUAS
RESIDUALES DEL RÍO ARZOBISPO
Lizeth Viviana Alvarado Huertas
Sandra Milena Bustos Fuentes
PONTIFICIA UNIVERSIDAD JAVERIANA FACULTAD DE CIENCIAS
6 Resumen
8
Tabla de contenido
1. Introducción ... 13
2. Justificación ... 15
3. Marco Teórico ... 16
4. Objetivos ... 18
4.1 Objetivo General ... 18
4.2 Objetivos específicos ... 18
5. Metodología ... 19
5.1 Obtencion de muestras ... ¡Error! Marcador no definido.19 5.2 Transporte de muestras ... ,.19
5.3 Procedimiento molecular ... .19
5.3.1 Extraccion de ADN cromosomal ………..…19 5.3.2 Amplificación de los genes ureA, vacA, y cagA………..……20
5.4 Detección de Antígenos fecales ………....22
5.5 Procedimiento microbiológico……….22
5.5.1 Identificación de E. coli………..22
5.5.2 Identificación de anaerobios sulfito reductores………….………...23
5.5.2 Identificación de anaerobios sulfito reductores………….………...23
6. Resultados.………...24
7. Discusión ………..29
8. Conclusiones ... ………..32
9. Recomendaciones ... 33
10.Bibliografía ... 34
9
Índice de tablas
10
Índice de figuras
Figura 1: Electroforesis de la amplificación de los genes vacA, cagAy ureA ..25
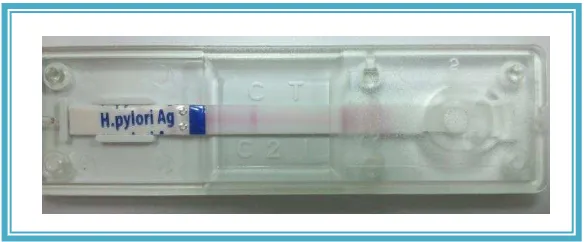
Figura 2: Resultado negativo por inmunocromatografía ………...25
Figura 3: Control Positivo E. coli ……….………...25
Figura 4: Resultado positivo E. coli ………..25
Figura 5: Confirmación prueba positiva E. coli………...25
Figura 6: Control positivo Clostridium perfringes……….………26
11
Índice Anexos
12
Agradecimientos
A nuestras familias que han estado en todo nuestro proceso de formación académica y nos han apoyado incondicionalmente
A Dios porque ha iluminado nuestro camino
A la Pontificia Universidad Javeriana, porque nos ha formado como profesionales íntegros
A la Dra. Alba Alicia Trespalacios, por sus enseñanzas y orientación durante este proceso
A la Dra. Claudia Campos por su asesoría en este trabajo
A nuestros amigos y compañeros que nos acompañaron durante nuestra formación y el desarrollo de este trabajo
13
1.
Introducción
Helicobacter pylori (H. pylori) es una bacteria que infecta la mucosa gástrica causando graves patologías como úlcera y atrofia gástrica, metaplasia intestinal y adenocarcinoma gástrico (1). Cuando la bacteria infecta, produce gastritis crónica que puede progresar a distintas manifestaciones: el 10-15% avanza a ulcera péptica, 1 de cada 10000 personas desarrolla linfomas tipo MALT y del 1-2% adenocarcinoma gástrico que una vez que se presenta causa altos porcentajes de morbimortalidad (2,3).
Se estima que el 50% de la población mundial se encuentra infectada por H. pylori; sin embargo su incidencia está fuertemente ligada a condiciones socioeconómicas, razón por la cual se presenta una prevalencia del 70-90% en países en desarrollo (4,5).
Se conocen distintas vías de transmisión de la bacteria como son: contacto persona a persona, contacto directo con secreciones y oral-fecal. En cuanto a la transmisión oro fecal se ha demostrado la importancia que tiene el agua tratada y no tratada, como vehículo y reservorio del microorganismo (6-9).
14 células epiteliales generando así su muerte y destrucción y por lo tanto un daño grave en el tejido. Al igual que la proteína VacA, la proteína CagA está implicada en un daño más severo al tejido y está relacionado primordialmente con la generación de cáncer gástrico. Siendo estos genes específicos para la identificación del microorganismo y de allí su importancia en este trabajo.
Para la identificación de H pylori en muestras fecales comúnmente se utiliza la inmunocromatografía, un test rápido que se basa en una reacción antígeno – anticuerpo que posee un anticuerpo monoclonal que detecta la bacteria (Anexo A). Teniendo en cuenta que el agua del río Arzobispo se encuentra contaminada con heces fecales y que la prueba presenta una sensibilidad del 100% y una especificidad de 97.8% en muestras de materia fecal, se sugiere que podría ser un buen método de detección de la presencia de la bacteria (14).
15
2.
Justificación
En Colombia el cáncer gástrico es la segunda causa de muerte en hombres y la tercera en mujeres (17). La causa de este cáncer es multifactorial y se sabe que uno de los factores predisponentes más importantes es la infección por H. pylori. Se han descrito distintas vías de transmisión de la bacteria: oral-oral contacto directo con secreciones y oral-fecal. En cuanto a la transmisión oro fecal se ha demostrado la importancia que tiene el agua tratada y no tratada, como vehículo y reservorio del microorganismo (6-9).
Las consecuencias de infecciones causadas por aguas contaminadas son de gran impacto para la población, y es por esta razón que dichas aguas deben ser estudiadas con el fin establecer la supervivencia de microorganismos patógenos en condiciones adversas y determinar si el agua es una fuente importante de infección.
Se han descrito varias metodologías para la identificación de H. pylori, siendo la PCR la más usada debido a su alta sensibilidad, sin embargo presenta la desventaja que pueden haber múltiples interferentes que alteren los resultados de esta prueba. Por esta razón se hace necesario buscar otras alternativas. Los indicadores son un modelo de detección tradicional de patógenos en el agua, debido a esto es importante determinar si hay algún tipo de relación directa entre los indicadores y H. pylori.
16
3.
Marco Teórico
3.1 Helicobacter pylori
H. pylori es una bacteria Gram negativa, microaerofíla, descubierta en 1983 la cual es capaz de vivir en el pH ácido del estómago; es el agente predisponente más importante de cáncer gástrico, induciendo inicialmente una inflamación y ulceración del estómago y el duodeno (18). A nivel mundial se ha clasificado el cáncer gastrointestinal como el segundo más común en el mundo (19). En cuanto al cáncer gástrico asociado a H. pylori, epidemiológicamente hay una recurrencia de infección de 2,27% / año en países desarrollados y del 13,0% /año en países en vía de desarrollo, con una tendencia a aumentar (20).
Cuando la bacteria infecta, produce gastritis crónica que puede progresar a distintas manifestaciones: el 10-15% avanza a ulcera péptica, el 1-2% produce tejido linfoide asociado a adenocarcinoma gástrico (21-22), causando graves problemas de morbimortalidad.
Sobre la trasmisión de H. pylori, varios autores han sugerido que puede ser de diversas formas: por contacto persona a persona, contacto directo con secreciones, oral-fecal y consumo de aguas contaminadas con heces (6-8). Sin embargo H. pylori también se ha encontrado en aguas subterráneas, aguas potables y tratadas indicando que el agua de consumo, también podría ser una importante fuente de infección (9-11)
17 3.2 Técnicas de identificación (PCR y antígenos)
Debido a que no hay una prueba estándar de oro ya que el microorganismo cambia su morfología a una cocoide, se han descrito diferentes estrategias para dicha identificación, entre las que se encuentran el cultivo, PCR, inmunofluorescencia, y FISH. Se ha publicado otro tipo de técnicas que no son muy utilizadas debido a su complejidad y costo, este tipo de técnicas son autoradiografía, ATP bioluminiscencia y la separación inmunomagnética; las cuales son usadas para investigación y no como análisis rutinario. (24)
La PCR es una técnica muy utilizada ya que es altamente específica y sensible y ha permitido la identificación de diferentes microorganismos de una manera rápida y confiable. Es usualmente utilizada como una técnica alterna al medio de cultivo especialmente en microorganismos de difícil crecimiento como H. pylori. También es muy utilizada por la poca cantidad de material genético que se identifica. (25)
La inmunocromatografía es una técnica altamente sensible para el diagnóstico de H. pylori en muestras fecales, comparada con métodos para la identificación clínica como ELISA, cultivo, histopatología y ureasa presenta una sensibilidad del 100% y una especificidad entre 79 y 82% (1,26)
3.3 Análisis indicadores de contaminación fecal
Los indicadores son microorganismos que tienen un comportamiento y concentración similar a la de los patógenos. Se utilizan debido a que su identificación es sencilla, rápida y económica. Un indicador debe cumplir con las siguientes características:
Ser un constituyente normal de la flora intestinal de individuos sanos. Estar presente, de forma exclusiva, en las heces de animales
18 Estar presente cuando los microorganismos patógenos intestinales lo
están.
Presentarse en número elevado, facilitando su aislamiento e identificación.
Debe ser incapaz de reproducirse fuera del intestino de los animales homeotérmicos.
Su tiempo de supervivencia debe ser igual o un poco superior al de las bacterias patógenas.
Debe ser fácil de aislar y cuantificar. No debe ser patógeno (27)
E. coli es un microorganismo cuya presencia refleja contaminación fecal reciente. Por otro lado las esporas de los anaerobios sulfito reductores indican contaminación fecal lejana, debido a la capacidad del microorganismo de esporular (28).
4.
Objetivos
4.1 Objetivo General
Identificar H. pylori en aguas residuales del río Arzobispo 4.2 Objetivos específicos
Identificar los genes ureA, cagA, vacA, de H. pylori en aguas residuales, mediante PCR.
19 Identificar esporas de anaerobios sulfito reductoras y Escherichia coli como
indicador de contaminación fecal para establecer su relación con la presencia de H. pylori en aguas.
5.
Metodología
5.1 Obtención de muestras: Se recolectaron 20 muestras de 700 mL a diferentes condiciones climáticas y a diferentes horas del día en río Arzobispo el cual se caracteriza por poseer contaminación de origen doméstico.
Con el fin de establecer las condiciones ambientales del muestreo y para garantizar la trazabilidad de la muestra se diligenció la cadena de custodia suministrada por el laboratorio de indicadores de calidad de aguas y lodos de la Pontificia Universidad Javeriana.
5.2 Transporte de la muestra: Una vez recolectadas las muestras se mantuvieron en cadena de frío a -4°C hasta su procesamiento, este procedimiento se realizó en base a las especificaciones dadas por la EPA.
5.3 Procedimiento molecular: La identificación de los genotipos cagA, vacA y ureA se realizó en todas las muestras obtenidas según los protocolos estandarizados según el laboratorio de bacteriología especial.
20 procedimiento. A partir de los pellet resultantes se extrajo el ADN utilizando el kit de extracción QIAGEN (Anexo B) a los cuales se les adicionó 20 µL de la proteinasa K, seguido a esto se le adicionó 200 µL del buffer AL y se mezcló en vortex a 2.500 rpm durante 15 segundos. Posteriormente se incubó a 56°C durante 10 minutos. Se adicionaron 200 µL de etanol al 96% y se mezcló en vortex a 2.500 rpm durante 15 segundos. La mezcla se transfirió a las columnas suministradas por el kit y se centrifugó a 8.000 rpm durante 1 minuto. Se descartó el filtrado. A continuación se adicionó 500 µL de buffer AW1 y se centrifugó a 8.000 rpm durante 1 minuto. Se descartó el filtrado. Enseguida se adicionó el buffer AW2 y se centrifugó a 14.000 rpm durante 3 minutos. Se hizo una nueva centrifugación a 18.000 rpm durante 1 minuto. Finalmente se adicionó 200 µL de buffer AE, se incubó a temperatura ambiente y se centrifugo 8.000 rpm durante 1 minuto. El ADN se almacenó a 4°C para la realización de la PCR.
21 Tabla 1: Componentes de la PCR
Reactivo urea / cagA /vacA s1/s2
Taq polimerasa 5 µL
Cebador forward 0.25 µL
Cebador reverse 0.25 µL
[image:21.792.31.766.261.477.2]DNA 4.5 µL
Tabla 2: Condiciones de la PCR
GEN CEBADORES FORWARD/ REVERSE DENATURACION
INICIAL DENATURACION HIBRIDACION EXTENSIÓN EXTENSIÓN FINAL CICLOS
TAMAÑO (pb)
ureA
5’ GGG TAT TGA AGC GAT GTT TCC T 3´
94°C
2 min 30 seg 94°C 20 seg 58°C 30 seg 72°C 5 min 72°C 40 214
5’ GCT TTT TTG CCT TCG TTG ATA GT 3´
vacA
s1/s2
5’ ATG GAA ATA CAA CAA ACA CAC 3’
95°C
3 min 1 min 95°C 1 min 50°C 1 min 72°C 5 min 72°C 35 s1: 259 s2: 286
5’ CTG CTT GAA TGC GCC AAA C 3’
cagA
5’ TTG ACC AAC AAC CAC AAA CCG AAG 3’
95°C
2 min 1 min 95°C 1 min 50°C 1 min 74°C 5 min 74°C 40 183
22 5.4 Detección de antígenos fecales: Se utilizó el kit de BioLine SD Ag H. pylori para la detección del microorganismo a partir de las muestras recolectadas.
Para aumentar la probabilidad de identificar H. pylori en aguas residuales se hizo una concentración de la muestra; sin embargo también se realizó la prueba a partir de muestras sin concentrar para determinar variabilidad de los resultados.
1. Se centrifugó 1.5 mL de agua, previamente homogenizadas, a 8.000 rpm, se descartó el sobrenadante y se realizó por segunda vez dicho procedimiento. Posteriormente el pellet se transfirió al pozo del cassette. Este procedimiento se realizó en 10 muestras recolectadas.
2. A partir de la muestra recolectada y previamente homogenizada, se adicionaron 100 µL al pozo del cassette. Este procedimiento se realizó en 10 muestras recolectadas.
Para interpretar los resultados se esperó 15 minutos. Finalmente para corroborar que el montaje de la prueba haya sido el adecuado se observó que el control de la misma fuera positivo.
5.5 Análisis de E. coli y esporas de anaerobios sulfito reductores
5.5.1 Identificación de E. coli: Este análisis se realizó por el método de filtración en membrana siguiendo el protocolo ISO 9308-1(2000)
23 5.5.2 Identificación de esporas de anaerobios sulfito reductores: Este análisis de realizó por el método de filtración de membrana siguiendo el protocolo ISO 6461-2 (1986).
Inicialmente la muestra fue homogenizada sometida a 75°C durante 15 minutos, seguido a esto se realizaron tres diluciones de la muestra y se pasó a montar la rampa de filtración a cual se le adicionó 20 ml de solución tamponada estéril, y posteriormente se filtró 1 mL utilizando una membrana estéril de 0.22 µm, seguido a esto el filtro se sobrepuso en un medio de cultivo SPS, y se llevaron a incubación durante 24 horas a 44°C en condiciones de anaerobiosis; pasado este tiempo se interpretaron los resultados (Anexo E) ( Las colonias presuntivas obtenidas se confirmaron con coloración de Gram )
5.5.3 Relación estadística entre la prueba molecular y la prueba de identificación de indicadores de aguas
24
6.
Resultados:
Los resultados se dividieron en tres partes, la primera describe la amplificación de los tres genes evaluados, la segunda hace referencia a los resultados obtenidos por la técnica de inmunocromatografía y la tercera se refiere a los resultados de los indicadores fecales.
Para el gen vacA se obtuvieron 8 muestras positivas de las 20 analizadas, al igual que el gen ureA; mientras que el gen cagA mostró una sola muestra positiva de las 20 que fueron recolectadas y procesadas. En la Figura 1 se puede observar el gel de electroforesis para cada uno de los genes.
Por otro lado para la prueba de antígenos fecales todos los resultados fueron negativos. Se debe tener en cuenta que el control fue positivo para todas las muestras por lo cual se corroboró el buen funcionamiento de la prueba. (Figura 2). Finalmente lo recuentos de E. coli como indicador de contaminación fueron positivos para todas las muestras (Figura 5), estas muestras positivas fueron confirmadas utilizando el reactivo de Kovac´s para observar si el indol era positivo o no (Figura 6), sin embargo el recuento de esporas de anaerobios sulfito reductores fuer positivo para 13 de las muestras. (Figura 7).
Se realizó una prueba estadística para realizar un análisis descriptivo utilizando el programa estadístico SPSS se relacionó la prueba molecular con cada uno de los indicadores ( E. coli y esporas de anaerobios sulfito reductores), en donde se obtuvo una relación del 60% de la presencia de H. pylori con la de E. coli y del 69% de la presencia de H. pylori con la de anaerobios sulfito reductores; y posteriormente haciendo uso del mismo programa se realizó un análisis de concordancia entre las pruebas mediante el hallazgo del índice Kappa en donde se obtuvo un índice de concordancia de 0.255, lo que indica que es una concordancia débil entre las dos pruebas.
25
Figura 1 Electroforesis de la amplificacion de vacA, cagA y ureA: Gen vacACarril 1, 21, 41 61: Patrón de peso
molecular, Carril 2: Blanco, Carril 3: Control Positvo NCTC 1137, Carril 4-24: Muestras del río arzobispo. Gen
cagA Carril 25: Blanco, Carril 26: Control Positvo NTCC 1137, Carril 27-47: Muestras del río arzobispo, Gen
ureACarril 48: Blanco, Carril 49: Control Positvo NTCC 1137 Carril 50-70: Muestras del río arzobispo 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
21 22 23 24 25 26 2728 29 30 31 32 33 34 35 36 37 38 39 40
41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60
61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 vacA
s1: 259
s2: 286
cagA
183 pb
ureA
26 Figura 2.Resultado negativo por inmunocromatografía
Figura 3. Control positivo E. coli
27
Figura 6. Control positivo Clostridium perfringes Figura 7. Resultado positivo Clostridium perfringes
28 Tabla 3. Resultados de los tres procedimientos en las 20 muestras
Muestra ureA Gen vacA Gen cagA Gen Antígenos fecales E. coli Anaerobios
1 Negativo Negativo Negativo Negativo 7 x 10 3
UFC/ml < 1000 UFC/ ml 2 Negativo Negativo Negativo Negativo 1,3 x 104
UFC/ml 1 x 10 3 UFC/ml 3 Positivo Negativo Negativo Negativo 1,3 x 104
UFC/ml 2 x 10 3 UFC/ml 4 Positivo Negativo Negativo Negativo 8 x 10 3
UFC/ml 1 x 10 3 UFC/ml 5 Negativo Negativo Negativo Negativo 3 x 10 3
UFC/ml < 1000 UFC/ ml 6 Negativo Negativo Negativo Negativo 4 x 10 3
UFC/ml < 1000 UFC/ ml 7 Positivo Negativo Negativo Negativo 1,4 x 104
UFC/ml 4 x 10 3 UFC/ml 8 Negativo Negativo Negativo Negativo 1,2 x 104
UFC/ml 1 x 10 3 UFC/ml 9 Negativo Positivo Positivo Negativo 1,8 x 104
UFC/ml 2 x 10 3 UFC/ml 10 Positivo Positivo Negativo Negativo 2 x 10 3
UFC/ml < 1000 UFC/ ml 11 Positivo Positivo Negativo Negativo 1,1 x 104
UFC/ml < 1000 UFC/ ml 12 Negativo Negativo Negativo Negativo 2,0 x 104
UFC/ml 3 x 10 3 UFC/ml 13 Negativo Positivo Negativo Negativo 3 x 10 3
UFC/ml < 1000 UFC/ ml 14 Negativo Positivo Negativo Negativo 1,2 x 104
UFC/ml 1 x 10 3 UFC/ml 15 Negativo Positivo Negativo Negativo 1,5 x 104
UFC/ml 3 x 10 3 UFC/ml 16 Positivo Negativo Negativo Negativo 1,4 x 104
UFC/ml 4 x 10 3 UFC/ml 17 Negativo Negativo Negativo Negativo 2,3x 104
UFC/ml 6 x 10 3 UFC/ml 18 Positivo Positivo Negativo Negativo 2 x 10 3
UFC/ml 1 x 10 3 UFC/ml 19 Negativo Negativo Negativo Negativo 1 x 10 3
UFC/ml < 1000 UFC/ ml 20 Positivo Positivo Negativo Negativo 3 x 10 3
UFC/ml 1 x 10 3 UFC/ml
29
7.
Discusión
H. pylori es un microorganismo de crecimiento exigente por lo que se han diseñado diferentes estrategias más rápidas y sensibles para su identificación. En este trabajo se tomaron 20 muestras con el fin de evaluar tres procedimientos diferentes uno molecular, uno inmunológico y uno microbiológico para determinar cuál de estos tres tiene una mayor sensibilidad durante su identificación.
Los resultados obtenidos para el procedimiento molecular (PCR) muestran resultados positivos para el gen ureA en 8 de las 20 muestras procesadas fueron positivas. Moreno et al. (2003) recolectó 6 muestras del río Turia en España, las cuales fueron sometidas a una pre enriquecimiento previo a la extracción del ADN, encontrando que para la PCR, 2 de las muestras fueron positivas (9). Hay que tener en cuenta que el pre-enriquecimiento se hace para aumentar la probabilidad que la PCR sea positiva, sin embargo en el presente estudio se omitió este procedimiento ya que se quería evaluar si no haciendo pre-enriquecimiento también era posible encontrar el microorganismo y se obtuvo una mayor identificación del mismo.
En un estudio hecho por Queralt et al. (2005) y colaboradores en el que obtuvieron 2 de 38 muestras positivas para el gen ureA, hicieron una extracción de ADN en el que utilizaron partículas de sílica con el fin de remover interferentes de la PCR y de esta manera aumentar la posibilidad de amplificar el gen de interés (10). En este trabajo no se hicieron modificaciones para la extracción y aun así se obtuvo un número mayor de muestras amplificadas.
Las diferencias entre los resultados de la literatura y del presente estudio probablemente se deban a las condiciones de la toma de la muestra, factores climáticos, inhibidores de la PCR, concentración de materia orgánica, entre otros elementos que pueden generar cambios en los mismos.
30 analizadas fueron positivas, los investigadores concluyen que esto se debe a una baja concentración del microorganismo en las muestras procesadas (34); sin embargo en el año 2007 Nayak et al; realizó la identificación de H. pylori en aguas residuales, mediante una PCR en tiempo real evaluando la presencia del gen vacA; en donde se obtuvieron 20 muestras positivas de las 23 que entraron al estudio. Probablemente esto se deba a que antes de la extracción de ADN se realizó una concentración de la muestra 1:10 por medio de una centrifugación (35). En este trabajo se obtuvieron 8 muestras positivas de las 20 procesadas y al igual que la investigación de Nayak se realizó una concentración del ADN, razón por la cual probablemente aumentó el número de muestras positivas para este gen. Este gen es de gran importancia ya que este se encuentra en todas las cepas de H. pylori y corrobora su identificación.
Yañez et al, (2009), realizó una identificación el cagA en donde analizó agua de una planta de tratamiento, de un río y de agua de consumo, los resultados obtenidos fueron: de la planta de tratamiento 3 muestras de 40 fueron positivas, en el agua de río ninguna fue positiva de las 19 muestras analizadas y en el agua potable todos los resultados fueron negativos para 10 muestras procesadas (13). En este trabajo 1 de las 20 muestras analizadas fue positiva para el gen cagA. Este bajo número de muestras positivas en ambos estudios posiblemente se deba a que solo el 50 – 70% de las cepas de H. pylori poseen este gen (36)
31 por la baja concentración en las muestras recolectadas, por lo cual las estrategias de concentración de la muestras podrían sugerirse para evaluar si éstas mejoran la detección del microorganismo en el agua.
Queralt et al, (2005) evaluó E. coli como indicador de contaminación fecal en aguas obteniendo recuentos superiores a 107 UFC/ mL (10), resultado que difiere con el de esta investigación en el que se encontró un recuento entre 103 y 104 UFC/ml en todas las muestras analizadas. Esta diferencia probablemente se debe a que en el trabajo realizado en el 2005 pudo haberse trabajado con aguas más contaminadas que las procesadas en el presente estudio donde las muestras se recolectaron en temporada de lluvia, diluyendo así la muestra.
32 ya que se ha buscado relación únicamente con coliformes totales, coliformes fecales y E. coli (10)
Es difícil establecer una relación entre los microorganismos indicadores y los patógenos como H. pylori, teniendo en cuenta que no se ha descrito una caracterización físico-química previa del agua en ninguna de las investigaciones reportadas (9,10).
8.
Conclusiones
1. De las técnicas evaluadas la PCR fue la que tuvo mayor sensibilidad para la identificación de H. pylori en aguas residuales del rio Arzobispo.
2. La inmunocromatografía es una técnica que presenta una sensibilidad y especificidad cercana al 100% en muestras de materia fecal sin embargo su límite de detección no fue suficiente para identificar H. pylori en aguas residuales del rio Arzobispo.
33
9.
Recomendaciones
Debido a que somos pioneras en este tipo de proyectos recomendamos:
1. Hacer una evaluación física química del agua previa a ser procesada.
2. Evaluar la PCR y la inmunocromatografía realizando un pre-enriquecimiento previo, con el fin de aumentar la probabilidad de encontrar el microorganismo
3. Realizar estudios de identificación de H. pylori en aguas en temporadas de sequía y comparar con los resultados obtenidos en este estudio en donde las muestras fueron obtenidas en temporada de lluvia.
34
10.
Bibliografía
1. Sato M, Shimoyama T, Takahashi R, Kajiyama H, Sano Y, Sakaedani N, et al. Characterization and usefulness of stool antigen tests using a monoclonal antibody to Helicobacter pylori catalase. J Gastroenterol Hepatol 2012; 27(3):23-28
2. Parsonnet J, Friedman GD, Vandersteen DP, Chang Y, Vogelman JH, Orentreich N, et al.Helicobacter pylori infection and the risk of gastric
carcinoma. N Engl J Med 1991;325: 1127–31.
3. Ernst PB, Gold BD. The disease spectrum of Helicobacter pylori: the
immunopathogenesis of gastroduodenal ulcer and gastric cancer. Annu Rev Microbiol 2000;54:615–40.
4. Boyanova L, Mitov I. Geographic map and evolution of primary Helicobacter pylori resistance to antibacterial agents. Expert Reviews. Anti Infect Ther. 2010; 8:59-70.
5. Correa P, Piazuelo MB. Evolutionary History of the Helicobacter pylori Genome: Implications for Gastric Carcinogenesis. Gut and Liver. 2012;6(1):21–8.
35 7. Brown LM (2000) Helicobacter pylori: epidemiology and routes of transmission.
Epidemiol Rev 22: 283.
8. Magalhães Queiroz DM, Luzza F. Epidemiology of Helicobacter pylori infection. Helicobacter 2006 10;11 Suppl 1:1-5.
9. Moreno Y, Ferrús MA, Alonso JL, Jiménez A, Hernández J. Use of fluorescent in situ hybridization to evidence the presence of Helicobacter pylori in water. Water Res 2003 5;37(9):2251-2256.
10. Queralt N, Bartolomé R, Araujo R. Detection of Helicobacter pylori DNA in human faeces and water with different levels of faecal pollution in the north-east of Spain. Journal of Applied Microbiology. 2005 Jan;98(4):889–95.
11. Benson JA, Fode-Vaughan KA, Collins MLP. Detection of Helicobacter pylori in water by direct PCR. Letters in Applied Microbiology. 2004 Jan;39(3):221–5.
12. Janzon A, Sjöling A, Lothigius A, Ahmed D, Qadri F, Svennerholm A-M. Failure to detect Helicobacter pylori DNA in drinking and environmental water in Dhaka, Bangladesh, using highly sensitive real-time PCR assays. Applied and Environmental Microbiology. 2009 May;75(10):3039–944.
13. Yáñez MA, Barberá VM, Soria E, Catalán V. Quantitative detection of Helicobacter pylori in water samples by real-time PCR amplification of the cag pathogenicity island gene, cagE. Journal of Applied Microbiology. 2009 Aug;107(2):416–24.
36 15. Campos C. Indicadores de contaminación fecal en la reutilización de aguas
residuales para riego agrícola. Tesis doctoral. Facultad de Biología. Universidad de Barcelona. 1999. 250 p.
16. Piñeros M, Ferlay J, Murillo R. Cancer incidence estimates at the national and district levels in Colombia. Salud Publica Mex 2006; 48: 455-465.
17. Handa O, Naito Y, Yoshikawa T. Redox biology and gastric carcinogenesis: the role of Helicobacter pylori. Redox Rep 2011;16(1):1-7.
18. Saxena A, Shukla S, Prasad KN, Ghoshal UC. Virulence attributes of Helicobacter pylori isolates & their association with gastroduodenal disease. Indian J Med Res 2011 05;133(5):514-520.
19. Buzás GM. [Helicobacter pylori -- 2010]. Orv Hetil 2010 12/05;151(49):2003-2010.
20. Parsonnet J, Friedman GD, Vandersteen DP, Chang Y, Vogelman JH, Orentreich N, et al. Helicobacter pylori infection and the risk of gastric carcinoma. N Engl J Med 1991;325: 1127–31.
21. Ernst PB, Gold BD. The disease spectrum of Helicobacter pylori: the immunopathogenesis of gastroduodenal ulcer and gastric cancer. Annu Rev Microbiol 2000;54:615–40.
37 23. Travis PB, Goodman KJ, O'Rourke KM, Groves FD, Sinha D, Nicholas JS,
VanDerslice J, Lackland D, Mena KD (2010) The association of drinking water quality and sewage disposal with Helicobacter pylori incidence in infants: the potential role of water-borne transmission. J Water Health 8: 192-203.
24. Premoli G, González A, Millan B, Percoco T, Vielma A Diagnóstico de Helicobacter pylori mediante la reacción en cadena de la polimerasa. Revista Cubana Medica Tropical 2004;56(2):85-90
25. Wu D, Wu I-, Wang S, Lu C, Ke H, Yuan SF, et al. Comparison of stool enzyme immunoassay and immunochromatographic method for detecting Helicobacter pylori antigens before and after eradication. Diagn Microbiol Infect Dis 2006 12;56(4):373-378.
26. Suhaila N, Hussin S, Rahman MM. Comparative efficacy sensitivity and specificity of the tests used for the diagnosis of Helicobacter pylori. Pak J Biol Sci 2010;13(21):1057-1061.
27. Arcos M, Avila S, Estupiñan S. Gomez G. Indicadores microbiológicos de contaminación de las fuentes de agua. Nova 2005:3(4):69-79
28. Moreno Y, Ferrus M.Specific detection of cultivable Helicobacter pylori cells from wastewater treatment plants. Helicobacter; 17 (5):327-332
38 30. C.H. Johnson, E.W. Rice, D.J. Reasoner Inactivation of Helicobacter pylori
by Chlorination Appl. Environ. Microbiol., 63 (1997), pp. 4969–4970
31. A.P. West, M.R. Millar, D.S. Tompkins Effect of physical environment on survival of Helicobacter Pylori J. Clin. Pathol., 45 (1992), pp. 228–231
32. Park SR, Mackay WG, Reid DC. Helicobacter sp. recovered from drinking water biofilm sampled from a water distribution system. Water Res 2001 4;35(6):1624-1626.
33. Fernández A, Molina M, Alvarez A, Alcántara M, Espigares A. Transmisión fecohÌdrica y virus de la hepatitis A. Higiene y Sanidad Ambiental 2001; 1: 8-24.
34. Shannon KE, Lee D-, Trevors JT, Beaudette LA. Application of real-time quantitative PCR for the detection of selected bacterial pathogens during municipal wastewater treatment. Sci Total Environ 2007 8/15;382(1):121-129.
35. Nayak AK, Rose JB. Detection of Helicobacter pylori in sewage and water using a new quantitative PCR method with SYBR green. Journal of Applied Microbiology. 2007 Nov;103(5):1931–41.
39 37. Baker, K.H. and Hegarty, J.P. (2001) Presence of Helicobacter pylori In drinking water is associated with clinical infection.ScandinavianJournal of Infectious Diseases33, 744–746
11.
Anexos
Anexo A: Fundamento de la inmunocromatografía
Se trata de una técnica rápida de detección cualitativa de antígenos de H. pylori. La muestra de materia fecal se diluye y posteriormente se adicionan 3 gotas en la ventana del cassette. La muestra corre por la tira y se encuentra con la zona (T), que tiene anticuerpos monoclonales específicos contra H. pylori conjugado con oro coloidal, si la muestra contiene el antígeno se unirá al anticuerpo fijado y se formara una línea en el cuadro de resultados. La muestra y el conjugado siguen corriendo hasta la línea de control en donde hay fijos anticuerpos anti-conjugado que validan la prueba al aparecer una línea en este recuadro.
40 Anexo B: Fundamento proteinasa K (Kit de extracción QIAGEN)
41 Anexo C: Fundamento y componentes del medio Chromocoult
Fundamento:
El sustrato X-glucurónido se utiliza para la identificación de ß-D-glucuronidasa que es característico de E.coli escinde tanto Salmon-GAL y X-glucurónido, por lo que colonias positivas adquieren un color azul oscuro al color violeta.
Estos se distinguen fácilmente de las otras colonias de coliformes que tienen un salmón al color rojo. La inclusión de triptófano mejora la reacción indol, aumentando de este modo fiabilidad de la detección cuando se utiliza en combinación con la Salmón-GAL y X-glucurónido de reacción como parte de un la confirmación adicional para E. coli.
Componentes del medio:
Peptona 3.0 g/L
Cloruro de sodio 5.0g/L
Fosfato de sodio dihidrogenado2.2g/L Fosfato de hidrogeno disodico 2.7g/L Piruvato de sodio 1.0g/L
Triptofano1.0g/L Agar-agar 10.0 g/L Sorbitol 1.0g/L Tergitol®7 0.15g/L
Mezcla cromogénica 0.4.g/L
42 Anexo D: Fundamento y componentes del reactivo de Kovac´s
Fundamento:
Mediante esta prueba se detecta la liberación de indol en un cultivo bacteriano. Esta liberación se debe a la degradación del triptófano por medio de la enzima triptofanasa.
Componentes:
Paradimetil amino benzaldehído Acido Clorhídrico
43 Anexo E: Fundamento y componentes del medio SPS
Fundamento:
La mayor parte de los Clostridios reducen el sulfito a sulfuro, que se puede poner de manifiesto por el color negroproducido con los iones de hierro. El Sulfato de Polimixinay la Sulfadiazina inhiben el crecimiento de la flora acompañante y la peptona de caseína y el extracto de levadura representan el aporte de nutrientes
Componentes del medio:
Sodio Sulfito 0,5g/L
Sulfato de Polimixina B0,01g/L Sulfadiazina0,12g/L
Peptona de Caseína15,0g/L Extracto de Levadura10,0g/L Hierro(III) Citrato0,5g/L Agar13,0g/L
44
Anexo No. 6
Doctor(a)
Janeth del Carmen Arias Palacios
Director(a) Carrera de Microbiología Industrial Facultad de Ciencias
Respetado(a) Doctor(a):
Con la presente comunicación, hacemos constar que el trabajo de grado titulado Identificación de Helicobacter pylori en aguas residuales del rio Arzobispo, realizado por las estudiantes Lizeth Viviana Alvarado Huertas y Sandra Milena Bustos Fuentes, ha sido revisado y corregido de acuerdo con las observaciones sugeridas por los jurados en la sustentación.
En constancia se firma, a los veinticuatro (24) días del mes de Junio del año 2014.
45 ANEXO 2
CARTA DE AUTORIZACIÓN DE LOS AUTORES
(Licencia de uso)
Bogotá, D.C., Junio 24 de 2014
Señores
Biblioteca Alfonso Borrero Cabal S.J.
Pontificia Universidad Javeriana
Cuidad
Los suscritos:
Lizeth Viviana Alvarado Huertas , con C.C. No 1022361039
Sandra Milena Bustos Fuentes , con C.C. No 1026274086
, con C.C. No
En mi (nuestra) calidad de autor (es) exclusivo (s) de la obra titulada:
Identificación de Helicobacter pylori en aguas residuales del rio Arzobispo
(por favor señale con una “x” las opciones que apliquen)
Tesis doctoral Trabajo de grado x Premio o distinción: Si No x
cual:
presentado y aprobado en el año , por medio del presente escrito autorizo
46
En consecuencia, las atribuciones de usos temporales y parciales que por virtud de la presente licencia se autorizan a la Pontificia Universidad Javeriana, a los usuarios de la Biblioteca Alfonso Borrero Cabal S.J., así como a los usuarios de las redes, bases de datos y demás sitios web con los que la Universidad tenga perfeccionado un convenio, son:
AUTORIZO (AUTORIZAMOS) SI NO
1. La conservación de los ejemplares necesarios en la sala de tesis y trabajos
de grado de la Biblioteca.
X
2. La consulta física (sólo en las instalaciones de la Biblioteca) X
3. La consulta electrónica – on line (a través del catálogo Biblos y el
Repositorio Institucional)
X
4. La reproducción por cualquier formato conocido o por conocer X
5. La comunicación pública por cualquier procedimiento o medio físico o
electrónico, así como su puesta a disposición en Internet
X
6. La inclusión en bases de datos y en sitios web sean éstos onerosos o
gratuitos, existiendo con ellos previo convenio perfeccionado con la Pontificia Universidad Javeriana para efectos de satisfacer los fines previstos. En este evento, tales sitios y sus usuarios tendrán las mismas facultades que las aquí concedidas con las mismas limitaciones y condiciones
X
De acuerdo con la naturaleza del uso concedido, la presente licencia parcial se otorga a título gratuito por el máximo tiempo legal colombiano, con el propósito de que en dicho lapso mi (nuestra) obra sea explotada en las condiciones aquí estipuladas y para los fines indicados, respetando siempre la titularidad de los derechos patrimoniales y morales correspondientes, de acuerdo con los usos honrados, de manera proporcional y justificada a la finalidad perseguida, sin ánimo de lucro ni de comercialización.
47
Sin perjuicio de los usos y atribuciones otorgadas en virtud de este documento, continuaré (continuaremos) conservando los correspondientes derechos patrimoniales sin modificación o restricción alguna, puesto que de acuerdo con la legislación colombiana aplicable, el presente es un acuerdo jurídico que en ningún caso conlleva la enajenación de los derechos patrimoniales derivados del régimen del Derecho de Autor.
De conformidad con lo establecido en el artículo 30 de la Ley 23 de 1982 y el artículo 11 de la
Decisión Andina 351 de 1993, “Los derechos morales sobre el trabajo son propiedad de los
autores”, los cuales son irrenunciables, imprescriptibles, inembargables e inalienables. En consecuencia, la Pontificia Universidad Javeriana está en la obligación de RESPETARLOS Y HACERLOS RESPETAR, para lo cual tomará las medidas correspondientes para garantizar su observancia.
NOTA: Información Confidencial:
Esta Tesis o Trabajo de Grado contiene información privilegiada, estratégica, secreta, confidencial y demás similar, o hace parte de una investigación que se adelanta y cuyos
resultados finales no se han publicado. Si No x
En caso afirmativo expresamente indicaré (indicaremos), en carta adjunta, tal situación con el fin de que se mantenga la restricción de acceso.
NOMBRE COMPLETO No. del documento
de identidad FIRMA
FACULTAD: CIENCIAS
48 ANEXO 3
BIBLIOTECA ALFONSO BORRERO CABAL, S.J. DESCRIPCIÓN DE LA TESIS O DEL TRABAJO DE GRADO
FORMULARIO
TÍTULO COMPLETO DE LA TESIS DOCTORAL O TRABAJO DE GRADO
Identificación de Helicobacter pylori en aguas residuales del rio Arzobispo
SUBTÍTULO, SI LO TIENE
AUTOR O AUTORES
Apellidos Completos Nombres Completos
Alvarado Huertas Lizeth Viviana
Bustos Fuentes Sandra Milena
DIRECTOR (ES) TESIS O DEL TRABAJO DE GRADO
Apellidos Completos Nombres Completos
Trespalacios Rangel Alba Alicia
Campos Pinilla Maria Claudia
FACULTAD
Ciencias
PROGRAMA ACADÉMICO
Tipo de programa ( seleccione con “x” )
Pregrado Especialización Maestría Doctorado
x
49 Microbiología Industrial
Nombres y apellidos del director del programa académico
Janeth del Carmen Arias Palacios
TRABAJO PARA OPTAR AL TÍTULO DE:
Microbiólogas Industriales y Bacteriólogas
PREMIO O DISTINCIÓN(En caso de ser LAUREADAS o tener una mención especial):
CIUDAD AÑO DE PRESENTACIÓN DE LA TESIS O DEL TRABAJO DE GRADO
NÚMERO DE PÁGINAS
Bogotá D.C. 2013 43
TIPO DE ILUSTRACIONES ( seleccione con “x” )
Dibujos Pinturas Tablas, gráficos y
diagramas Planos Mapas Fotografías Partituras
X X
SOFTWARE REQUERIDO O ESPECIALIZADO PARA LA LECTURA DEL DOCUMENTO
Nota: En caso de que el software (programa especializado requerido) no se encuentre licenciado por la Universidad a través de la Biblioteca (previa consulta al estudiante), el texto de la Tesis o Trabajo de Grado quedará solamente en formato PDF.
MATERIAL ACOMPAÑANTE
TIPO
[image:49.612.78.550.67.474.2]50
(minutos) CD DVD Otro ¿Cuál?
Vídeo
Audio
Multimedia
Producción electrónica
Otro Cuál?
DESCRIPTORES O PALABRAS CLAVE EN ESPAÑOL E INGLÉS
Son los términos que definen los temas que identifican el contenido. (En caso de duda para designar
estos descriptores, se recomienda consultar con la Sección de Desarrollo de Colecciones de la Biblioteca Alfonso Borrero Cabal S.J en el correo biblioteca@javeriana.edu.co, donde se les orientará).
ESPAÑOL INGLÉS
RESUMEN DEL CONTENIDO EN ESPAÑOL E INGLÉS
(Máximo 250 palabras - 1530 caracteres)