Em
abierta
aliiernpo
UNIVERSIDAD AUTÓNOMA METRLPOLITANA
Unidad Iztapalapa
DEPARTAMENTO DE QU~MICA
'DMSIÓN DE CIENCIAS BÁSICAS E INGENIE&
/
Estudio de Desnaturalización de
quimopapaína
por
medio de fluorescencia
PROYECTO TERMINAL DE LA~ICENCIATURA
EN QUIMICA
PRESENTA
'JOSE ARELLANO COBIAN
Este trabajo se realizó,
bajo la dirección de la Dra. Silvia Solís
Mendiola, en el &ea de Biofisicoquímica, Departamento de
1. INTRODUCCION 1.1 Estructura Nativa 1.2 Desnaturalización
1.3 Dicroísmo circular 1.4 Fluorescencia
1.5 Estabilidad de proteínas y la radiación UV 1.6 Quimopapaína
1.7 Objetivo
2. METODOS EXPERIMENTALES 2.1 Materiales
2.2 Dicroísmo circular
2.3 Emisión de fluorescencia
3. RESULTADOS Y CONCLUSIONES
3.1 Constantes de velocidad 3.2 Fotooxidación
3.3 Conclusiones
1. INTRODUCCION
1 .l Estructura nativa
La función biológica de una proteína está determinada por su
estructura, en la cual se definen cuatro niveles o tipos de organización: primaria. secundaria, terciaria y cuaternaria.
La estructura primaria
se
refierea
la secuencia de aminoácidos en la cadena polipeptídica. Puesto que el número de aminoácidos diferentes quepueden aparecer en dicha cadena es de 20, el número de posibles
combinaciones en una secuencia lineal es extremadamente elevado. Estos
aminoácidos tienen en su cadena lateral diversos grupos funcionales, que
pueden diferir en su carácter aromático o alifático, así como en su carga eléctrica, volumen y polaridad (Lehninger et ai., 1993; Bohinski, 1978).
La estructura secundaria consiste en conformaciones regulares y
repetitivas de aminoácidos en distintas secciones de la cadena, en donde los ángulos diedros de la cadena principal tienen valores bien determinados. Los enlaces de hidrógeno entre los átomos de los grupos
amida y de los grupos laterales pueden hacer que la cadena de
aminoácidos se pliegue en el espacio y adopte configuraciones estables. Las estructuras secundarias más comunes son las hélices a y las hojas
p.
En la primera de éstas, la cadena se enrolla en sentido dextrógiro y los
grupos laterales se colocan en el exterior de la hélice. En la segunda, la
cadena de aminoácidos adopta una conformación en zig-zag, debido a los
puentes de hidrógeno que se establecen entre secciones distintas de la
cadena principal. Si bien son posibles otros tipos de estructura secundarias
(como los giros), las dos mencionadas anteriormente son las más comunes,
ya que, por una parte, son estabilizadas por puentes de hidrógeno y por
La estructura terciaria se refiere a la manera en que la cadena
polipeptídica se pliega en el espacio tridimensional, acomodando las
estructura secundarias y las secuencias aleatorias que las unen en una
configuración estrechamente plegada y, en el caso de las proteínas
globulares, compacta. La mayoría de los residuos hidrofóbicos se orientan hacia el interior de la proteína y la mayoría de los polares hacia el exterior, es decir, hacia el solvente acuoso La estructura cuaternaria se refiere al modo en que se ubican en el espacio la cadenas polipeptídicas de las proteínas que constan de más de una cadena entre dichas cadenas puede
no haber enlaces covalentes. Las estructuras terciaria y cuaternaria se
estabilizan por enlaces químicos, fundamentalmente de naturaleza no
covalente.
Las proteínas tienden a adoptar, bajo ciertas condiciones, una
estructura específica singular, llamada estructura nativa, aunque, en
principio, sea posible un número muy elevado de configuraciones
tridimensionales para una secuencia determinada de aminoácidos. La
información codificada en dicha secuencia y en las condiciones del solvente son suficientes para dirigir el plegamiento de la cadena polipeptídica
(Jaenicke, 1993).
Hasta la fecha no se ha comprendido a un nivel molecular la relación precisa entre secuencia y estructura tridimensional. Se sabe, sin embargo,
que el proceso de plegamiento debe estar unívocamente definido, pues sólo
un sistema con una conformación tridimensional claramente definida -y, a la
vez, flexible- la estructura nativa, puede desempeñar funciones tan
delicadas y específicas como las de las proteínas (Schmid, 1992).
El estudio de las interacciones intramoleculares puede conducir a la comprensión de la estructura de la proteína y de la forma en que los
componentes
de
un polipéptido se ensamblan en la proteína nativa. Esteúltimo sistema ha sido definido como el estado de energía libre mínima (esto
que los estados desplegado y nativo existen en equilibrio termodinámico
dentro de la transición de desdoblamiento, este estado nativo
es
el mismo que se encuentra en condiciones fisiológicas, como temperatura cercana ala ambiente, pH neutro y ausencia de agentes desestabilizantes. Por otra
parte, se ha argumentado que, desde un punto de vista biológico, es
importante alcanzar la estructura nativa en un tiempo relativamente corto.
Ambos puntos de vista pueden ser conciliadas si se toma en cuenta la posibilidad de que haya procesos de plegamiento ordenados que, por una
sucesión de estados intermedios específicos, conduzcan a la formación
rápida del estado nativo termodinámicamente más favorecido. De acuerdo
con lo que se ha llamado "principio de consistencia cinética" (Go, 1983), las
rutas de plegamiento favorecidas cinéticamente conducen al estado de
energía libre mínima. Estos procesos ordenados garantizan la formación rápida de la estructura nativa y provocan la aparición de barreras
energéticas más grandes para los procesos que llevan a estructuras no
funcionales que para los que llevan a la estructura nativa (Schmid, 1992).
1.2 Desnaturalización
Se da el nombre de desnaturalizaci6n al proceso por el cual la proteína pierde sus estructuras cuaternaria, terciaria y secundaria y se
transforma en una distribución de conformaciones aleatorias de
aminoácidos. La secuencia primaria permanece inalterada. Sin embargo,
las propiedades físicas y químicas cambian y, en particular, se pierden las
propiedades enzimáticas. Una consecuencia de la desnaturalización es la
disminución en la solubilidad de la molécula, pues los grupos hidrofóbicos
ubicados en el interior quedan expuestos al solvente acuoso. La
disminución en la solubilidad puede facilitar la interacción entre grupos no
polares de diferentes cadenas conformadas
al
azar, lo que puede provocar,El proceso de desnaturalización de una proteína puede ser
desencadenado por un cambio en las condiciones del entorno de la
molécula, como un aumento en la temperatura, un aumento o disminución del valor del pH o la presencia de algún agente desnaturalizante (como urea o clorhidrato de guanidina) (Privalov, 1979; Timasheff, 1993). Se tiene
evidencia de que el grado de desdoblamiento que puede alcanzar una proteína es independiente, desde un punto de vista termodinámico, del
método de desnaturalización (Privalov, 1979). Una proteína en condiciones
de disolución extremas no necesariamente estará completamente desplegada, es decir, desprovista de cualquier estructura residual ni será capaz de comportarse como una cadena aleatoria. Tampoco se tendrán
necesariamente formas similares al final de procesos de desnaturalización
diferentes, pues la naturaleza heteroghnea de las secuencias de
polipéptidos hará que, bajo condiciones de desnaturalización semejantes,
los segmentos se comporten como si estuvieran en solventes distintos. Sin embargo, no es sencillo decidir cuán cerca de la cadena aleatoria se halla la conformación desplegada obtenida. En consecuencia,
es
necesariosuponer, al menos inicialmente, que el proceso de desnaturalización
idealizado no es sino la transición
a
un estado desplegado con estructurapróxima
a
la de la cadena aleatoria (Privalov, 1979).Los estudios sobre el desplegamiento de las proteínas pueden permitir
obtener información acerca de los mecanismos de plegamiento. En general,
bajo condiciones de plegamiento, las proteínas desnaturalizadas llegan
rápidamente al replegamiento completo pasando por una serie de equilibrios
entre conformaciones intermedias (Creighton, 1988). La velocidad de
reacción de desnaturalización cambia uniformemente con el cambio de las
condiciones ambientales; por el contrario, como la naturaleza del estado
desplegado difiere de acuerdo con las condiciones de desnaturalización
no uniforme. En consecuencia, es más sencillo analizar la cinética del
desplegamiento (Fersht et al., 1990).
Se ha comprobado que, para proteínas pequeñas de un solo dominio y
con masas moleculares inferiores a 35,000 Da, la desnaturalización es un
proceso completamente reversible, de modo que, en principio, es posible
aplicar la termodinámica de
los
sistemas en equilibrio al estudio de ladesnaturalización a través de la determinación de
los
cambios de entalpía,energía libre, entropía y capacidad calorífica (Labhardt, 1982; Sturtevant,
1987). Con este enfoque del problema se ha encontrado que la
desnaturalización de las proteínas es un proceso altamente cooperativo,
que involucro a toda la macromolécula y que está acompañado por un
enorme incremento en la entalpía y en la entropía (Privalov, 1979; Ptitsyn,
1994). La estabilidad de la proteína se define como
el
trabajo requeridopara
el
rompimiento cooperativo de toda la estructura de la molécula. Para una proteína en solución, este trabajo corresponde a la diferencia deenergía libre de Gibbs entre los estados nativo y desnaturalizado (Privalov,
1979).
Para proteínas mayores o de varios dominios, con una masa molecular
superior
a
los
35,000 Da,el
proceso de desnaturalizaciónes
más complejo,ya que puede haber especies intermediarias estables (Hernández-Arana y
Soriano-Garcia, 1988). Hay proteínas multidominio cuyo desplegamiento es
reversible (Dill, 1990). Sin embargo, el desplegamiento suele ser
irreversible, es decir, la molécula no recupera totalmente su estructura ni
sus propiedades nativas al volver a someterla a las condiciones iniciales: un
ejemplo de este comportamiento se encuentra en la termolisina (Sánchez-
Ruiz et ai, 1988). Se ha sugerido que la desnaturalización irreversible
puede ajustarse a dos modelos de primer orden distintos: el de dos estados
1.3 Dicroísmo circular (DC)
El dicroísmo circular (DC), es un método espectroscópico que mide la
actividad óptica de moléculas asimétricas en solución (las proteínas son
estructuras asimétricas). Las proteínas en DC presentan señales en dos regiones espectrales: el UV-lejano (170-250 nm) donde podemos encontrar
predominantemente la contribución de los enlaces peptídicos; y el UV
cercano (250-300 nm) donde se muestran bandas ocasionadas
principalmente por la presencia de aminoácidos aromáticos.
En esta técnica, es generalmente aceptado que los espectros de DC de una proteína son un reflejo directo de su estructura secundaria
(Hennessey and Johnson 1981), por lo que se utiliza ampliamente para
monitorear la transición cinética desde un estado nativo a un estado
desnaturalizado, ya que las diferencias estructurales en DC entre ambos
estados son muy notables (Creighton 1989).
Esta técnica se utilizó en un trabajo de Solis-Mendiola y
colaboradores [Q], en donde encontraron evidencias experimentales
suficientes, que determinaron que la desnaturalización térmica de la
quimopapaína implica un proceso irreversible y que además sigue un
mecanismo de dos estados N -z D. Este paso se lleva a cabo bajo una cinética de primer orden, donde la rapidez de la reacción -en este caso la
desnaturalización de la proteína- es directamente proporcional a una sola
1.4 Fluorescencia
Es conveniente probar otra técnica alternativa, aparte de dicroísmo circular que nos permita verificar el mecanismo de desnaturalización térmica
de la quimopapaína.
La fluorescencia es una técnica muy sensible a los cambios que
suceden
en
la conformación estructural de las proteínas, teniendo comoventaja que permite trabajar a concentraciones de muestra muy bajas. De
esta técnica, se obtiene información acerca de parámetros termodinámicos y
cinéticos relacionados con el proceso de plegamiento y desplegamiento de las proteínas y está directamente relacionada con la población de
macroestados en una transición de desplegamiento (Eftink 1994)
Las señales de fluorescencia de las proteínas están determinadas principalmente por la presencia de los aminoácidos aromáticos, como son:
el triptófano, la tirosina y la fenilalanina, por lo que las transiciones de
desplegamiento/plegamiento que son inducidas por agentes químicos, por temperatura y pH, se ven reflejadas en el tipo de señales que dichos
aminoácidos muestran. Por otro lado, es importante tener muy en cuenta
que la señal de fluorescencia es dominada predominantemente por la
presencia del triptófano (Creighton 1989).
Las propiedades de fluorescencia de los residuos de triptófano son
particularmente sensibles a las perturbaciones estructurales de las
proteínas, lo que además resulta ventajoso, ya que a menudo hay pocos residuos de este aminoácido en las proteínas.
Dependiendo de la longitud de onda de la excitación, podemos
evaluar solo a uno de los tres aminoácidos, por ejemplo para observar solo
fluorescencia del triptófano, la excitación debe ser a 295 nm (Creighton
1.5 Estabilidad de proteínas y ia radiación UV.
La exposición de las proteínas a la radiación ionizante y no ionizante
(como la radiación
UV)
causa efectos directos en la inactivación de lasproteínas (Methods in Molecular Biology 1995). De acuerdo con varios
trabajos experimentales, se observó que el triptófano tiene un importante
papel en la inactivación de las enzimas por radiación UV, lo que ha llevado
a generalizar que
los
aminoácidos aromáticos son sensibles a la fotólisis enla mayoría de las proteínas (Sellers,
R.
y Giron 1973). La fotosensibilidaddel triptófano, es determinada más por la naturaleza del aminoácido
adyacente, por su entorno local, así como si esta expuesto o no a un medio acuoso.
Sellers y colaboradores (Sellers,
R.
y Giron 1973), mencionan que a 295 nm aproximadamente el 97 % de la luz absorbida por la P-tripsina (la proteínacon la cual ellos trabajaron), realmente es absorbida por
los
residuos detriptófano; y que generalmente hay una pérdida de la actividad así como un
aumento en la fotodegradación de
los
triptófanos presentes en la enzima.Se ve entonces que una buena parte de
los
cambios observados enabsorción y en el espectro de fluorescencia de emisión deben ser atribuidos
a la fotólisis del triptófano, es decir, a la degradación del mismo y/o a la
formación de fotoproductos del triptófano (Pigault 1984), como la N- forinilquinurenina (NFK), el principal fotoproducto en disoluciones acuosas y
en condiciones aeróbicas (Tassin y Birkman 1980). En condiciones de
anaerobiosis el grado de aparición de fotoproductos disminuye.
En revisiones posteriores, se ha comprobado que el indol presente
en la estructura del triptófano, es un fluoróforo cuya fluorescencia, es
altamente sensible al ambiente, haciendo de esto una excelente elección
para reportar los cambios conformacionales en proteínas e interacciones
Por otro lado, Creed [15] reporta que en presencia de oxígeno, la
irradiación del dipeptido Gly-Trp y Trp-Gly produce N-tormilquinurenina y
quinurenina, mientras que con Trp-Gly y con el triptófano en N-terminal hay
mas resistencia a la oxidación.
De la observación de la secuencia de aminoácidos, podemos ver que hay 4
triptófanos (W). Sus respectivos aminoácidos adyacentes se encuentran
ubicados de la siguiente manera (figura 1.4):
.aminoácidos posición
DWR 6-8
CWA 25-27
GWE 66-68
SWG 180-182
NWG 184-186
Fig. 1.4 Posición de los triptófanos en la secuencia de
aminoácidos
De acuerdo con el trabajo de Tassin y Borkman [131, para una glicina
adyacente antes de un triptófano, no se observará fluorescencia
significativa debido a la degradación del triptófano. Resulto, distinto en el
modelo Trp-Gly, en donde se observó, un incremento en la intensidad de
la fluorescencia después de una irradiación, concretamente se manifiesta
una banda para a aproximadamente 440 nm cuando había una excitación
a 360 nm, lo cual además se debe a la aparición de fotoproductos como
1.6 Quimopapaína
La familia de enzimas denominadas proteasas sulfhidrílicas vegetales
(PSV) tiene una actividad proteolítica que depende del grupo tiol de un
residuo de cisteína de su secuencia de aminoácidos. La familia de las
proteasas sulfhidrílicas también incluye enzimas análogas que se
encuentran en animales (las catepsínas) y algunos microorganismos
(Kamphuis et ab, 1985a; Moore, 1969). Las PSV pueden obtenerse del
látex, endocarpio y zumo, o bien del tallo, las hojas o las raíces de una gran variedad de plantas. Por ejemplo, en el endocarpio de la piña (Ananas
comosus) y del kiwi (Actinidia chinenis) se encuentran la bromelaína
(Takahashi et ab, 1973) y la actinidina (Carne y Moore, 1978),
respectivamente, y de las hojas de la cebada (Hordeum vulgare) se puede
obtener la aleuraína (Holwerda y Rogers, 1992).
La quimopapaína, la papaína y la caricaína se extraen del látex de
papaya (Carica apava) (Baker y Drenth, 1987). Si bien se obtienen de una
misma fuente vegetal, son tipos particulares de proteínas que pueden ser
identificados tanto por su secuencia de aminoácidos como por sus
propiedades fisicoquímicas. Tienen una elevada homología en sus
secuencias de aminobidos; al comparar pares de proteínas se encuentra
que el porcentaje de residuos idénticos va del 40 al 90 % (Padilla-Zúñiga, 1993).
La quimopapaína es una de las PSV más ampliamente estudiadas. Su
secuencia de aminoácidos ha sido completamente determinada (Watson et
ai., 1990); se sabe que consta de 218 residuos, con una masa molecular de 24,000 Da (Solís-Mendiola et ai., 1989) y un punto isoeléctrico entre 10.1 y
10.6 (Brocklehurst et ab, 1981). La proteína consta de 68 grupos
1 10 20 30
Y P Q S I D W R A K G A V T P V K N Q G A C G S C W A F S T I
A T V
40 50 60
E G I N K I V T G N L L E L S E Q E L V D C D K H S Y G C K G
G Y Q T 7 0 80 90
1
O0
T S L Q Y V A N N G V H T S K V Y P Y Q A K Q Y K C R A T D K
P G P
K V K I T G Y K R V P S N C E T S F L G A L A N Q P L S V L V E
A G
G K P F Q L Y K S G V F D G P C G T K L D H A V T A V G Y G T
S170 180 190
200
D G K N Y I I I K N S W G P N W G E K G Y M R L K R Q S G N S
Q G T
C G V Y K S S Y Y P F K G F A
110 120 130
140 150 160
210 218
FIGURA 1 . 1 Secuencia de aminoácidos de la quimopapaína (Watson et
al.,
Tipo de residuo
Tyr: N-ter (+)
Ala: C-ter
Arg (+)
LYS
(+I
His (+)ASP
(-1
GIU (-)
CYS (-1
TYr
(4
Número de residuos
1 1 5 21 3 7 7 8 75 PK,
9.1 1
2.34 12.48 10.79 6.00 3.65 4.25 8.35 10.13
TABLA
1.
1 Residuos protonables de
la
quimopapaína. Se indica el valor
de pKa para residuos aislados en agua.
También se tiene información acerca de la actividad catalítica de la
quimopapaína (Buttle y Barret, 1984),
su
topografía (Watsonet
al., 1990; Dubois eta/., 1988; Carey et a/., 1983), reactividad (Enzyme Handbook, 1991), contenido de
estructura secundaria (Solís-Mendiola et ai., 1992) y estabilidad termodinámica
(Solís-Mendiola et
ai,
1993).Se ha determinado por difracción de rayos X la estructura tridimensional de tres
PSV: papaína (Kamphuis
et
aL, 1985b), actinidina (Baker, 1980) y caricaína (Pickersgill et a/., 1991). De la estructura de estas tres proteínas es posible deduciralgunas características estructurales comunes, probablemente comparadas por
otras PSV, entre ellas la quimopapaína. Se sabe que constan de dos dominios estructurales de aproximadamente 100 residuos cada uno, es decir, unos 200 por
cadena polipeptídica. El sitio catalítico, que se localiza en la hendidura
interdominio, está formado por tres aminoácidos: cisteína del primer dominio, e
interacciona fuertemente con el imidazol de la histidina, que a su vez se relaciona
con el carbonilo de la cadena lateral de la asparagina (Padilla-Zúñiga, 1993).
1.7 Objetivos
Un objetivo de este trabajo consiste en determinar, por medio de estudios de cinética de desnaturalización de la quimopapaína a diversas temperaturas, los
valores de la entropía y la entalpía de activación por la técnica de fluorescencia,
observar su posible variación con los valores obtenidos por dicroísmo circular.
de la fotooxidación en la desnaturalización térmica de la quimopapaína.
2.
MÉTODOS
EXPERIMENTALES
2.1 Materiales
Se utilizó quimopapaína parcialmente purificada (Sigma Chemical Co., St. Louis,
Missouri, U.S.A.). Se aisló la forma molecular más abundante (SolísMendiola, 1994)
por cromatografía líquida de intercambio catiónico de alta resolución, de acuerdo con
la metodología reportada por diversos autores (Solís-Mendiola et al., 1992). Con el
fin de impedir la autólisis durante los experimentos de desdoblamiento,
el
sitio activofue inhibido irreversiblemente con iodoacetamida (Solfs-Mendiola et a/., 1989).
Todos los demás reactivos fueron de grado analítico. La concentración de la proteína
fue determinada por medio del coeficiente de absorción de luz ultravioleta a 280 nm
(o coeficiente de extinción, A'%lcm,-=l 8.3) (Robinson, 1975; Polgár, 198 I).
2.2
Dicroismo circular
Se utilizó un espectropolarímetro JASCO J-500A con celdas de 1 mm y de 1 cm de trayectoria óptica, con chaqueta de circulación de agua conectada a un baño
térmico HAAKE NK-22. La temperatura se registró directamente en la celda óptica
con un termómetro digital COLE-PARMER 8402-20.
Se efectuaron estudios de cinética a diversas temperaturas y a un pH de 2.2. Se colocaron en la celda óptica 2 ml de un regulador de glicina 0.05 M con un valor de pH de 2.2. Se repitió el procedimiento mencionado anteriormente, inyectando 0.2 ml de una solución concentrada de quimopapaína (entre 1.35 y 1.60 mgimi) en el
mismo regulador y se observó la variación de la señal de dicroísmo circular a 220 nm.
2.3 Emisión de fluorescencia
Las determinaciones cinéticac de fluorescencia fueron hechas en un
concentración espectrofotométricamente. Antes de trabajar se puso en una celda de
trayectoria óptica de un 1 cm, 2000 pl de la misma disolución reguladora con la
finalidad de que se alcanzara el equilibrio térmico a la temperatura elegida, para
luego añadir 200 pI de una disolución de quimopapaína nativa con una concentración de 303 @mi y así obtener una Concentración final en la celda de 16 @mi.
La temperatura fue controlada con un baño de agua Fisher Scientific modelo
90, desde donde circulaba el agua por un sistema de enchaquetado hacia la celda,
obteniéndose una desviación de las lecturas de la temperatura de f
0.65°C. Se
observó en esta técnica, un tiempo muerto de mezclado de 25 segundos, teniendo
una anchura de banda de 16 nm en la excitación y de 4 nm para la emisión. Las
cinéticas de la desnaturalización térmica de la proteína, se realizaron a una longitud
de onda de emisión de 340 nm y 280 nm para la excitación (la
.
,
h
de la proteínanativa es de 340 nm). En todos los casos se obtuvieron curvas de intensidad relativa
contra tiempo. Después de que las cinéticas concluyeron se obtuvo el espectro de la
muestra desnaturalizada para luego ser enfriada hasta temperatura ambiente y sacar
-
3. RESULTADOS Y CONCLUSIONES
3.1 Consiantes de velocidad
Las curvas de las cinéticas observadas por monitoreo de la elipticidad
a
220mn, siguieron un patrón monoexponencial para todas las temperaturas
experimentales, lo cual confirma que por este método, se detecta un modelo de
desdoblamiento de
dos
estados:N
+
O,
el cual fue analizado con la ecuación (3.1.9)para calcular mediante una propiedad física que varia con la temperatura a cierto tiempo, las constantes de velocidad.
la quimopapaína en una fase a diferentes temperaturas, monitoreadas por DC:
A la longitud de onda de 280 nm, las constantes de velocidad son
esencialmente idénticas, estos resultados sugieren que no hay intermediarios presentes en la desnaturalización térmica seguida por DC, además el valor de estas constantes determinadas experimentalmente, representan una medida de un
desplegamiento global (Solís-Mendiola, et. a/. 1998).
DATOS DE QUIMOPAPAINA A pH 2.2 GLlClNA 0.05 M
Tabla
2
Valores de las constantes de velocidad para el desplegamiento de quimopapaína por monitoreo de la elipticidad a 220 nm.
Valores de las constantes de velocidad para el desplegamiento de quimopapaína monitoreado por fluorescencia
Usando el valor de las constantes de velocidad calculadas
a
partir de losestudios d dicroísmo circular, hacemos ajustes lineales utilizando la ecuación de
Eyring (para conocer el AH de activación a partir de la pendiente y
AS
de activación mediante la ordenada al origen. La ecuación de Arrhenius para calcular la energía de activación (Solís-Mendiola,et
ai 1998). Resultando de esto excelentes ajustes para todas las temperaturas. Los ajustes y las gráficas fueron hechas con elprograma Origin 4. 1 (Datos tesis).
Sin embargo, en las cinéticas del desplegamiento para la quimopapaína
monitoreadas por fluorescencia (Fig. 3.1 y 3.2), estas siguen un patrón biexponencial
(Fig. 3.3), del cual se deduce claramente que hay un mecanismo de tres estados:
Se obtienen dos constantes de velocidad, las cuales fueron calculadas de las
curvas de fluorescencia y utilizando la ecuación (3.2.33), dichos resultados se
IOOOOO
80000
6oooO
40000-
20000
Quirmpnpaino pH 7.0 O 0375 m@hi
I
-1
~~
-
-
-
-
-
-
I I I I
300
320
340
360 380Longitud de Onda (nm)
400
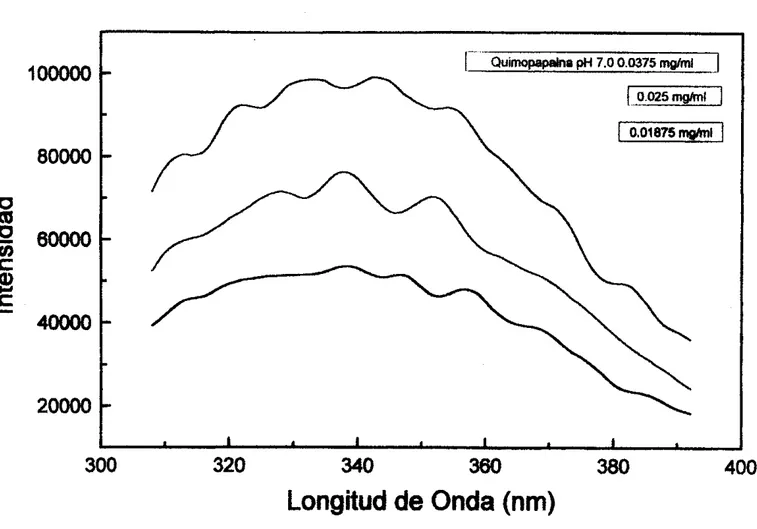
Fig. 3.1 Gráfica con las curvas de los datos obtenidos variando la concentración de la quimopapaína, a pH 7.0. Donde se eligió la concentración final a la cual se trabajó, siendo esta de 0.0375 mg/mi.
300 320 340
360
380
400Longitud de Onda
(nm)
40000
30000
U
m
W
UJ
c
20000
Ql
c
.-
CI
-
lo000
\
300
320
340
360
380400
Longitud de onda (nm)
40000
30000
W R1
T
u>
c
20000
a,
c
w-
10000
300
320
340
360380
400
Longitud de Onda (nm)
35000
3oooo
U
v)
C
Q,
25Ooo
3
20000
-
15000
10000
'
1 I 1 1 1 I I II
O
2000
4000 6OOO 8000 loo00 IZO0014OOO
16000
Tiempo
(seg)
f
\
35000
30000
25000
20000
15000
~
i Ciiieiica
Quirnopapaina
-
.
-
-
.
-
-
.
TEMPERAiüRA 58'C
1 m o
'
1 I I I I I I IO
2000
4000 6OOO8OOO
loo00
12000 14000 16000
35000
[
ClNETlCAQUlMOPAPAINA1
30000
35000
[
ClNETlCAQUlMOPAPAINA1
30000
-
n
üj
8 2 5 0 0 0 -
5 2 o o o o -
z
u
O 2000 4000 8ooo 8Ooo
12000
Tiempo
(seg)
3.2 Fotooxldación
Espectros
de
FluorescenciaLos espectros de emisión de fluorescencia de quimopapaina nativa y de quimopapaína irradiada desnaturalizada, se efectuaron a una longitud de onda de
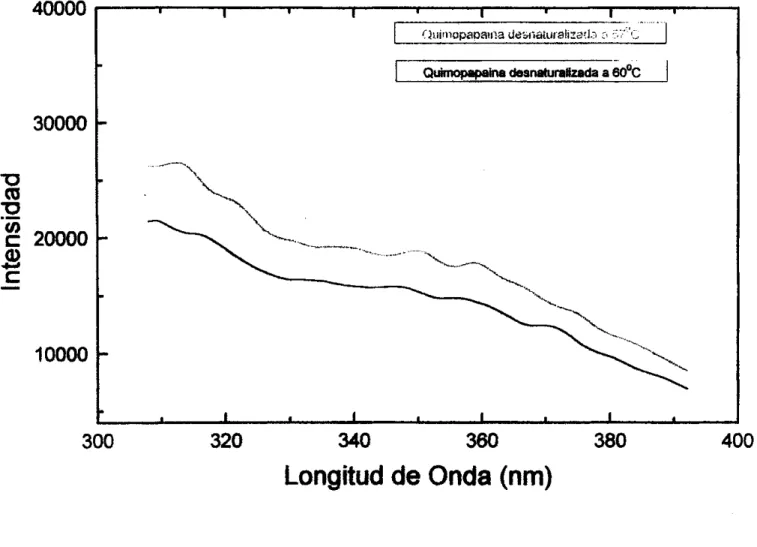
excitación de 325 nm. Tal como se ilustra en la figura 3-6,
los
espectros dequimopapaina desnaturalizada (a 57, 58, 60 y 62
O C )
revelan la producción de nuevos productos fluorescentes, ya que la quimopapaína nativa no emite en elintervalo de 360-500 nm. Espectros similares son descritos por Pigault y Gerard [l
21.
La N-formilquinurenina (NFK) y la quinurenina (Kyn) son fotoproductos que pueden aparecer (entre otros) como consecuencia de la fotólisis de los triptófanos
3.3 Conclusiones
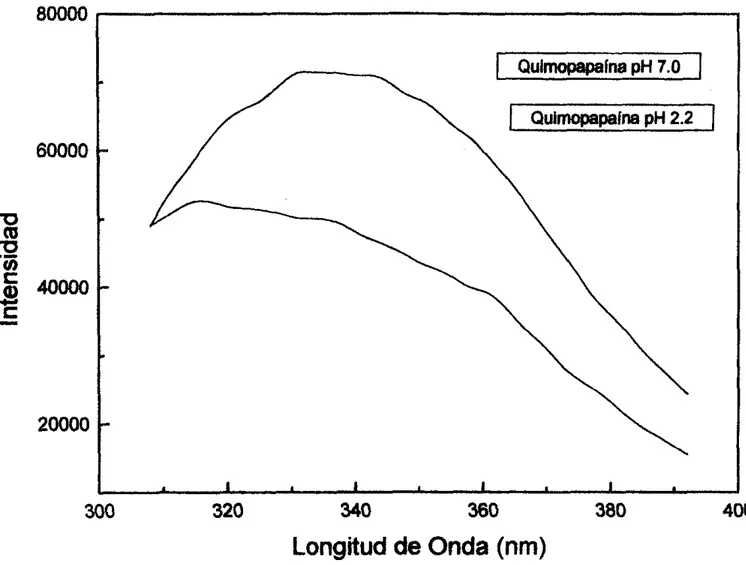
A partir de los resultados obtenidos en DC, se puede confirmar que el modelo de desplegamiento para la quimopapaína a pH 2.2, sigue un mecanismo cinético de
dos estados, y que este es irreversible.
AI analizar los resultados de emisión de fluorescencia se observó un mecanismo cinético de desplegamiento de tres estados, con una constante de
velocidad para cada transición. Si observamos
los
parámetros de activación, se notaclaramente que
los
valores de la segunda fase cinética no corresponden a unatransición de desplegamiento. Además experimentalmente, el método ha hecho notar que hay una reacción de fotólisis donde aparecen fotoproductos (probablemente NFK
y otros productos de fotodegradación), que obviamente reflejan su existencia por
medio de absorción de luz a una longitud de onda donde las proteínas normalmente
ya no absorben y además a través de una transición, la cual seguramente
corresponde a la segunda fase cinética que se observa en la fluorescencia.
Se observó que conforme transcurría el tiempo, la especie nativa va
desapareciendo, para dar lugar a un estado desnaturalizado y que al mismo tiempo,
este va siendo fotooxidado. Es importante hacer notar que cuando D esta a un
tiempo máximo, la concentración de DI es pequeña,
lo
cual pudiera deberse a unaausencia de estructura secundaria y terciaria en D .
Queda bastante claro, que la única fase cinética detectada por DC,
corresponde a la primera fase observada por fluorescencia, es decir que D y DI,
-
dadas las concentraciones utilizadas (1 Sggími), son indistinguibles una de otra por
dicroísmo circular [27].
En investigaciones experimentales previas, donde se trabajó con el método de DC,
utilizando
a
la quimopapaína [26], se encontró que la cinética de desnaturalización seajustaba muy bien a una reacción irreversible de primer orden y de dos estados, así
Evidentemente la desnaturalización térmica de la quimopapaína se ve
afectada por la exposición de la misma a los rayos UV. Así entonces la contribución
de la fotooxidación de la proteína a la desnaturalización de la misma, es digna de
tomarse en cuenta, ya que como se ha visto dependiendo de la cantidad de los
triptófanos y de la posición de estos, será el grado de fotooxidación de la proteína,
lo
cual se verá reflejado en la posible aparición de fotoproductos. Estos a su vez
provocarán la aparición de bandas en los espectros de la proteína ya desnaturalizada
y enfriada a la temperatura ambiente.
AI revisar los parámetros de activación vemos que el efecto de la fotooxidación
se refleja directamente en la segunda fase cinética detectada en fluorescencia, cuyos
valores como ya se dijo antes no son propios de una transición efectiva de