CARACTERIZACIÓN MOLECULAR DE CEPAS NATIVAS COLOMBIANAS DE
Azotobacter spp. MEDIANTE EL ANALISIS DE RESTRICCION DEL DNA RIBOSOMAL 16S
DIEGO JAVIER JIMÉNEZ AVELLA
PONTIFICIA UNIVERSIDAD JAVERIANA
FACULTAD DE CIENCIAS BÁSICAS
MICROBIOLOGÍA INDUSTRIAL
Bogotá, D. C. Colombia
CARACTERIZACIÓN MOLECULAR DE CEPAS NATIVAS COLOMBIANAS DE
Azotobacter spp.MEDIANTE EL ANALISIS DE RESTRICCION DEL DNA RIBOSOMAL 16S
DIEGO JAVIER JIMÉNEZ AVELLA
TRABAJO DE GRADO
PRESENTADO COMO REQUISITO PARCIAL PARA OBTAR EL TITULO DE
MICROBIOLOGO INDUSTRIAL
DIRECTOR:
JOSE SALVADOR MONTAÑA
CO-DIRECTORA:
MARIA MERCEDES MARTINEZ
PONTIFICIA UNIVERSIDAD JAVERIANA
FACULTAD DE CIENCIAS BÁSICAS
MICROBIOLOGÍA INDUSTRIAL
Bogotá, D. C. Colombia
NOTA DE ADVERTENCIA
Articulo 23 de la resolución No 13 de julio de 1946
“La universidad no se hace responsable por los conceptos omitidos por sus alumnos
en sus trabajos de tesis. Solo se velara porque no se publique nada contrario al
dogma y a la moral católica y porque las tesis no contengan ataques personales
contra persona alguna, antes bien se vea en ellas el anhelo en buscar la verdad y la
justicia”
CARACTERIZACIÓN MOLECULAR DE CEPAS NATIVAS COLOMBIANAS DE
Azotobacter spp. MEDIANTE EL ANALISIS DE RESTRICCION DEL DNA RIBOSOMAL 16S
DIEGO JAVIER JIMÉNEZ AVELLA
APROBADO
José Salvador Montaña Msc. Director
María del Pilar Márquez Msc. Jurado 1
María Mercedes Martínez Msc. Codirectora
Andrea Juliana Mantilla Microbióloga Industrial
Jurado 2
Bogotá. D. C. Colombia
CARACTERIZACIÓN MOLECULAR DE CEPAS NATIVAS COLOMBIANAS DE
Azotobacter spp.MEDIANTE EL ANALISIS DE RESTRICCION DEL DNA RIBOSOMAL 16S
DIEGO JAVIER JIMÉNEZ AVELLA
APROBADO
Ángela Umaña MPhil. Decana Académico
David Gómez Msc. Director de Carrera
Bogotá. D. C. Colombia
DEDICATORIA
AGRADECIMIENTOS
A mis directores José Salvador Montaña y María Mercedes Martínez, por su apoyo incondicional, por compartir sus invaluables enseñanzas y conocimientos conmigo y por acompañarme en este valioso paso en mi vida.
A todas las personas del Laboratorio de Microbiología Ambiental y Suelos de la Pontificia Universidad Javeriana, por su colaboración y por haber permitido el desarrollo de esta investigación.
A los investigadores de las Unidades de Saneamiento y Biotecnología Ambiental (USBA) y Biotecnología Vegetal (UBV) por su colaboración y asesoría.
Al Ingeniero agrónomo Andrés Siabato por su colaboración y asesoría en el muestreo.
A mis hermanos Julis, Caro y Chepe, por su apoyo y cariño en toda mi carrera.
A mi linis por amarme tanto, por su compañía, por su comprensión y por ser un apoyo incondicional.
A todas las personas de mi familia que me apoyaron y creyeron en mí día a día.
TABLA DE CONTENIDO
1. Introducción... 1
2. Marco teórico... 3
2.1. Importancia y problemática del nitrógeno en la agricultura... 3
2.2. La rizosfera... 3
2.3. Microorganismos fijadores de nitrógeno atmosférico (diazótrofos)... 5
2.3.1. Fijación biológica de nitrógeno (FBN)... 5
2.3.2. Microorganismos diazotróficos simbióticos... 7
2.3.3. Microorganismos diazotróficos asimbióticos... 9
2.5. Familia Azotobacteraceae... 11
2.6. Generalidades del género Azotobacter spp... 12
2.6.1. Azotobacter chroococcum... 16
2.6.2. Azotobacter vinelandii... 17
2.7. Técnicas moleculares para la caracterización e identificación de bacterias diazotrófas... 18
2.7.1. Aplicaciones del DNA ribosomal 16S y su utilización en filogenia y taxonomía... 19
2.7.2. Análisis de restricción del DNA ribosomal 16S amplificado (ARDRA)... 21
2.7.3. Caracterización, identificación y filogenia del género Azotobacter y otras bacterias diazótrofas mediante el análisis de restricción del RNA ribosomal 16S (ARDRA)... 21
3. Formulación del problema y justificación... 25
4. Objetivos... 27
4.1. Objetivo general... 27
4.2. Objetivos específicos... 27
5. Metodología... 28
5.1. Lugar de muestreo... 28
5.2. Muestreo del suelo... 28
5.3. Procesamiento de muestras... 29
5.3.1. Medición de pH de las muestras de suelo... 29
5.3.2. Aislamiento primario... 29
glicerol al 50% (v/v)...
5.4.2. Pigmentación de los aislamientos... 30
5.4.3. Identificación Bioquímica…... 31
5.4.4. Cepa control... 31
5.5. Extracción de DNA... 32
5.6. Amplificación de DNA ribosomal 16S con iniciadores Y1 y Y3... 33
5.7. Análisis de restricción del DNA ribosomal 16S amplificado (ARDRA)... 34
5.7.1. Perfil electroforético para el ARDRA... 34
5.7.2. Análisis filogenético... 35
5.8. Restricción virtual... 35
6. Resultados... 36
6.1. Medición de pH de los cultivos muestreados... 36
6.2. Porcentaje de recuperación de bacterias diazótrofas aerobias asimbióticas.. 36
6.3. Identificación morfológica de bacterias diazótrofas aerobias asimbióticas... 37
6.3.1. Morfología de las colonias... 37
6.3.2. Morfología de las células... 37
6.3.3. Pigmentación... 38
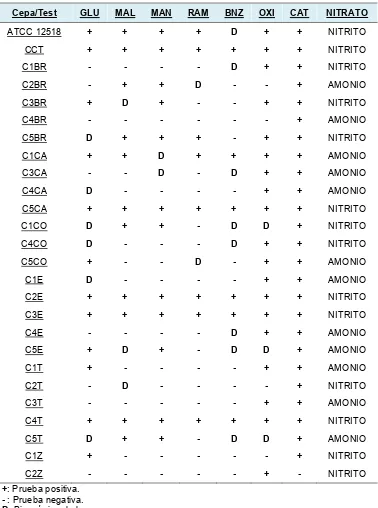
6.4. Identificación bioquímica... 39
6.5. Extracción de DNA... 41
6.6. Amplificación del DNA ribosomal 16S con los primers Y1 y Y3... 42
6.7. Análisis de restricción del DNA ribosomal 16S (ARDRA)... 43
6.7.1. Análisis con la enzima AluI... 43
6.7.2. Análisis con la enzima HpaII... 44
6.7.3. Análisis con la enzima RsaI... 46
6.8. Cladograma generado del análisis con las tres enzimas... 48
6.9. Restricción virtual... 48
6.9.1. Análisis virtual con la enzima AluI... 50
6.9.2. Análisis virtual con la enzima HpaII... 52
6.8.3. Análisis virtual con la enzima RsaI... 53
7. Discusión de resultados... 56
8. Conclusiones... 67
9. Recomendaciones... 69
10. Referencias... 70
INDICE DE FIGURAS
Figura. 1. Genes implicados en el flujo de electrones hacia el sitio activo de la
nitrogenasa para la reducción del N2... 6
Figura. 2. Quistes de Azotobacter spp... 12
Figura. 3. Pigmentación de cepas de Azotobacter... 13
Figura. 4. Colonias de Azotobacter spp. en medio libre de nitrógeno y morfología celular de Azotobacter spp... 14
Figura. 5. Esquema de la organización de los genes involucrados en la síntesis de las tres nitrogenasas utilizadas por A. vinelandii en la fijación de nitrógeno... 18
Figura. 6. Lugar de muestreo en el departamento de Boyacá... 28
Figura. 7. Recolección de las muestras de suelo... 29
Figura 8. Aislamiento primario... 30
Figura. 9. Siembra en estria en agar Ashby con benzoato... 31
Figura. 10. Baterías bioquímicas de azucares... 31
Figura. 11. Morfología de colonia encontrada en los aislamientos de bacterias fijadoras de nitrógeno aisladas de cultivos de hortalizas... 37
Figura. 12. Morfología celular de los aislamientos... 38
Figura. 13. Pigmentación de los aislamientos en agar Ashby-Benzoato... 38
Figura. 14. Caracterización bioquímica de los aislamientos... 41
Figura. 15. Amplificación del DNAr 16S... 42
Figura. 16. Electroforesis en gel de agarosa al 2.0%. Digestión con la enzima AluI... 43
Figura. 17. UPGMA con la enzima AluI para los aislamientos... 44
Figura. 18. Electroforesis en gel de agarosa al 2.0%. Digestión con la enzima HpaII... 45
Figura. 19. UPGMA con la enzima HpaII para los aislamientos... 46
Figura. 20. Electroforesis en gel de agarosa al 2.0%. Digestión con la enzima RsaI... 46
Figura. 21. UPGMA con la enzima RsaI para los aislamientos... 47
Figura. 22. Análisis de agrupamiento UPGMA obtenido con las enzimas AluI, HpaII y RsaI para los aislamientos... 48
Figura. 24. Restricción virtual con la enzima AluI... 51
Figura. 25. UPGMA del análisis de restricción virtual con la enzima AluI para las secuencias obtenidas del Genbank... 51
Figura. 26. Restricción virtual con la enzima HpaII... 52
Figura. 27. UPGMA del análisis de restricción virtual con la enzima HpaII para las secuencias obtenidas del Genbank... 53
Figura. 28. Restricción virtual con la enzima RsaI... 54
Figura. 29. UPGMA del análisis de restricción virtual con la enzima HpaII para las secuencias obtenidas del Genbank... 55
INDICE DE ECUACIONES
INDICE DE TABLAS
Tabla 1. Colores de pigmentos solubles en agua producidos por bacterias del
género Azotobacter... 13
[image:12.612.132.522.102.615.2]Tabla 2. Movilidad, formación de filamentos y pruebas bioquímicas utilizadas para la caracterización del género Azotobacter spp.... 15
Tabla 3. Secuencia y número de pares de bases de los iniciadores utilizados para la amplificación del DNAr 16S de bacterias diazótrofas... 33
Tabla 4. Enzimas de restricción utilizadas en los ensayos de ARDRA... 34
Tabla 5. Valores de pH de suelo de cada cultivo... 36
Tabla 6. Recuento de gránulos con colonias mucilaginosas y porcentaje de recuperación por cultivo... 36
Tabla 7. Numero de cepas aisladas en agar Ashby- Sacarosa por cada cultivo... 37
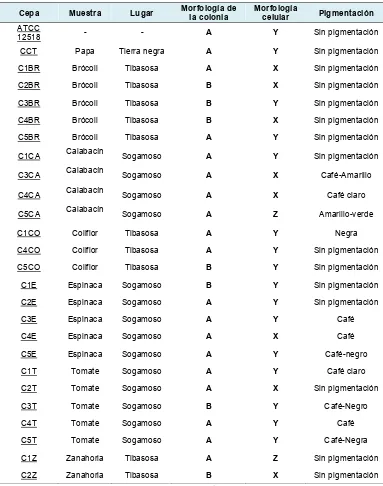
Tabla 8. Características fenotípicas de los aislamientos de cepas diazotrófas encontradas en cultivos de hortalizas en el departamento de Boyacá... 39
Tabla 9. Caracterización bioquímica de los aislamientos, el control positivo (ATCC 12518) y la cepa CCT... 40
Tabla 10. Identificación preliminar de los aislamientos... 41
Tabla 11. Morfotipos obtenidos con la enzima AluI... 43
Tabla 12. Morfotipos obtenidos con la enzima HpaII... 45
Tabla 13. Morfotipos obtenidos con la enzima RsaI... 47
Tabla 14. Secuencias obtenidas en el Genbank para el análisis de restricción virtual... 49
Tabla 15. Número de bandas y pesos moleculares mayores a 100pb generados con la enzima AluI en las secuencias obtenidas del Genbank... 50
Tabla 16. Número de bandas y pesos moleculares mayores a 100pb generados con la enzima HpaII en las secuencias obtenidas del Genbank... 52
RESUMEN
Con el fin de aislar y caracterizar bacterias diazotrófas del genero Azotobacter se realizó un muestreo de suelo en cultivos de hortalizas ubicados en el departamento de Boyacá, el aislamiento se realizó en medio Ashby-Sacarosa libre de nitrógeno. El pH de los suelos se encontró cercano a la neutralidad y el porcentaje de recuperación obtenido fue alrededor de 40%, los aislamientos fueron clasificados e identificados fenotípica y bioquímicamente, encontrando bacterias de las especies A. vinelandii, A. chroococcum, A. negricans y A. paspali. La caracterización molecular se realizó utilizando un análisis de restricción del DNAr 16s amplificado (ARDRA), para esto se realizó una extracción de DNA por medio de la utilización de solventes orgánicos (Fenol), el gen fue amplificado con iniciadores universales utilizados para bacterias diazótrofas (Y1-Y3) que amplifican 1487pb del gen. Las enzimas utilizadas en el ARDRA fueron AluI, HpaII y RsaI las cuales presentaron porcentajes de polimorfismo alrededor de 16%. Los aislamientos se distribuyeron en tres grupos que presentaron morfotipos similares para las tres enzimas.
ABSTRACT
With the aim to isolate and characterized nitrogen fixing bacteria from
Azotobacter genera, a soil sample was made in vegetable cultures located at the department of Boyacá, the isolation was made in the Ashby-Sucrose medium free of nitrogen. The PH of soils was found near neutrality, and the porcentage of recovery obtained was around the 40%, the isolations were classified and identified phenotypical and biochemiclally, finding bacterias of the species A. vinelandii, A. chroococcum, A. negricans y A. paspali. The molecular characterization was made using an amplified ribosomal DNA restriction analysis (ARDRA), for this an extraction of the DNA was made by using organic solvents (phenol), the gen was amplified with universal primers used for nitrogen fixing bacterias (Y1-Y3) that amplifies 1487pb of the gen. The enzymes used in the ARDRA were AluI, HpaII y RsaI which presented polymorphism percentages around 16%. The isolations were distributed in three groups that presented similar morphotypes for the three enzymes.
1. INTRODUCCIÓN.
Actualmente la gran mayoría de cultivos agrícolas Colombianos dependen de los fertilizantes químicos sintetizados, los cuales proporcionan nutrientes asimilables por las plantas, buscando optimizar los procesos de producción. El uso indiscriminado de fertilizantes químicos, ocasiona contaminación de cuerpos de agua y suelos, sumado al incremento de los costos de producción y el impacto negativo en la salud animal y humana. Por esto, el uso de agricultura orgánica, tecnologías limpias y seguras, han hecho que los biofertilizantes se conviertan en una gran alternativa para disminuir estos inconvenientes. Los biofertilizantes son preparaciones de células vivas o latentes que cumplen funciones específicas en los cultivos, como, la fijación biológica de nitrógeno, la solubilización del fósforo, la producción de hormonas promotoras de crecimiento, la producción de antibióticos y la generación de agentes quelantes de hierro.
Las bacterias fijadoras de nitrógeno (diazótrofas) fueron las primeras en producirse comercialmente con fines de biofertilización pero posteriormente las bacterias diazótrofas asimbióticas cobraron importancia en la agricultura. De este grupo, las más utilizadas como biofertilizantes corresponden al género
Azotobacter, compuesto por 7 especies (Azotobacter chroococcum, A. vinelandii, A. beijerinckii, A. paspali, A. armeniacus, A. nigricans y A. salinestris), siendo A. chroococcum y A. vinelandii las que se encuentran en gran proporción en la rizosfera de suelos tropicales que se caracterizan por poseer altos contenidos de materia orgánica, fosfatos y valores de pH cercanos a la neutralidad. Las bacterias del género Azotobacter son fijadores de nitrógeno de vida libre, solubilizadores de fósforo, productores de sustancias promotoras del crecimiento vegetal (PGPR) y degradadoras de plaguicidas. Por estas razones se consideran indicadores de salud en cultivos agrícolas.
Los métodos de cultivo más utilizados para aislar bacterias del género
en diferentes concentraciones de NaCl y de oxigeno. La identificación también se realiza mediante observación de la morfología celular, de colonias y la formación de estructuras de resistencia. Sin embargo, estos métodos generan ambiguedad en el momento de diferenciar especies dentro del género
Azotobacter u otros géneros diazótrofos como Beijerinckia y Derxia. Por esta razón su utilización es insuficiente para la identificación y caracterización de bacterias diazotrófas, además de ser útiles solo para bacterias cultivables. Debido a esto, se han desarrollado técnicas de huella genética como DGGE, RFLP, RAPD, ARDRA y secuenciación que permiten una identificación y caracterización mas precisa.
El DNA ribosomal 16S es utilizado en estudios de filogenia y taxonomía ya que su estructura y función han permanecido constantes a través del tiempo de modo que las alteraciones en la secuencia reflejan cambios aleatorios, su transmisión es principalmente vertical ya que no está sujeto a transferencia génica horizontal entre microorganismos. La técnica molecular de amplificación del DNA ribosomal 16S es utilizada constantemente en microbiología, ya que por sus características precisas permite identificar y caracterizar bacterias no cultivables, bacterias cuyas características bioquímicas no se adaptan a las de ningún género o especie reconocido; bacterias con elevados requerimientos nutricionales; cuya caracterización fenotípica no es suficiente o aquellas con crecimiento muy lento.
En este estudio se pretende obtener un acercamiento a la identificación, caracterización molecular y filogenia de cepas nativas Colombianas del género
2. MARCO TEORICO
2.1 Importancia y problemática del nitrógeno en la Agricultura
Debido a que el nitrógeno es el elemento más limitante para el crecimiento de las plantas en suelos tropicales (Franco & Dobereiner, 1994), se ha incrementado el uso de productos de síntesis, con el ánimo de aumentar la producción agrícola. Existen reportes de 77 x 106 toneladas de nitrógeno aplicados mundialmente como fertilizante en diversos cultivos de gran importancia agronómica, como hortalizas, caña, sorgo, maíz, arroz, flores y ornamentales entre otros. Lo anterior ha provocado efectos negativos en los recursos naturales, tales como acumulación de nitratos en las aguas freáticas, toxicidad en las plantas por la presencia de altos niveles de NO2 en los suelos,
contribuyendo con la muerte de la biota del suelo, ocasionando desequilibrios en los procesos naturales biogeoquímicos que se traducen en un alto costo económico, social y ecológico. (Marín et al., 2003).
2.2 La rizósfera
El termino rizósfera se refiere a la zona del suelo influenciada por el desarrollo de raíces, en donde se activa la proliferación de microorganismos (Hiltner, 1904); este efecto rizosférico se debe al suministro de exudados radicales que contienen azúcares, aminoácidos, vitaminas y enzimas, además de señales que modulan la interacción microbio-planta (Kennedy & Smith, 1995; Bowen & Rovira, 1999). Las propiedades físicas, químicas y biológicas de la rizósfera son muy diferentes a las del suelo no rizosférico particularmente, la población microbiana desciende entre 10 a 100 veces al alejarse pocos milímetros de la superficie radical (Collados, 2006).
pero no cultivables (Barea et al., 2005), por lo que dichas técnicas permiten caracterizar los microorganismos y establecer sus relaciones filogenéticas.
Análisis pioneros de la complejidad microbiana en suelos, realizados por Torsvik
et al., (1990), utilizando técnicas de reasociación de DNA, pusieron en evidencia la gran diversidad genética bacteriana presente. Así el número de genomas bacterianos diferentes estimados en una muestra de suelo forestal fue de unos 4.000, en claro contraste con la diversidad genética de las bacterias aisladas en cultivo a partir de la misma muestra (200 veces menor). En otras muestras de suelo de la amazonia brasilera la diversidad genómica fue incluso superior (8.000-10.000 genomas diferentes) (Torsvik et al., 1998).
Los microorganismos en la rizosfera desempeñan funciones de gran importancia en relación con procesos de edafogénesis; ciclos biogeoquímicos de elementos como el carbono, el nitrógeno, oxígeno, el azufre, el fósforo, el hierro y otros metales; fertilidad de las plantas y protección frente a patógenos; degradación de compuestos xenobióticos y producción de fitohormonas (Nogales, 2005).
Dentro de la microflora de la rizosfera se encuentran especies pertenecientes a los géneros Agrobacterium, Alcaligenes, Arthrobacter, Enterobacter, Erwinia, Flavobacteria, Hafnia, Klebsiela, Serratia Xanthomonas, Azotobacter,
Azospirillum, Clostridium, Pseudomonas, Acetobacter, Burkholderia y Bacillus
(Kloepper et al., 1989; Bashan & Levanony, 1990; Tang, 1994; Barea et al., 2004). Otras bacterias importantes en la microflora del suelo y en la agricultura son las fosfato solubizadoras, como las aisladas por Useche et al., (2004), en un estudio de la diversidad en diferentes suelos de la Amazonía, Pseudomonas spp., P. cepacia, P. gladioli, Xanthomonas spp., X. maltophilia, Enterobacter agglomerans, Chromobacterium sp., X. maltophilia, y Chromobacterium sp.; constituyeron nuevos reportes de bacterias solubilizadoras de fósforo en Colombia.
importante para la funcionalidad y estructura del suelo. Las bacterias se organizan en microcolonias compuestas de pocas células que pueden pertenecer a diferentes morfotipos (Nunan et al., 2003).
2.3 Microorganismos fijadores de nitrógeno atmosférico (diazotrófos)
La fijación biológica de nitrógeno molecular la llevan a cabo diversos géneros de bacterias de vida libre, algunas de estas se encuentran en la rizósfera en vida libre, y otros géneros bacterianos forman asociaciones mutualistas con plantas (Saribay, 2003). Las bacterias fijadoras de nitrógeno presentan una amplia diversidad taxonómica, con diferentes estilos de vida y de asociación con los vegetales. Sin embargo, sólo una pequeña proporción de especies es capaz de hacerlo; 87 especies en dos géneros de arqueobacterias, 38 de bacterias, y 20 géneros de cianobacterias se han identificado como diazótrofas (Hussein, 1999).
En la rizósfera la fijación del nitrógeno se realiza aparentemente sólo por ciertos tipos de bacterias y por algunos miembros del taxón Archea; estos diazotrófos incluyen algunas especies de Bacillus spp., Clostridium spp. y Klebsiella spp., miembros de la familia Azotobacteraceae (A. vinelandii y A. chroococcum),
Rhizobiaceae y del orden Rhodospirillales (Singleton, 2004). Además de estos, se han descrito géneros en diferentes hábitats con la capacidad de fijar nitrógeno atmosférico, entre estos están: Beijerinckia, Chromatium, Rhodopseudomonas, Rhodospirillum, Rhodomicrobium, Chlorobium, Azospirillum, Desulfovibrio,
Desulfotomaculum y Pseudomonas (Atlas & Bartha 2002).
2.3.1 Fijación biológica de nitrógeno (FBN)
La fijación biológica de nitrógeno esta catalizada por el complejo enzimático
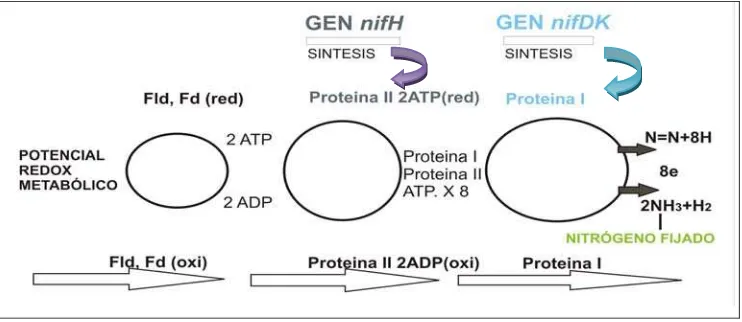
nitrogenasa. El complejo de la enzima tiene dos coproteínas, la proteína I contiene hierro y molibdeno y la proteína II contiene solamente hierro, aunque se han encontrado una enzima alterna de Azotobacter vinelandii que contiene vanadio (Drummond et al., 1995; Eady et al., 1988). La proteína I es un tetrámero de alrededor de 220.000 Da, formado por dos tipos de subunidades α2 β2, de masa molecular semejante, y producto de los genes nifDK (Telisa et al.,
N2, (Figura 1) (Baca et al., 2000). Se han descrito alrededor de 20 genes
[image:20.612.133.504.270.431.2]involucrados en la fijación biológica de nitrógeno, cuya secuencia y funciones han sido ya determinadas. Igualmente se conoce que los genes estructurales de la nitrogenasa están sumamente conservados, así como un cierto número de otros genes cuyos productos juegan un papel en la maduración de la enzima, la síntesis del cofactor, el transporte de molibdeno y la regulación (Elmerich, 1993; Lee et al., 2000; Desnoues et al., 2003). Las proteínas NifL y NifA son las encargadas de la regulación y el control de la expresión de los genes nif en la fijación biológica del nitrógeno, en Azotobacter vinelandii estas dos proteínas están codificadas por el operon nifLA (Martínez et al., 2005).
Figura 1. Genes implicados en el flujo de electrones hacia el sitio activo de la nitrogenasa para la reducción del N2. Fuente: Baca et al., (2000).
el proceso se realiza en estructuras de resistencia como los heterocistes. (Singleton, 2004).
Ecuación 1. Reacción en la fijación de nitrógeno. Fuente: Baca et al., (2000) N2 + 8H+ + 8e- + 16MgATP → 2NH3 + H2 + 16MgADP +16Pi
La nitrogenasa purificada es inactivada rápida e irreversiblemente por el O2, la
proteína (Fe) es mucho más sensible que la proteína (Mo-Fe) con una vida media en presencia de aire de 45 segundos y 10 minutos respectivamente (Robson & Postgate, 1980). El efecto de O2 sobre la nitrogenasa restringe la
fijación de N2 en la mayoría de las especies de eubacterias a condiciones
anaeróbicas o de microaerobiosis. Azotobacter y Beijerinckia pueden fijar nitrógeno a la presión normal de oxígeno, ya que protegen su nitrogenasa de la inactivación oxidativa por una combinación de compartimentación y de complejos mecanismos bioquímicos entre los que se encuentran, la protección conformacional o switching off (Moshiri et al., 1994); la expresión de la citocromo oxidasa cytbd; la autoprotección, la cual consiste en reducir por medio de la nitrogenasa reductasa el oxigeno a peróxido de hidrogeno y posiblemente a agua y por lo tanto, reduciría el oxigeno en la vecindad de la nitrogenasa
(Thorneley & Ashby, 1989). Además de esto, el uso de nitrogenasas alternativas y la producción de polímeros como alginato y los PHBs (Polihidroxibutiratos) son alternativas eficientes para proteger el complejo (Atlas & Bartha, 2002; Espin, 2002 <on line>).
2.3.2 Microorganismos diazotróficos simbióticos.
Las bacterias diazotróficas simbióticas son aquellas que son capaces de fijar nitrógeno atmosférico en simbiosis con las raíces de plantas leguminosas (guisantes, judías, tréboles, alfalfa, entre otras). Estas raíces poseen pequeños engrosamientos llamados nódulos que contienen bacteroides o rizobios, en esta alianza, la planta suministra nutrientes y protección, mientras que el rizobio proporciona a la planta nitrógeno fijado de la atmósfera (Espín, 2002 <online>). Se han descrito 40 especies en 9 géneros de bacterias que forman nódulos en leguminosas, sin embargo, existen bacterias simbióticas que pueden colonizar otras plantas, Por ejemplo, Rhizobium leguminosarum y cepas fotosintéticas de
Bradyrhizobium se han encontrado en raíces de arroz (Yanni et al., 2001) ;
caulinodans en las raíces de la oleaginosas (Brassica napus); los nódulos de las raíces de aliso (Alnus) contienen bacterias del género Frankia, mientras que el pequeño helecho flotante Azolla contiene Anabaena azollae dentro de cavidades especializadas. Así mismo, se han detectado cepas pertenecientes a la cianobacteria Nostoc en las raíces de corraloides de cícadas (Cycas, Encephalatos, Zamia) de un jardín botánico mediante métodos basados en PCR. Pisa, (2002), realizó una análisis filogenético amplificando el DNAr 16S con primers especificos para diazótrofas de cepas simbióticas capaces de nodular cultivos de frijol (Phaseolus vulgaris) en Brasil, encontrando un 42% de bacterias del genero Rhizobium, 17% de Ralstonia y 6% de Burkholderia.
Una de las simbiosis más efectivas es la que se establece entre Bradyrhizobium japonicum-Soya, donde un 70% de la fijación de nitrógeno por la bacteria es asimilada por la planta. En Brasil se ha empleado con éxito la fertilización biológica de soya con B.japonicum y 0% de aporte de nitrógeno como fertilizante químico, ello ha conducido a que este país sea el segundo productor de soya a nivel mundial (Baldani et al., 1997). En Colombia se han evaluado biofertilizantes a base de Rhizobium en cultivos de maní, frijol y soya con resultados positivos en la producción y en la disminución de costos (Torres, 2000 <on line>).
Asi mismo, la utilización de plantas fijadoras de nitrógeno como el trébol y la alfalfa, pueden incluirse dentro de los esquemas de rotación de las cosechas; plantas como la Azolla se utiliza como abono orgánico, una práctica común en el sudeste de Asia, mientras que en los arrozales la fertilidad puede incrementarse potenciando el crecimiento de cianobacterias autónomas fijadoras de nitrógeno (Singleton, 2004). Por otro lado, Santillana et al., (2005), comprobaron la capacidad de Rhizobium para promover el crecimiento de plantas de tomate (Lycopersicon esculentum Miller), evidenciando la producción de sustancias promotoras de crecimiento de tipo citoquininas, ácido indol acético y ácido giberélico.
sintetizadas por los genes de nodulación del microsimbionte (genes nod), en respuesta a la excreción por la planta de sustancias de tipo flavonoide (Baca et al., 2000). Se han reportado que existen alrededor de 26 genes nod expresados en el proceso de nodulación y 26 genes nif expresados en la fijación de nitrógeno, los cuales codifican para 416 proteínas involucradas en estos dos sistemas (Freiberg et al., 1997). Sin embargo, Kaneko et al., (2000), identificaron 39 genes cromosomales involucrados en el proceso de nodulación y 46 en la fijación de nitrógeno.
2.3.3 Microorganismos diazótrofos asimbióticos.
Las bacterias diazótrofas asimbióticas son aquellas que pueden fijar nitrógeno atmosférico sin la necesidad de formar una simbiosis con plantas, ya que estas poseen diferentes estrategias para proteger el complejo nitrogenesa. Estas bacterias se encuentran prácticamente en todos los hábitats: suelo, mar, fuentes de agua dulce y sedimentos. Entre los principales géneros bacterianos que se hallan en vida libre o endófitos asociados a la rizosfera se encuentran:
Azotobacter spp., Azotococcus spp., Azospirillum spp., Beijerinckia spp., Azotomonas spp., Bacillus spp., Citrobacter spp., Clostridium spp., Chromatium
spp., Chlorobium spp., Desulfovibrio spp., Desulfomonas spp., Gluconacetobacter spp., Herbaspirillum spp., Klebsiella spp. (Rodríguez et al., 2003).
Magalãhes et al., (2001), aislaron y caracterizaron molecularmente 38 cepas fijadoras de nitrógeno de la rizosfera, hojas y tallos de cultivos de piña y banano en Rio de Janeiro (Brasil); caracterizaron cepas de Herbaspirillum seropedicae,
Herbaspirillum rubrisubalbicans, Burkholderia brasilensis, y Burkholderia tropicalis, mostrando la gran diversidad de bacterias fijadoras de nitrógeno asimbioticas en suelos tropicales. Torres et al., (2000), aislaron 18 cepas del género Azotobacter de 20 cultivos de arroz en el Tolima (Colombia), las cuales producían importantes concentraciones de de acido indol acético (AIA), incluyendo A. vinelandi con 32.22 ppm y A. chroococcum con 30.07 ppm de AIA.
miembros del géneros Beijerinckia, Azomonas y Agrobacterium. Tejera et al.,
(2005), aislaron cepas de A. chroococcum y Azospirillum spp. de la rizósfera de cultivos de caña de azúcar en Granada España, utilizando medio libre de nitrógeno (Burk`s) (Martínez et al., 1985) para su aislamiento, las cepas fueron identificadas y caracterizadas por medio de métodos convencionales (motilidad, producción de pigmentos, fermentación de azúcares, producción de PHBs y morfología de la colonia y celular) y moleculares (Rep- PCR y ARDRA).
Entre las bacterias diazótrofas asimbióticas utilizadas como biofertilizantes una de las más importantes es Azotobacter spp, la importancia agronómica de esta radica especialmente en la capacidad de producir antibióticos, sustancias estimulantes del crecimiento vegetal (SPCV) del tipo auxinas, giberelinas y citoquininas (Pandey & Kumar, 1990); además de la fijación de nitrógeno, producción de vitaminas, pigmentos, (Pandey et al., 1998) aminoácidos y otras moléculas con actividad biológica de interés industrial y comercial como polisacáridos (Sabra et al., 2001; Cuesta et al., 2006.). CORPOICA produce biofertilizantes a base de A. chroococcum (Monibac), su forma de presentación es líquida, su concentración es de 107 UFC/g y ha sido utilizado en cultivos de algodón, tomate y ají (CORPOICA, 2007 <online>). Bacterias del género
Azotobacter han sido utilizadas como bioinoculantes en cultivos agrícolas colombianos. El Instituto Colombiano Agropecuario (ICA) mediante la resolución 00375 define bioinoculante como aquel producto elaborado con base en una o más cepas de microorganismos benéficos que, al aplicarse al suelo o a las
semillas, promueven el crecimiento vegetal o favorece el aprovechamiento de
los nutrientes en asociación con la planta o su rizósfera (ICA, 2004), su uso
favorece a procesos limpios y rápidos de origen microbiano, están elaborados de diferentes grupos de microorganismos: bacterias, hongos y algas (Garzón et al., 2001).
El control de calidad y la vigilancia de estos productos en Colombia esta a
cargo del ICA, en general un bioinoculante debe cumplir con adecuada
concentración bacteriana, especificidad para la estimulación del crecimiento
inóculo debe presentar una concentración de 1x105 UFC/ml, sin embargo, estas concentraciones varían de acuerdo al cultivo a manejar. En Colombia existen diversas empresas encargadas en la producción e importación de estos bioinoculantes, entre las empresas registradas por el ICA se encuentra Agrotecnia LTDA, Biocultivos S.A, Eco Organics LTDA, Microagro LTDA, Safer Agrobiologicos, Fingifert Oriente Colombiano LTDA entre otras (ICA, 2007 <online>)
Neeru et al., (1991), utilizaron A. chroococcum como bioinoculante en la India para una variedad de cereales, oleaginosas y vegetales obteniendo resultados favorables en el peso seco de la planta y la producción de grano. Igualmente Galindo et al., (2006), inocularon bacterias diazotrófas (A. vinelandii) y solubilizadores de fosfato en plántulas de mangle y plantas de Citrullus vulgaris
en la isla de San Andrés (Colombia) incrementando la altura de las plantas, el número de hojas y de nodos. Los bioinoculantes en Colombia también se han utilizado en cultivos de crisantemo (Santana et al., 2002), pompón y clavel (FUNDASES, 2007 <online>).
García et al., (2005), mediante la inoculación de Azotobacter beijerinckii y Azospirillum spp. lograron aumentar la absorción de urea y la trasformación de los exudados radicales en SPCV, incrementando el peso fresco y seco del trigo inoculado. Estudios realizados en la India indican que los beneficios obtenidos en la inoculación de semillas con Azotobacter son marginales en suelos con pobre contenido de materia orgánica, pero muy favorables en suelos con mucha carga orgánica (Stevenson et al., 2000). Está documentado que bacterias del género Azotobacter y Azospirillum han utilizadas en sistemas de acuacultura y en producción de vermicompost para aumentar el contenido de nitrógeno y fósforo del producto (Garg et al., 2001; Kumar & Singh, 2001).
2.5 Familia Azotobacteraceae.
La familia Azotobacteraceae pertenece a la subclase gamma de las proteobacterias (Tchan, 1984), esta compuesta por bacterias fijadoras de nitrógeno de vida libre que comúnmente habitan en suelo, agua y sedimentos. Estudios de DNA ribosomal 16s (DNAr 16S) han identificado dos géneros en esta familia, Azotobacter y Azomonas El género Azotobacter se diferencia de
morfológicamente de muchas otros géneros de bacterias diazotrófas como
Azospirillum y Beijerinckia; comprende siete especies: A. chroococcum, A. vinelandii, A. beijerinckii, A. paspali (Doberienener & Day, 1975), A. armeniacus, A. nigricans (Tchan & New, 1984(a)) y A. salinestris (Page & Shivprasad, 1991). El género Azomonas comprende tres especies: A. macrocytogenes, A. agilis, y A. insignis (Tchan & New, 1984(b)).
Los miembros de esta familia Azotobacteraceae tienen la capacidad de sintetizar antibióticos y generar sustancias promotoras del crecimiento vegetal, (Pandey & Kumar, 1990) además de fijar nitrógeno no simbióticamente, especies como A. chroococcum y A. vinelandii son utilizadas como bioinoculantes en suelos tropicales y alcalinos. Igualmente muchos miembros de la familia
Azotobacteraceae son utilizados para producción de compuestos de interés comercial como polisacáridos, (Sabra et al., 2001) vitaminas y pigmentos (Pandey et al., 1998).
2.6 Generalidades del género Azotobacter spp.
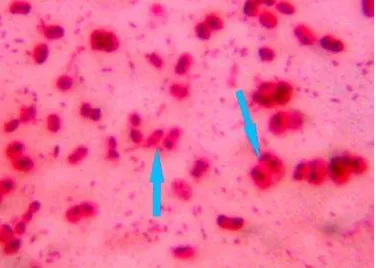
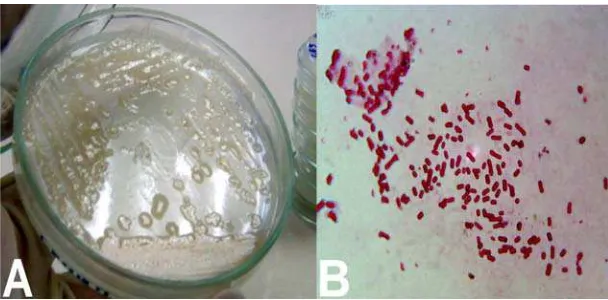
[image:26.612.226.414.549.683.2]Los microorganismos de este género comprenden bacterias con forma bacilar, reaccionan a la tinción de Gram como Gram negativos y en cultivos viejos como Gram variables, las células son ovoides y miden aproximadamente de 2µm a 4µm de diámetro, siendo las de mayor tamaño las de A. chroococcum que llegan a medir hasta 6µm, puede llegar a formar cadenas de tamaños variables, la forma de resistencia es el quiste (Figura 2), son aerobios pero algunos pueden vivir en tensiones bajas de oxígeno y su movilidad se debe a flagelos perítricos (Tabla 2), además, producen pigmentos solubles en agua en medios específicos (Tabla 1) (Figura 3) (Saribay, 2003).
En medio libre de nitrógeno con glucosa como única fuente de carbono, las células jóvenes de diferentes especies presentan una forma bacilar con extremos redondeados (Figura 4B). Las células de cultivos viejos tienden a ser elipsoidales y en ciertos casos es común observar gránulos sudanofílicos y metacromáticos (PHBs) (Tejera et al., 2005; Segura & Espín, 1998). Las colonias jóvenes de estos microorganismos son generalmente lisas, opacas poco convexas y viscosas (Figura 4A).
Figura 3. Colonias de diferentes especies de Azotobacter spp. en medio diferencial LG. A)
Azotobacter vinelandii DSM2289. B) Azotobacter armeniacus DSM2284 C) Azotobacter paspali
DSM2283. Fuente: Aquilanti et al., (a) (2004). Pigmentación de diferentes especies en medio Ashby con benzoato. D) Pigmentación café característica de A. armeniacus, A. chroococcum y A. nigricans. E) Pigmentación negra-café característica de A. nigricans y A. chroococcum. F) Pigmentación verde-amarillo característica de A. vinelandii y A. paspali. Fuente: Autor.
Tabla 1. Colores de pigmentos solubles en agua producidos por bacterias del género Azotobacter. Fuente: Holt, 2000
Pigmento A.
vinelandii
A. beijerinckii
A. paspali
A. armeniacus
A. nigricans
A. chroococcum
Amarillo-Verde
fluorescente + - + - - -
Verde D - - - - -
Café-Negro - - - - D D
Café-Negro a
Violeta-Rojo - - - + + -
Rojos-Violetas D - + + D -
[image:27.612.134.511.524.659.2]Figura 4. A) Colonias de Azotobacter spp. en medio libre de nitrógeno. B) Morfología de las células de Azotobacter spp. Fuente: Autor.
Bioquímicamente son catalasa y oxidasa positivo, reducen el nitrato, producen el sulfuro de hidrógeno e hidrolizan almidón, producen promotores de crecimiento (giberelinas, auxinas y citoquininas) (Santana et al., 2002), las bacterias de este género fijan asimbióticamente nitrógeno y son solubizadoras de fosfato, además, realizan procesos de biodegradación de plaguicidas como el endosulfan (Castillo
et al., 2005). Son quimioorganotróficas, utilizan para su crecimiento azúcares, alcoholes y sales inorgánicas. Son fijadores de nitrógeno en vida libre, fijan al menos 10 mg de N2 por gramo de carbohidrato consumido (Holt, 2000).
Requieren molibdeno para fijar nitrógeno que puede ser parcialmente reemplazado por vanadio. Al igual que los demás fijadores de nitrógeno
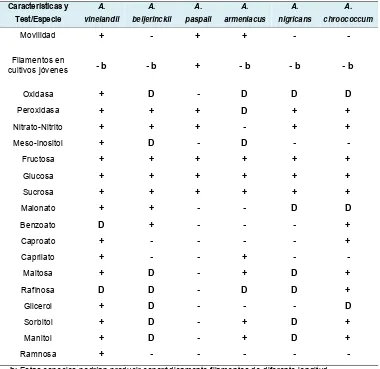
[image:28.612.167.471.78.228.2]Tabla 2. Movilidad, formación de filamentos y pruebas bioquímicas utilizadas para la caracterización del género Azotobacter spp. Fuente: Holt, 2000
Características y Test/Especie A. vinelandii A. beijerinckii A. paspali A. armeniacus A. nigricans A. chroococcum
Movilidad + - + + - -
Filamentos en
cultivos jóvenes - b - b + - b - b - b
Oxidasa + D - D D D Peroxidasa + + + D + + Nitrato-Nitrito + + + - + + Meso-inositol + D - D - - Fructosa + + + + + + Glucosa + + + + + + Sucrosa + + + + + + Malonato + + - - D D Benzoato D + - - - + Caproato + - - - - + Caprilato + - - + - - Maltosa + D - + D + Rafinosa D D - D D + Glicerol + D - - - D Sorbitol + D - + D + Manitol + D - + D + Ramnosa + - - - - - - b: Estas especies podrían producir esporádicamente filamentos de diferente longitud
D: 11% - 89% de las cepas son positivas +: 90% o más de las cepas son positivas - : 90% o más de las cepas son negativas
Respecto a la fuente de nitrógeno pueden utilizar nitrato, sales de amonio y aminoácidos, pero ellos pueden además fijar el nitrógeno del ambiente (Garzón
2.6.1 Azotobacter chroococcum.
En medios libres de nitrógeno A. chroococcum produce un pigmento café-negro no difusible, estos se producen en presencia de benzoato. También produce pigmentos grises-cafés en medios adicionados con 0.2% de gluconato. Sobre medios libres de nitrógeno esta bacteria forma colonias mucilaginosas pardas las cuales aparecen a las 48 horas a 30ºC. A. chroococcum presenta colonias mucosas opacas, inicialmente el color del pigmento es claro y brillante, pero a medida que la colonia se desarrolla se torna de color café oscuro, la fuente principal de carbono es el manitol (Santana et al., 2002). A. chroococcum
puede llegar a crecer en un pH alrededor de 5.5 (Saribay, 2003).
A. chroococcum puede utilizar diferentes fuentes de nitrógeno inorgánico como amonio, nitrato, nitrito o dinitrógeno, este microorganismo realiza la asimilación de nitrógeno en tres pasos: transporte del nitrato dentro de la célula, reducción del nitrato a nitrito (Nitrato reductasa) y la reducción de nitrito a amonio (Nitrito reductasa), sin embargo, estos pasos requieren de dos condiciones nutricionales, la ausencia de amonio (represor) y la presencia de nitrato o nitrito (inductores). Se ha reportado la presencia de dos polipéptidos con masas moleculares de 22kDa (P22) y 35kDa (P35), la expresión de estos genes es regulada por las fuentes de nitrógeno. La P22 esta asociada a la membrana citoplasmática y es fosforilada en respuesta al nitrato, este polipéptido es una proteína sensorial para la asimilación de nitrato en A. chroococcum (Muñoz et al., 1996).
endosulfán por medio de enzimas deshalogenasas, dioxigenasas e hidroxilasas (Castillo et al., 2005). Por otro lado, Sudhir et al.,(1983), reportaron que A. chroococcum tiene la capacidad de inhibir el crecimiento de Rhizoctonia solani
en cultivos de papa a temperaturas de 15°C. Bansal et al., (1999) determinaron el efecto de bacterias rizosféricas en cultivos de maíz infestados con el nematodo patógeno Heterodera avenae, reportando que la máxima reducción de infección la produjo A. chroococcum con un 48% seguido por Pseudomonas con 11% y Azospirillum con 4%.
2.6.2 Azotobacter vinelandii.
Azotobacter vinelandii es una bacteria poliploide, es decir posee varias copias de su cromosoma, se calcula que pueden tener hasta 80 copias. El número de copias varía dependiendo del medio y las condiciones de cultivo así como de la fase de crecimiento. Es de tamaño muy grande de 2 a 5 µm de diámetro es decir de 5 a10 veces el volumen de E. coli, se ha asociado el tamaño con la poliploidía (Nagpal et al., 1989).
La capacidad metabólica y genética por las que A. vinelandii ha sido y es objeto de estudio en biofertilización y biotecnología incluyen la fijación de nitrógeno en presencia de oxígeno por tres sistemas diferentes de nitrogenasa; presencia de mecanismos de protección de la nitrogenasa ; alta capacidad respiratoria que en condiciones diazotróficas o de fijación de nitrógeno es hasta 10 veces más alta que la de E. coli ; la formación de estructuras de resistencia frente al estrés ambiental (quistes) y la producción de polímeros de uso industrial como el alginato y el polihidroxibutirato (Espin, 2002). A. vinelandii fija nitrógeno en aerobiosis gracias a que posee un sistema bien integrado de protección de su
nitrogenasa que comprende: protección conformacional, protección respiratoria, autoprotección y otros cambios morfológicos y fisiológicos que le permiten crecer diazotróficamente en condiciones totalmente aeróbicas (Manchal et al., 2000).
A. vinelandii posee tres tipos de nitrogenasas diferentes para la fijación de nitrógeno; la nitrogenasa 1 codificada por el gen nifHDK, la nitrogenasa 2 es dependiente de vanadio y los genes que codifican para las dos proteínas están designados con el nombre de vnf y se encuentran en los operones vnfHorfFd y
molibdeno y vanadio esta codificada por el gen anfHDK (Figura 5) (Betancourt, 2002). La comparación de las secuencias entre estos genes indican que el porcentaje de identidad entre los genes nifH y vnfH es del 88.5%, sugiriendo que provienen de un gen ancestral. Por otro lado, el porcentaje de similitud entre las secuencias de los genes nifH y anfH se encuentra alrededor del 70%. Los genes vnfDK y anfDK muestran una mayor similitud en su secuencia al compararlos con lo genes nifDK, reportando un porcentaje de similitud de 65.8% entre los genes vnfK y anfK y una similitud de 65.8% entre los genes vnfD y anfD
(Joerger et al., 1989; Joerger et al., 1990)
Figura 5. Esquema de la organización de los genes involucrados en la síntesis de las tres nitrogenasas utilizadas por A. vinelandii en la fijación de nitrógeno. Fuente: Betancourt, 2002
2.7 Técnicas moleculares para la caracterización e identificación de
bacterias diazotrófas
Diversos estudios se han realizado en la caracterización, identificación y taxonomía de la diversidad biológica de bacterias diazotrófas. Las técnicas moleculares mas utilizadas a nivel mundial son DGGE (Denaturing Gradient Gel Electrophoresis), RFLP (Restriction Fragment Lenght Polymorphisms), ARDRA (Amplified Ribosomal DNA Restriction Analysis), RAPD (Random Amplified Polymorphic DNA) y la secuenciación. Estas técnicas permiten conocer la
DGGE para evaluar la diversidad de bacterias diazótrofas en cultivos de Spartina alternifora y realizaron una PCR de genes nifH, logrando identificar bacterias diazótrofas de la subdivisión beta de las proteobacterias. Yeager et al., (2004), realizaron un T-RFLP del gen nifH utilizando la enzima RsaI para caracterizar la comunidad diazótrofa en un bosque australiano el cual había sufrido un incendio forestal, reportando cepas diazótrofas del genero Clostridium y Paenibacillus
R`sch et al., (2002) utilizaron la secuenciación de los genes nifH, nosZ, nirS y
nirK para realizar un estudio de diversidad de bacterias diazótrofas y desnitrificantes en bosques de suelos ácidos en Alemania. Igualmente Roesch et al., (2006), realizaron una caracterización de bacterias diazótrofas asociadas a cultivos de maíz, amplificando el gen nifH del DNA extraído directamente del suelo con iniciadores nifHFor y nifHRev, y realizaron un análisis RFLP con enzimas de restricción TaqI y HaeIII, reportando bacterias del género
Herbaspirillum, Azoarcus, Burkholderia y Azospirillum. Además de esto, Soares
et al., (2003), realizaron una comparación entre RFLP, RAPD y secuenciación parcial del gen DNAr 16S en cepas de Herbaspirillum en Curitiba (Brasil). Muchos estudios han dado por manifiesto la importancia de las nuevas técnicas moleculares en la caracterización de bacterias diazótrofas, sin embargo, estos estudios son una pequeña muestra de la gran cantidad de investigaciones y de herramientas moleculares para la caracterización e identificación de bacterias diazótrofas.
2.7.1 Aplicaciones del DNA ribosomal 16S y su utilización en filogenia y
taxonomía
El DNAr 16S es un polirribunucleotido codificado por el gen rrs también denominado RNA ribosomal 16S, apartir de cuya secuenciación genética se puede obtener información filogenética y taxonómica. Como cualquier secuencia de nucleótidos de cadena sencilla, el DNAr 16S se pliega en una estructura secundaria, caracterizada por la presencia de segmentos de doble cadena, alternando con regiones de cadena sencilla (Neffs et al.,1990). En eucariotas el DNAr 18S es la macromolécula equivalente. Dado que los DNAr 16S y 18S proceden de las subunidades pequeñas de los ribosomas, el acrónimo DNAr
comunes a todos los organismos, pero contienen variaciones que se concentran en zonas específicas. El análisis de la secuencia de los DNAr 16S de distintos grupos filogenéticos reveló un hecho adicional de gran importancia práctica: La presencia de una o más secuencias características que se denominan oligonucleótidos firma, se trata de secuencias específicas cortas que aparecen en todos (o en la mayor parte de) los miembros de un determinado grupo filogenético, y nunca (o sólo raramente) están presentes en otros grupos, incluidos los más próximos. Por ello, los oligonucleótidos firma pueden utilizarse para ubicar a cada bacteria dentro de su propio grupo (Rodicio & Mendoza., 2004; Heyndrickx et al., 1996).
La comparación de las secuencias del DNAr 16S es una herramienta poderosa para deducir la filogenia, la evolución y la relación entre bacterias, arqueobacterias y organismos eucariotas. Estas secuencias han sido derivadas previamente de métodos como catalogación de oligonucleotidos, secuenciación de clones, secuenciación directa de DNA usando trascriptasa reversa y secuenciación de material amplificado por reacción de cadena de polimerasa (PCR) (Weisburg et al., 1991). Aunque existen cronómetros moleculares alternativos al DNAr 16S, hasta el momento ninguno ha conseguido desplazarlo.
Este marcador molecular presenta una serie de ventajas: 1) está presente en todos los organismos y tiene la misma función en todos ellos; 2) debido a restricciones estructurales, diferentes regiones de la molécula presentan distinto grado de variabilidad en secuencia, lo que permite realizar comparaciones con diferente nivel de resolución; 3) su transmisión es principalmente vertical ya que se considera que no está sujeto a transferencia génica horizontal entre microorganismos; 4) la longitud de su secuencia tiene un tamaño adecuado como para proporcionar suficiente información, con un coste asumible, y 5) el análisis de la secuencia permite realizar reconstrucciones filogenéticas de los microorganismos. (Rodicio & Mendoza, 2004; Nogales, 2005).
las de ningún género o especie reconocido, esta situación puede presentarse cuando se trata de nuevas especies; cepas de especies comunes que exhiben un perfil bioquímico ambiguo; bacterias para las cuales la caracterización fenotípica sea sustancialmente deficiente; bacterias fastidiosas, a consecuencia de sus requerimientos nutricionales complejos y bacterias de crecimiento lento, que retrasa considerablemente la identificación convencional. (Rodicio & Mendoza, 2004)
2.7.2 Análisis de restricción del DNA ribosomal 16S amplificado (ARDRA)
Una alternativa para buscar la información filogenética y taxonómica que encierra el DNAr 16S es el análisis del patrón electroforético obtenido después de la digestión del gen con enzimas de restricción. La relación matemática entre el número de los sitios compartidos por la enzima de restricción o de los fragmentos compartidos del DNA es lo que entra en discusión para identificar la divergencia genética. Los modelos matemáticos para hallar la similaridad de los patrones de corte se basan en el uso de matrices binarias utilizando coeficientes de similitud como el de Dice, Jaccard, Nei & Li, entre otros. Estos coeficientes permiten agrupar a los morfotipos en clústers que identifican la distancia genética mediante el uso de métodos de análisis de agrupamiento tales como el Neigboring-Joing y UPGMA. En los últimos años, la amplificación del DNAr 16S y el análisis de restricción se ha realizado en diferentes géneros como:
Clostridium, Streptocooccus, Mycobacteria, Moraxella, Leptospira Rhizobia,
Brevibacterium, Acinetobacter, entre otros (Heyndrickx et al., 1996).
2.7.3 Caracterización, identificación y filogenia del género Azotobacter y
otras bacterias diazótrofas mediante ARDRA
restricción ya que permiten simular los cortes de una enzima en determinada secuencia, y de esta forma distinguir las enzimas que pueden proporcionar una mayor información. Existen diferentes programas en internet para realizar restricciones virtuales, entre los que se encuentran el FastPCR, CLC Gen Worbech 2.2.4 y el Webcutter 2.0, además, existen diferentes enzimas de restricción reportadas para la caracterización de diazótrofas. Aquilanti et al., (a) (2004), compararon diferentes métodos de aislamiento preliminar y tomaron muestras de suelo en el centro de Italia para realizar un análisis ARDRA, utilizando los iniciadores universales 27f y1495r, que amplificaban el DNAr 16S completo (1500 bp) para posteriormente realizar una digestión con las enzimas de restricción RsaI y HhaI, identificando miembros de la familia
Azotobactereaceae, además especies del género Azomonas, Beijerinckia, Azospirillum y Agrobacterium, reportando que la enzima Rsa I genera un patrón de 5 bandas en la electroforesis para las especies del género Azotobacter.
De la misma forma, Magalãhes et al., (2001), realizaron una caracterización de diazótrofas mediante ARDRA en aislamientos de muestras de suelo obtenidas en cultivos de piña y banano utilizando los iniciadores universales Y1 y Y3 que amplifican aproximadamente 1500 pb, utilizando enzimas de restricción AluI, HaeII, HinfI y RsaI identificando bacterias diazotróficas: Herbaspirillum seropedicae, Herbaspirillum rubrisubalbicans, Burkholderia brasilensis, y
Burkholderia tropicalis. Tejera et al., (2005), amplificaron el DNAr 16S utilizando los iniciadores universales 41f y 1488r para especies de Azotobacter y utilizaron las enzimas TaqI, NdeII y MspI para el análisis de restricción, reportando la presencia de A. chroococcum y Azospirillum brasilense en cultivos de caña de azúcar.
Perin et al., (2006), realizaron una caracterización de bacterias diazótrofas del género Burkholderia aisladas de cultivos de maíz y caña de azúcar en México y Brasil amplificando el DNAr 16S con los iniciadores universales para el dominio bacteria fD1 y rD1 utilizando las enzimas AluI, DdeI, HaeIII, HhaI, HinfI, MspI, y
RsaI. Junior et al., (2004), realizaron una identificación de cepas de Azospirillum
los aislados fueron agrupados por el método de medias distancias y representados gráficamente por un dendrograma con el software NTSYS 2.0.
Aquilanti et al., (b) (2004), realizaron un análisis de restricción del DNAr 16S para la caracterización de la familia Azotobactereaceae, utilizando iniciadores universales 27f y 1495r amplificando 1500 bp y posteriormente utilizando unos iniciadores internos Y1 y Y2 amplificando 300 bp en el extremo 5', para después realizar la secuenciación del gen, utilizaron las enzimas RsaI, HhaI, HpaII, FnuDII y AluI e identificaron las diferentes especies del genero Azotobacter,
seleccionaron las enzimas con ayuda de una restricción virtual del gen DNAr 16S de secuencias de Azotobacter sp. y Azomonas sp. tomadas del Genbank, utilizaron el programa Genetool software (Version 2.0) (Wishart et al., 2000), los datos los organizaron en una matriz binaria de presencia (0) ausencia (1) de bandas en los patrones de corte. Los diferentes patrones de corte de cada cepa caracterizada con cada enzima fueron diferenciando y definiendo los haplotipos y la combinación de los haplotipos obtenidos con las 5 enzimas fueron agrupados generando los filotipos (Tiedje et al., 1999).
Asi mismo, Aquilanti et al., (b) (2004), hallaron la diversidad de los haplotipos, la cual fue estimada usando el coeficiente de Shannon–Weaver: H= - £pi lnpi,
donde pi es la frecuencia de cada haplotipo (Shannon & Weaver, 1949). La distancia genética entre los filotipos fue calculada utilizando el coeficiente de Nei y Li (Nei & Li, 1979), y el resultado de la distancia en la matriz fue usado para el análisis UPGMA y el análisis Neighbor-joining mid-point. La secuencia del gen 16S en todos los aislamientos estudiados fue comparada empleando el programa Basic BLAST y los alineamientos usando el programa CLUSTALW (Higgins et al., 1992) disponible en http://www.ebi.ac.uk/clustalw/. Los autores reportaron el porcentaje de polimorfismo de cada enzima (RsaI 16%, HhaII 24%,
AluI 23%, HpaII 22%, FnuDII 15%) y concluyeron que la enzima RsaI posee un bajo porcentaje diversidad 1.79, comparado con las enzimas HpaII y AluI
3. FORMULACION DEL PROBLEMA Y JUSTIFICACION
Los procedimientos convencionales para la caracterización e identificación de microorganismos diazótrofos empleando características fenotípicas, (forma bacteriana, presencia de flagelos y test de Gram) bioquímicas (asimilación de azucares, crecimiento en medios libres de nitrógeno, pruebas de oxidasa y catalasa) y fisiológicas (crecimiento a diferentes temperaturas, condiciones atmosféricas, concentración de sal y valores de pH) son métodos que permiten identificar solamente algunos géneros de bacterias cultivables presentes en un agroecosistema, además, son métodos que manifiestan un mayor grado de ambigüedad entre especies y por esto la identificación y caracterización se hace mas difícil.
Los microorganismos diazótrofos, descritos desde el siglo pasado, han sido objeto no solo de estudio en microbiología de suelos, sino también en el desarrollo de productos biológicos comerciales, especialmente en Cuba, México y Estados Unidos. En este momento en Colombia, donde se han aislado numerosas cepas asociadas a cultivos de importancia agrícola (arroz, caña, maíz, hortalizas, algodón, banano, flores, etc.,), se cuenta con productos comerciales registrados que contienen bacterias fijadoras de nitrógeno de vida libre o simbiótica, con miras a ser utilizados para reducir o complementar la fertilización química nitrogenada. Sin embargo, el control de calidad de estos productos y la caracterización de los aislamientos se dificultan por la similitud macro y microscópica de los mismos, haciendo que los análisis moleculares sean la única herramienta realmente precisa.
4. OBJETIVOS
4.1 Objetivo general.
9 Caracterizar molecularmente cepas nativas Colombianas de Azotobacter
spp. aisladas de cultivos de hortalizas mediante el Análisis de Restricción del DNA Ribosomal 16S Amplificado (ARDRA)
4.2 Objetivos específicos.
9 Aislar cepas nativas colombianas de Azotobacter spp. en suelos de cultivos de hortalizas ubicados en el departamento de Boyacá
9 Identificar fenotípica y bioquímicamente cepas de Azotobacter spp. aisladas de suelos de cultivos de hortalizas ubicados en el departamento de Boyacá.
9 Estandarizar un protocolo para extraer DNA total y amplificar el DNA
ribosomal 16S de cepas nativas de Azotobacter spp.
9 Analizar y comparar los patrones de corte generados por las enzimas de
restricción (AluI, HpaII y RsaI) sobre el DNA ribosomal 16S amplificado a partir de cada uno de aislamientos.
5. METODOLOGIA
5.1 Lugar de muestreo
El muestreo de suelo se realizó en los municipios de Sogamoso y Tibasosa en el departamento de Boyacá. Se muestrearon 6 cultivos de hortalizas (brócoli, calabacín, coliflor, espinaca, tomate y zanahoria).
Se obtuvieron muestras de suelos de cultivos de calabacín (Cucurbita pepo), espinaca (Spinacia oleracea) y tomate (Lycopersicon esculentum Mill) en la granja agroindustrial de Cementos Paz del Rio en el municipio de Sogamoso ubicado a 170 Km de la ciudad de Bogotá, la cual cuenta con una ubicación geográfica de 5°43’41’’de latitud norte y 72°55’13’’ longitud oeste, con una temperatura promedio de 17°C y una altura de 2500 msnm. Las muestras de suelos de los cultivos de brócoli (Brassica oleracea itálica); coliflor (B. oleracea botrytis) y zanahoria (Daucus carota var. Sativus) fueron colectadas en una finca particular productora de hortalizas del municipio de Tibasosa, ubicado geográficamente a 5°44’00’’ latitud norte y 73°00’04’’ longitud oeste, cuenta con una temperatura promedio de 15°C y una altura de 2748 msnm. (Figura 6)
Figura 6. Lugar de muestreo en el departamento de Boyacá.
5.2 Muestreo del suelo
De cada cultivo de hortalizas se tomaron cinco muestras de 500g (Figura 7A) a una profundidad de 10 a 15 cm (Martyniuk & Martyniuk 2003; Tejera et al.,
[image:42.612.141.512.400.600.2]al., 2000). Las muestra se empacaron en bolsas de papel que se colocaron dentro de bolsas de plástico herméticas y posteriormente fueron trasportadas en neveras de icopor a temperatura ambiente al laboratorio de Microbiología Ambiental y Suelos de la Pontificia Universidad Javeriana (Figura 7B).
Figura 7. Recolección de las muestras de suelo. A) Muestras de 500 gramos; B) Trasporte de las muestras en bolsas herméticas y neveras de icopor. Fuente: Autor.
5.3 Procesamiento de muestras
Una vez en el laboratorio, las muestras de suelo fueron tamizadas en un tamiz estándar de 4.75 mm (Aquilanti et al., 2004 (a)), con el fin de obtener gránulos uniformes, para realizar el aislamiento primario.
5.3.1 Medición de pH de las muestras de suelo
La medición del pH se realizó siguiendo el protocolo descrito por Van Lierop, 1981; se colocó en un frasco de vidrio limpio suelo tamizado y agua destilada en proporción 1:1 p/v. Posteriormente, la suspensión se dejó precipitar durante 5 min y se tomó el valor de pH del sobrenadante con un potenciómetro OAKTON®. Los valores de pH fueron promediados a partir de cuatro mediciones en cada cultivo.
5.3.2 Aislamiento primario
(% de recuperación = No. de gránulos con colonias mucilaginosas/total de gránulos sembrados X 100) y se realizó coloración de Gram.
Figura 8. Aislamiento primario. A) Siembra de gránulos en agar Ashby-Manitol; B) gránulos mucilaginosos después de la incubación. Fuente: Autor.
5.4 Aislamiento secundario
El aislamiento secundario se realizó mediante siembra por agotamiento en cajas con Agar Ashby-Sacarosa a partir de las colonias obtenidas en el aislamiento primario. Las cajas fueron incubadas por 7 días a 28°C, posteriormente se observó el crecimiento en el medio selectivo Ashby-Sacarosa, la morfología de la colonia y de las células por medio de la coloración de Gram. (Tejera et al., 2005). Las cepas aisladas que presentaron crecimiento en el segundo aislamiento se codificaron de acuerdo al cultivo de donde fueron aisladas.
5.4.1 Conservación de los aislamientos mediante la técnica de
criopreservación en glicerol al 50% (v/v)
A partir de las cepas aisladas y evaluadas se realizaron cultivos en erlenmeyer de 100ml con 50ml de caldo nutritivo (Anexo 2), el cual se incubó a 28°C por 24 horas con agitación de 150 rpm. El cultivo fue depositado en 10 viales eppendorf de 1.5ml con 50% v/v de glicerol puro y estéril. Los tubos eppendorf se almacenaron en bolsas ziploc a - 80°C (Aquilanti et al., 2004 (a) Poutou et al., 1994).
5.4.2 Pigmentación de los aislamientos
realizando una estría en toda la caja, se incubo a 28°C por 7 días y se observó pigmentación (Figura 9). (Martyniuk & Martyniuk 2002)
.
Figura 9. Siembra en estria en agar Ashby con benzoato. Fuente: Autor.
5.4.3 Identificación Bioquímica
[image:45.612.249.396.126.220.2]A cada aislamiento se le realizó una caracterización con base en el comportamiento bioquímico frente a la fermentación de glucosa, maltosa, manitol y ramnosa (Anexo 3) también se realizó una prueba de asimilación y crecimiento en benzoato como fuente de carbono, test de catalasa, oxidasa y desnitrificación en caldo nitrato (Holt, 2000; Tejera et al., 2005). La batería de azúcares fue sembrada con una colonia de cada cepa, se llevó a incubar a 28°C por 24 horas. Posteriormente se verificó la acidificación del medio como indicativo de la fermentación de azúcares y benzoato (Figura 10). Finalmente se realizó la prueba de Nessler para determinar la desnitrificación, y las pruebas de catalasa y oxidasa a partir de las colonias. (Anexo 4). Todas las pruebas bioquímicas se realizaron por triplicado.
Figura 10. Baterías bioquímicas de azucares sin sembrar. A) Baterías bioquímicas servidas en placas de 24 pozos; B) Baterías bioquímicas servidas en tubos de 13X100. Fuente: Autor.
5.4.4 Cepa control
5.5 Extracción de DNA
Inicialmente la extracción de DNA se realizó siguiendo el protocolo descrito por Aquilanti et al., (a) (2004) Para esto, se tomaron tres colonias y se suspendieron en 500µl de agua destilada estéril. Se llevaron a centrifugar a 5000 rpm por 5 min, el “pellet” obtenido se resuspendió en 100µl de agua estéril más 100µl de Tris-HCL 10mM a pH 8.2. Finalmente se adicionó 0.65µl de proteinasa K (20mg/ml), se llevó a incubar a 55°C por 2 horas, posteriormente se centrifugó a 5000 rpm por 5 min y se tomó 2µl del sobrenadante para la reacción de amplificación (PCR).
Considerando que la extracción a partir de colonias no fue exitosa se procedió a realizar la extracción de DNA a partir de medio de cultivo liquido, siguiendo el protocolo propuesto por Maloy (1989) (http://www.research.umbc.edu), que utiliza solventes orgánicos como Fenol Cloroformo alcohol Isoamílico (FCI) y Cloroformo alcohol Isoamílico (CI) para la remoción de proteína. Este protocolo ha sido utilizado con éxito por investigadores del Instituto Amazónico de Investigaciones Científicas (SINCHI) para la extracción de DNA de bacterias diazótrofas. El procedimiento consistió en realizar una suspensión a partir de una colonia de Azotobacter en 5ml de caldo nutritivo en tubo falcón de 15ml la cual se llevó a incubar de forma inclinada 48 horas a temperatura ambiente a 150 rpm. Posteriormente el cultivo se colocó en 2 tubos eppendorf de 1.5ml (c/u con 1.5ml del cultivo); se centrifugaron a 13000 rpm por 2 min y se desechó el sobrenadante.