Reservados todos los derechos © El autor
han sido los más importantes. Es el momento pues, de hablar de todo y todos
los que estáis detrás de este trabajo, de ensalzar la grandeza de lo cotidiano, de
enseñar la magia de estos años, en definitiva, de dar las gracias… Y es que
sobre lo elemental también se escriben odas, para ayudarnos a valorar lo que es
realmente importante.
La primera de ellas va dirigida a mis directoras, las Dras. M. Carmen
Mendoza y M. Rosario Rodicio, que han permitido que yo pudiera realizar
este camino. Ya hace seis años desde aquel primer pasito: entrar en el despacho
de la Dra. M. Carmen Mendoza para pedirle consejo e información. Y de
todo este tiempo compartido me quedo con la paciencia, espíritu luchador,
calidad profesional y humana que ambas demuestran día a día. Gracias por
vuestra dedicación y sabias enseñanzas, pero sobre todo, gracias de corazón por
vuestro cariño y por querernos siempre como verdaderos “hijos en la ciencia”.
Las Dras. M. Cruz Martín y Rosaura Rodicio han protagonizado
muchas odas elementales, colaborando en el “cacharreo” del trabajo, estando
siempre dispuestas a echarme una mano cuando más apurada estaba. De
vosotras he tenido la suerte de aprender trucos de la cocina del laboratorio,
siempre demostrando experiencia e ingenio a la hora de resolver un problema.
Sin vuestra ayuda esta tesis no hubiera sido posible.
hoja verde
caída en la ventana,
minúscula
claridad
recién nacida,
elefante sonoro,
deslumbrante
moneda,
a veces
ráfaga quebradiza,
pero
más bien
pan permanente,
esperanza cumplida,
deber desarrollado.
(…)
Y de puertas hacia dentro del laboratorio es donde se cocina una tesis. Cada día, cada persona cuenta, y
continuamente se aprende. Ahí se dan lecciones importantes y se ganan grandes amistades…La mayor parte de
esta tesis se realizó en el antiguo laboratorio B, y a los que fueron sus inquilinos en el pasado van dedicadas mis
primeras odas: Ana del Cerro, Ana Herrero, Lucía, Aurora, Javi y Bea, brindo por los buenos momentos
vividos. A los que son hoy miembros del grupo: Esther, Roberto, Pili, David y Jéssica, gracias por hacer que
todo esto haya merecido la pena. A Pili y David, por poder contar siempre con vosotros, porque me hacéis reír
cuando más lo necesito, por tantas muestras de cariño… vuestros abrazos son verdaderas odas elementales. A
Jéssica porque siempre estás ahí, aportando buenas ideas y siendo una gran compañera de viaje, me quedo con
los infinitos trayectos Gijón-Oviedo compartidos, las innumerables charlas y risas, por escucharme y animarme
siempre. Una oda muy especial se la dedico a Noe, mi primera maestra en el laboratorio, de la que más he
aprendido en todo este tiempo, porque siempre puedo contar con tu paciencia y amistad dentro y fuera del
laboratorio.
Ahora comparto mi día a día con mis compañeros del laboratorio M1, con quienes se aprende que cada
momento se puede acompañar de una dosis de buen humor. Y sois Marián, Mateo, Patri y Nacho los que lo
hacéis posible, gracias de corazón por toda vuestra ayuda, por hacer que afrontara la última etapa de mi tesis
con energías renovadas y sobre todo, por vuestra cariñosa acogida. Un guiño especial para Patri, por esa sonrisa
que todo lo ilumina... Gracias también a toda la gente del Área de Microbiología que siempre me ha echado una
mano cuando grité “¡ayuda!” en tantas ocasiones…
He tenido la suerte de poder hacer estancias en otros laboratorios, y de escribir así más odas elementales
con la gente que me ha acompañado en mis viajes. En primer lugar, gracias a Aurora Echeita por darme la
oportunidad de ir al Centro Nacional de Microbiología, porque me hiciste sentir como en mi “casa” y por sacar
siempre un huequecito para mí. Una oda muy especial también a Silvia, que siempre respondes con una sonrisa
a todas las consultas y por ayudarme a que me integrara rápidamente en el grupo. Un abrazo para Cris,
Rubén, Sylvia, Raquel y el resto de compañeros del CNM.
Luego vendría Berlín, que se convirtió en mi hogar por más tiempo del esperado… Esos milagros sólo
los puedes obrar tú, Bea. Gracias de todo corazón por todo lo que me has enseñado, por haberme cuidado y
porque en ti encontré una amiga además de una jefa. Reiner, thanks to your kindness I felt very comfortable in
the BfR and also in Berlin. Thank you for helping me in time of need. Janine, thank you for all your help
inside and outside the lab, but above all, I am very happy of having found your friendship in Berlin; I hope we
will continue sharing good moments together. Además, hay personas con las que compartí grandes momentos
allí, con las que el día a día se deshacía en risas y charlas, y con las que cualquier plan, improvisado o no, es
maravilloso: Sara, Alessandro y Yinth, todo mi cariño.
Las odas elementales inundan mi vida porque tengo magas, magos y hadas a mi alrededor que me
enseñan a ver la luz en cada detalle…Así mis niñas del sur, Patri, Cova yMirta, con su varita mágica y sus
conjuros son capaces de llenarlo todo de color y alegría…mis niñas, a cada una os corresponde un pedacito de
esta tesis, y mi corazón entero es vuestro. No me olvido de un duendecillo aventurero que ahora está por
Senegal…Javi, aunque no lo creas, también tú estás escondido entre las líneas de este trabajo. Te echamos de
menos…Mi corazón sigue dividido, y ahora viaja hasta Madrid, porque con quién mejor que con Jesús para
compartir viajes, cafés y aventuras riéndose hasta desfallecer…Gracias por tantos años de amistad y por hacer
que los kilómetros sean sólo números y no distancia.
También hay magas en Gijón, las más importantes, las que siempre han estado y siguen estando ahí
cuando más lo necesito. Conchi, Lorena, Irene, Natalia y Meri…dais luz, color y sentido a todo, sin vosotras,
sin vuestra risa y forma de ser, nada sería igual. En especial a mi tocayina, por nuestras vidas paralelas y por
encontrarnos en el instituto. A ti te dedico una oda de fieltro y tul. Y Meri… qué te voy a decir, ¡si tú me
enseñaste las odas elementales! soy como el agua para ti y seguro que me lees el pensamiento ahora mismo…por
supuesto gracias por toda tu ayuda en el diseño de esta tesis…pero sobre todo gracias por tu amistad, por
escucharme siempre, por estar en los buenos y malos momentos, por ser como eres. Os llevo en el corazón mis
niñas…¡que siga la magia!
Pero soy lo que soy por mis orígenes, por lo que me han enseñado que no está en los libros, por el amor
incondicional que recibo todos los días. Mamá, papá, gracias. Sin vuestra confianza no hubiera llegado hasta
aquí. El camino no ha sido fácil pero vosotros lo habéis hecho posible, ayudándome a levantarme cuando me he
caído, demostrándome vuestro cariño, apoyándome continuamente y enseñándome a vivir entre amor y
serenidad. Os quiero.
Son odas elementales también los que un día fueron. A mis abuelos, fuentes inagotables de sabiduría, por
el amor que siempre dieron y por crear firmes pero acogedoras raíces donde se apoyaron y se apoyan nuevas
vidas.
A todos los que no menciono pero que han sido tan importantes…
A los herrerillos, todos ellos mágicos…
ABREVIATURAS
... vRESUMEN
Español ... viiEnglish ... viii
1. INTRODUCCIÓN
... 1.a. RESISTENCIA A ANTIMICROBIANOS ... 11.b. BASES BIOQUÍMICAS DE LA RESISTENCIA EN BACTERIAS GRAM NEGATIVAS ... 2
1.b.1. ENZIMAS INACTIVADORAS Y MODIFICADORAS ... 2
1.b.2. EXPULSIÓN ACTIVA DEL ANTIBIÓTICO ... 5
1.b.3. MODIFICACIÓN DE LA DIANA CELULAR ... 6
1.c. BASES GENÉTICAS DE LA RESISTENCIA ... 9
1.d. MUTACIÓN ... 10
1.e. TRANSFERENCIA GÉNICA HORIZONTAL ... 10
1.e.1. CASETES GÉNICAS ... 11
1.e.2. INTEGRONES ... 12
1.e.3. ELEMENTOS GENÉTICOS TRANSPONIBLES ... 17
1.e.4. PLÁSMIDOS ... 23
1.e.5. ELEMENTOS INTEGRATIVOS Y CONJUGATIVOS ... 24
1.f. Salmonella COMO OBJETO DE ESTUDIO ... 26
1.f.1. CARACTERÍSTICAS GENERALES ... 26
1.f.2. ESTRUCTURA ANTIGÉNICA: SEROTIPIFICACIÓN ... 27
1.f.3. CUADROS CLÍNICOS ... 30
1.g. VIRULENCIA ... 32
1.g.1. ESTRUCTURAS DE SUPERFICIE ... 33
1.g.2. ISLAS DE PATOGENICIDAD ... 35
1.g.3. ISLOTES DE PATOGENICIDAD Y GENES SUELTOS ... 38
1.g.4. PLÁSMIDOS DE VIRULENCIA ... 39
3. RESULTADOS
...3.a. ARTÍCULO 1 ... 45
Rodríguez, I., M. R. Rodicio, M. C. Mendoza y M. C. Martín. 2006. Large conjugative plasmids from clinical strains of Salmonella enterica serovar Virchow contain a class 2 Integron in addition to class 1 integrons and several non-integron-associated drug resistance determinants. Antimicrob. Agents Chemother. 50: 1603-7.
3.b. ARTÍCULO 2 ... 51
Rodríguez, I., M. C. Martín, M. C. Mendoza y M. R. Rodicio. 2006. Class 1 and class 2 integrons in non-prevalent serovars of Salmonella enterica: structure and association with transposons and plasmids. J. Antimicrob. Chemother. 58: 1124-32.
3.c. ARTÍCULO 3 ... 61
Rodríguez, I., M. R. Rodicio, S. Herrera-León, A. Echeita y M. C. Mendoza. 2008. Class 1 integrons in multidrug-resistant non-typhoidal Salmonella enterica isolated in Spain between 2002 and 2004. Int. J. Antimicrob. Agents 32: 158-64.
3.d. ARTÍCULO 4 ... 69
Rodríguez, I., W. Barownick, R. Helmuth, M. C. Mendoza, M. R. Rodicio, A. Schroeter y B. Guerra. ESBLs and AmpC beta-lactamases in ceftiofur resistant Salmonella enterica isolates from food and livestock obtained in Germany during 2003-2007 (en preparación).
3.e. ARTÍCULO 5 ... 79
Rodríguez, I., M. C. Mendoza y M. R. Rodicio. pUO-SeVR1 is an emergent and large virulence-resistance plasmid of Salmonella enterica serovar Enteritidis (en preparación).
3.f. ARTÍCULO 6 ... 85
Martínez, N., M. C. Mendoza, I. Rodríguez, S. Soto, M. Bances y M. R. Rodicio. 2007. Detailed structure of integrons and transposons carried by large conjugative plasmids responsible for multidrug resistance in diverse genomic types of Salmonella enterica serovar Brandenburg. J. Antimicrob. Chemother. 60: 1227-34.
3.g. ARTÍCULO 7 ... 93
Martínez, N., I. Rodríguez, R. Rodicio, M. C. Mendoza, y M. R. Rodicio. Molecular basis, evolution and trends of multiple drug resistance in clinical isolates of Salmonella enterica serovarOhio (en preparación).
3.h. ARTÍCULO 8 ... 103
Soto, S. M., I. Rodríguez, M. R. Rodicio, J. Vila y M. C. Mendoza. 2006. Detection of virulence determinants in clinical strains of Salmonella enterica serovar Enteritidis and mapping on macrorestriction profiles. J. Med. Microbiol. 55: 365-73.
4. DISCUSIÓN
...4.a. INTEGRONES DE CLASE 1 Y 2 EN SEROTIPOS NO TIFOIDEOS DE S. enterica ... 113
4.a.1. EN CEPAS AISLADAS EN EL PRINCIPADO DE ASTURIAS ... 113
4.b. BLEEs Y ENZIMAS AMP-C EN CEPAS AISLADAS DE ALIMENTOS Y ANIMALES... 124
4.c. PLÁSMIDO HÍBRIDO DE VIRULENCIA-RESISTENCIA EN S. Enteritidis ... 128
4.d. CONSIDERACIONES GENERALES ... 133
5. CONCLUSIONES
... Español ... 135 English ... 137FIGURAS
...
Figura 1
.
Puntos diana de los antimicrobianos y mecanismos de resistencia ... 9Figura 2
.
Componentes de una casete génica ... 12Figura 3
.
Inserción de casetes en un integrón ... 13Figura 4
.
Esquema del integrón In2 de clase 1 ... 14Figura 5
.
Esquema de un integrón de clase 2 ... 15Figura 6
.
Esquema de la secuencia de inserción IS1 ... 18Figura 7
.
Ejemplo de una secuencia de inserción ISEcp1 y del integrón complejo In7 ... 20Figura 8
.
Representación esquemática del transposón compuesto Tn9 ... 21Figura 9
.
Transposón Tn21 portador del integrón de clase 1 In2 ... 22Figura 10
.
Transposón Tn7 portador de un integrón de clase 2 ... 23Figura 11
.
Organización genética de la isla SGI1 ... 25Figura 12
.
Características del género Salmonella ... 27Figura 13
.
Pared celular de una bacteria Gram negativa ... 28Figura 14
.
Variación de fase de los flagelos en Salmonella ... 29Figura 15
.
Factores de virulencia representativos de S. enterica ... 33Figura 16
.
Características generales de una isla de patogenicidad ... 36Figura 17
.
Mapa genético de los plásmidos pSTV, pSEV, pSCV y pSDV ... 41Figura 18
.
Evolución de la subfamilia Tn21 ... 116Figura 19
.
Diferentes estructuras derivadas de Tn2411 encontradas en nuestras cepas ... 117Figura 20
.
Esquema del plásmido conjugativo pUO-SwR1 ... 119attC:sitio secundario de recombinación localizado en una casete génica BLEE:β-Lactamasa de Espectro Extendido
CR: Región Común
determinante-R/gen-R: Gen de Resistencia
determinante-V/gen-V: Gen de Virulencia
DR: Repeticiones Directas
Fenotipo/genotipo-R: Fenotipo/Genotipo de Resistencia
IR: Repeticiones Terminales Invertidas
IS: Secuencia de Inserción
LPS: Lipopolisacárido
orf:Pauta abierta de lectura
pSAV: Plásmido de Virulencia de Salmonella Abortus-ovis
pSCV: Plásmido de Virulencia de Salmonella Choleraesuis
pSDV: Plásmido de Virulencia de Salmonella Dublin
pSEV o pENT: Plásmido de Virulencia de Salmonella Enteritidis
pSGV: Plásmido de Virulencia de Salmonella Gallinarum
pSPV: Plásmido de Virulencia de Salmonella Pullorum
pSTV o pSLT: Plásmido de Virulencia de Salmonella Typhimurium
pUO-SeVR: Plásmido Universidad de Oviedo Salmonella Enteritidis Virulencia-Resistencia
pUO-SpR: Plásmido Universidad de Oviedo Salmonella Panama Resistencia
pUO-StVR: Plásmido Universidad de Oviedo Salmonella Typhimurium Virulencia-Resistencia
pUO-SwR: Plásmido Universidad de Oviedo Salmonella Worthington Resistencia
SGI1: Isla Genómica 1 de Salmonella
SPI: Islas de Patogenicidad de Salmonella
SST1: Sistema de Secreción de Tipo 1
SST3: Sistema de Secreción de Tipo 3
SST4: Sistema de Secreción de Tipo 4
La emergencia y diseminación de resistencias a antimicrobianos supone un problema para la salud pública y la sanidad animal puesto que pone en serio peligro la eficacia del principal tratamiento contra las infecciones bacterianas. Por este motivo son especialmente interesantes los estudios centrados no sólo en la identificación de los determinantes de resistencia, sino también de los mecanismos que intervienen en su dispersión. En este contexto, la presente Tesis Doctoral profundiza en la incidencia y caracterización molecular de elementos genéticos implicados en la captura, diseminación y mantenimiento de genes de resistencia a antimicrobianos por cepas de serotipos no tifoideos de Salmonella enterica, un patógeno bacteriano que, por su carácter zoonótico, podría estar ocupando un lugar relevante como donador y receptor de genes de resistencia. Los puntos desarrollados en este trabajo fueron fundamentalmente tres:
1.- Se demostró el importante papel que juegan los integrones de clases 1 y 2, constatando su presencia en un gran número de cepas multirresistentes (mayoritariamente clínicas), aisladas en el Principado de Asturias y el resto de España. Dos hechos destacables fueron: a) la nueva descripción de los integrones de clase 1 con las regiones variables: 2300 pb/estX-smr-aadA1 y 2100 pb/dfrA1-597pb-aadA24; y b) la primera identificación de integrones de clase 2 en los serotipos S. Virchow, S. Grumpensis, S. Panama y S. Worthington. Se confirmó la asociación de estas unidades genéticas con elementos transponibles, observando diferentes configuraciones integrón de clase 1-transposón tipo Tn21 o integrón de clase 2-transposón tipo Tn7. Además, en la mayoría de los casos estas estructuras se encontraban localizadas en plásmidos conjugativos de gran tamaño, a veces portadores de resistencias adicionales no asociadas a integrones.
2.- Dada la creciente emergencia de las resistencias a cefalosporinas de tercera y cuarta generación, se rastrearon β-lactamasas de espectro extendido (BLEEs) y enzimas plasmídicas tipo AmpC en cepas alemanas procedentes de alimentos y animales. Subrayar que la familia de cefotaximasas CTX-M-1 era la más frecuente, y se encontraba asociada a secuencias de inserción tipo ISEcp1. Tanto los genes de BLEEs como de enzimas AmpC, se localizaban en plásmidos de tamaño variable, la mayoría conjugativos.
3.- Otro hecho alarmante, dentro del panorama de la multirresistencia, es la aparición de plásmidos híbridos de virulencia-resistencia. En esta Tesis Doctoral se identificó y caracterizó parcialmente un plásmido, denominado pUO-SeVR1, que se encontró en un aislamiento de S. Enteritidis, el serotipo con mayor incidencia en infecciones humanas. Los resultados obtenidos sugieren que deriva de pSEV (el plásmido de virulencia específico de S. Enteritidis) por haber adquirido genes de resistencia asociados a un integrón y a diversos elementos transponibles. A la vez se desvelan algunos de los mecanismos que controlan la transmisión estable del plásmido dentro de la población bacteriana.
The emergence and dissemination of antimicrobial resistance is a worrisome problem for human and veterinary medicines, threatening the effectiveness of the main treatment against the bacterial infections. For this reason, studies focused not only on the identification of the resistance determinants but also on the mechanisms involved in their spread, are especially interesting. In this context, this Doctoral Thesis analyzed the incidence and molecular characterization of mobile genetic elements related to the capture, maintenance and dissemination of antimicrobial resistance genes. For this purpose, multidrug resistant strains belonging to different non-typhoidal serovars of Salmonella enterica were studied. This bacterial pathogen, because of its zoonotic quality, might perform a relevant function as donor and recipient of resistance genes. The main objectives developed in this work were the following:
1.- The important role played by class 1 and class 2 integrons was demonstrated by ascertaining their presence in many multidrug resistant strains (mostly from clinical origin), isolated in the Principality of Asturias and the rest of Spain. Two remarkable facts were: a) the new description of class 1 integrons with the variable regions: 2300 bp/estX-smr-aadA1 and 2100 bp/dfrA1
-597bp-aadA24; and b) the first identification of class 2 integrons in serovars S. Virchow, S. Grumpensis, S. Panama and S. Worthington. The linkage between these genetic units and transposable elements was also established, finding different class 1 integron-Tn21-like transposon or class 2 integron-Tn7 -like transposon configurations. Moreover, in most cases these structures were located on large conjugative plasmids, some of them carrying additional non integron-associated resistances.
2.- Due to the increasing emergence of the resistance to the third- and fourth-generation cephalosporins, the presence of extended spectrum β-lactamases (ESBLs) and plasmidic AmpC enzymes was analyzed in German isolates of animal and food origin. The CTX-M-1 family was the prevalent, and was associated with insertion sequences ISEcp1-like. The ESBL and AmpC encoding genes were in all cases located on plasmids of variable sizes, mainly self-transferable.
3.- Another alarming trend in the multidrug resistance background, is the emergence of virulence-resistance hybrid plasmids. In this Doctoral Thesis, the identification and partial characterization of the plasmid pUO-SeVR1 (found in a S. Enteritidis isolate) was carried out. The results obtained suggest that it derived from pSEV (the virulence plasmid specific of S. Enteritidis), through acquisition of resistance genes associated with a class 1 integron and diverse transposable elements. Some mechanisms involved in the stable inheritance of the plasmid were also identified.
1.a. RESISTENCIA A ANTIMICROBIANOS
Las bacterias patógenas y el hombre han mantenido una lucha constante a lo largo
de la historia. Cuando en la primera mitad del siglo XX se desarrollaron los primeros agentes
antimicrobianos y se aplicaron en clínica, el hombre creía haber ganado la batalla frente a
los patógenos bacterianos. Sin embargo, al poco tiempo las bacterias demostraron una vez
más el porqué de su éxito evolutivo: su enorme plasticidad para adaptarse a nuevas
presiones selectivas. De esta forma pocos años después de la introducción de los antibióticos
en clínica, aparecían las primeras bacterias resistentes. Con el desarrollo posterior de nuevos
compuestos antimicrobianos, el hombre sólo ha conseguido poner obstáculos en la carrera
armamentística de las bacterias, que una vez más han sabido responder a los nuevos
agentes selectivos aumentando el nivel y la complejidad de los mecanismos de resistencia.
Además, el uso abusivo de antimicrobianos en terapia humana, y la utilización más o
menos indiscriminada en otros campos como ganadería, acuicultura y agricultura no ha
hecho más que favorecer la selección de bacterias resistentes, y hoy en día esta situación se
ha convertido en un reto para la salud pública y la sanidad animal, en tanto que
compromete considerablemente el que sigue siendo principal mecanismo de control de
estos agentes patógenos.
Por los motivos apuntados, son de gran interés los estudios acerca de las estrategias
de resistencia (determinantes-R), puesto que su conocimiento permitirá desarrollar futuras
actuaciones más eficaces que las actuales en nuestra lucha contra las enfermedades
infecciosas.
1.b. BASES BIOQUÍMICAS DE LA RESISTENCIA
EN BACTERIAS GRAM NEGATIVAS
Los antimicrobianos dañan a las bacterias interfiriendo en diferentes procesos celulares
esenciales, lo que puede conllevar a la muerte del microorganismo (si se trata de un
antimicrobiano bactericida) o bien al cese reversible de su crecimiento (antimicrobiano
bacteriostático). Sin embargo, las cepas que son resistentes a alguno de estos compuestos,
consiguen mantener su crecimiento incluso en presencia de los mismos o de otros, más o
menos afines, en casos de resistencia cruzada.Por eso, a la hora de abordar el tema de las
resistencias es preciso conocer, en primer lugar, las principales estrategias bioquímicas de
que disponen las bacterias para eludir la acción de los agentes utilizados contra ellas. En
nuestro caso nos centraremos en aquellas principalmente desarrolladas por bacterias Gram
negativas (Figura 1).
1.b.1. ENZIMAS INACTIVADORAS Y MODIFICADORAS
Son enzimas específicas que hidrolizan o modifican el antibacteriano inactivándolo
antes o después de penetrar en la bacteria (Wright, 2005). Dentro de este grupo
encontramos diversos tipos de enzimas, que varían también según la familia de
antimicrobianos sobre la que actúan. Algunos ejemplos son:
Enzimas inactivadoras de β-lactámicos: β-lactamasas
Los β-lactámicos son una gran familia de antibióticos que se caracterizan por presentar
un anillo β-lactámico en su estructura química. Este componente va unido generalmente a
un anillo secundario, cuya composición va a determinar los diferentes grupos de compuestos
que incluye esta familia: penicilinas, cefalosporinas, carbapenemes y monobactámicos
(Mirelis y Gurguí, 2006).
• Las penicilinas están formadas por la fusión de un anillo β-lactámico con uno tiazólico.
Dentro de ellas distinguimos penicilinas naturales (como la penicilina G) y
(ampicilina), las carboxipenicilinas (ticarcilina) y las acilureidopenicilinas (piperacilina),
ambas activas frente a Pseudomonas aeruginosa.
• Las cefalosporinas poseen el grupo cefem, formado por la fusión del anillo β-lactámico
con otro dihidrotiacina. Se clasifican por generaciones de compuestos, que van en un
orden creciente de espectro de acción. Así de espectro más reducido son las de
primera (por ejemplo, la cefalotina) y segunda generación (como el cefuroxime y la
cefoxitina). Los de la tercera generación son de amplio espectro, como cefotaxime,
ceftriaxone, ceftacidime y cefpodoxime. Por último, la cuarta generación es más
estable frente a β-lactamasas, como es el caso de cefepime.
• Los carbapenemes, entre ellos el imipenem, están formados por un anillo β-lactámico
unido a un anillo insaturado de 5 componentes, y son activos frente a un gran número
de patógenos.
• Por último, los monobactámicos son compuestos monocíclicos y entre ellos se
encuentra el aztreonam.
Los β-lactámicos son agentes bactericidas e interfieren en la síntesis de la pared
bacteriana, bloqueando la formación de su principal componente estructural: el
peptidoglicano. (Mirelis y Gurguí, 2006). Entre todos los mecanismos de resistencia que se han
descrito frente a esta familia de compuestos, la producción de β-lactamasas se considera el
principal entre bacterias Gram negativas. Estas enzimas hidrolizan el anillo β-lactámico
originando derivados ácidos que son biológicamente inactivos. En la actualidad se conocen
más de 700 β-lactamasas diferentes, y se pueden clasificar según su estructura molecular (en
cuatro clases de la A a la D; Ambler et al., 1991) o en base a características estructurales y
funcionales (en grupos del 1 al 4; Bush et al., 1995). La relevancia clínica que puede tener un
tipo u otro de β-lactamasa dependerá del rango de sustratos que puede hidrolizar, así como
su susceptibilidad frente a inhibidores de β-lactamasas, entre ellos el ácido clavulánico. De
esta manera, y siguiendo el esquema de Bush et al. (1995):
• El grupo 1 incluye las cefalosporinasas de tipo AmpC, como aquéllas codificadas por
genes blaCMY, y que son capaces de resistir la acción del ácido clavulánico y median
resistencia a casi todos los β-lactámicos, exceptuando carbapenemes y cefalosporinas
de cuarta generación.
• El grupo 2 engloba enzimas que pueden hidrolizar penicilinas, cefalosporinas o
carbapenemes, la mayoría sensibles a la acción del ácido clavulánico. Por ejemplo,
penicilinasas de espectro reducido (codificadas por genes como blaTEM-1, blaSHV-1,
blaOXA-1) y β-lactamasas de espectro extendido (BLEEs), que pueden hidrolizar
• El grupo 3 comprende las metalo-β-lactamasas, que poseen cuatro átomos de zinc en
su sitio activo y son capaces de hidrolizar carbapenemes y cefalosporinas, como
aquéllas codificadas por genes del tipo blaIMP.
• Por último, el grupo 4 es muy heterogéneo y contiene penicilinasas que son muy
resistentes a la acción del ácido clavulánico. Sin embargo ninguno de los genes que
las codifican ha sido secuenciado aún, por lo que su pertenencia a este grupo es
provisional, hasta tener más datos que permitan clasificarlas de forma adecuada.
Enzimas modificadoras de aminoglicósidos
Los aminoglicósidos son agentes bactericidas que inhiben la síntesis proteica al unirse a
la subunidad 30S del ribosoma bacteriano (Mirelis y Gurguí, 2006). Estos antimicrobianos se
forman por la combinación de azúcares no aminados (glicósidos) o aminados
(aminoglicósidos) con alcoholes cíclicos no aminados (ciclitoles) o aminados
(aminociclitoles). De esta manera podemos distinguir entre aminoglicósidos aminociclitoles
(estreptomicina, gentamicina o kanamicina), glicósidos aminociclitoles (espectinomicina) y
aminoglicósidos ciclitoles (kasugamicina) (Mella et al., 2004). Los antibacterianos de
importancia en clínica pertenecen al primer grupo y se puede subdividir en:
• Estreptidínicos: contienen un anillo estreptidina, como es el caso de la estreptomicina y
derivados.
• Deoxiestreptamínicos: poseen un anillo 2-deoxiestreptamina, y a su vez se agrupan en
función de las posiciones del anillo ciclitol donde se establecen las uniones con los
glicósidos:
- hidroxilos 4,5: neomicina, paramomicina, ribostamicina, butirosina, lividomicina.
- hidroxilos 4 y 6: kanamicina, gentamicina, tobramicina, amikacina.
- hidroxilos 4 y 2: apramicina.
- hidroxilos 5 y 2: higromicina.
Las enzimas modificadoras de aminoglicósidos aminociclitoles son transferasas que
catalizan la modificación de grupos amino o hidroxilo de la molécula del antibiótico, de
forma que el nuevo compuesto no puede unirse a las moléculas ribosómicas diana. Las
transferasas constituyen el mecanismo más frecuente de resistencia frente a estos
compuestos, y se pueden distribuir en diferentes clases según el tipo de grupo químico que
transfieren al aminoglicósido. Así, las O-nucleotidiltransferasas (ANT) catalizan una reacción
de adenilación de un grupo hidroxilo del antibiótico, y según la posición del anillo sobre la
ANT(3”)), que confieren resistencia a estreptomicina y espectinomicina; o aadB (ANT(2”)) que
modifican gentamicina, kanamicina y tobramicina entre otros (Schmidt et al., 1988). Las
enzimas modificadoras capaces de transferir grupos acetilo a un grupo amino del
antimicrobiano se llaman N-acetiltransferasas (AAC) y los grupos de genes aacA (AAC(6’)),
aacB (AAC(2’)) y aacC (AAC(3)) codifican enzimas con diferente perfil de sustratos.
Destacan los genes aacC1, aacC2 o aacC4, que median resistencia a gentamicina, y el gen
aacA1 descrito por nuestro grupo que codifica una enzima de amplio rango de sustrato,
incluyendo los semisintéticos tobramicina y amikacina (Terán et al., 1991). Una mención
especial merece el reciente hallazgo de la variante cr de la enzima AAC(6’)-Ib, una
aminoglicósido acetil transferasa que, gracias a dos cambios aminoacídicos (Trp102 a Arg, y
Asp179 a Tyr), es capaz también de inactivar por acetilación a antimicrobianos de la familia
de las quinolonas (Robicsek et al., 2006; Park et al., 2006). Por último están las
O-fosfotranferasas (APH), codificadas por genes aph, que fosforilan grupos hidroxilo de
diferentes anillos, y así podemos distinguir entre genes aphA (APH(3’)) como aphA1,
responsable de la resistencia a kanamicina; aphB (APH(5”)); y aphC (APH(3”)), grupo al que
pertenecen los genes strA-strB, que codifican fosfotransferasas modificadoras de la
estreptomicina (Novick et al., 1976; Vanhoof et al., 1998). Las características de estas
transferasas y las diferencias estructurales existentes entre aminoglicósidos estreptidínicos y
deoxiestreptamínicos evitan que se den fenómenos de resistencia cruzada entre ambos tipos
de compuestos.
Enzimas modificadoras de fenicoles
A este grupo de agentes bacteriostáticos pertenece el cloranfenicol, un agente de
amplio espectro de acción que inhibe la síntesis proteica uniéndose a la subunidad 50S del
ribosoma, bloqueando la transpeptidación (Mirelis y Gurguí, 2006). Este tipo de
antimicrobianos es principalmente inactivado a través de la acción de acetiltransferasas,
como es el caso de las cloranfenicol acetiltransferasas (Cat, codificadas por genes cat) que
acetilan los dos radicales hidroxilo del antibacteriano y que, según su secuencia
aminoacídica y estructura tridimensional, pueden ser de tipo A o B.
1.b.2. EXPULSIÓN ACTIVA DEL ANTIBIÓTICO
Este mecanismo de resistencia se basa en la síntesis de proteínas que actúan como
bombas de expulsión activas específicas para diferentes antibióticos, y que evitan la
Proteínas de expulsión de fenicoles
Constituyen un mecanismo alternativo de resistencia a fenicoles mediado por proteínas
transportadoras específicas con diferente espectro antibiótico. Como ejemplos podemos
señalar las proteínas CmlA (codificada por genes cmlA) que se encargan de la expulsión del
cloranfenicol de la célula; o la bomba codificada por el gen floR, que es capaz de exportar
tanto el cloranfenicol como el florfenicol (George y Hall, 2002; Michael et al., 2006).
Bombas de expulsión de tetraciclinas
Las tetraciclinas son antimicrobianos bacteriostáticos que comparten una estructura
de cuatro anillos a los que están ancladas diversas cadenas laterales. Inhiben la síntesis
proteica uniéndose a la subunidad 30S del ribosoma, y la tetraciclina, doxiciclina y
minociclina son algunos ejemplos de este grupo (Mirelis y Gurguí, 2006). El mecanismo de
resistencia mejor estudiado y más frecuente entre bacterias Gram negativas es el mediado
por bombas de expulsión, que transportan activamente a las tetraciclinas fuera de la célula
impidiendo el daño.Hasta la fecha hay al menos 19 genes descritos que codifican este tipo
de proteínas, las cuales a su vez se clasifican en 6 grupos en función de la identidad de su
secuencia aminoacídica. De entre ellos destaca el grupo 1, que engloba la mayoría de
bombas de flujo encontradas en Gram negativos, como las codificadas por los genes tet(A),
tet(B) y tet(G). Estos polipéptidos presentan 12 dominios transmembrana y la mayoría de ellos
sólo son capaces de exportar tetraciclina desde el citoplasma al exterior de la bacteria,
aunque el producto de tet(B) confiere además resistencia a minociclina (Chopra y Roberts,
2001).
1.b.3. MODIFICACIÓN DE LA DIANA CELULAR
Con esta estrategia la célula evita el daño causado por el antimicrobiano sintetizando
una molécula diana alternativa o modificándola de modo que se haga resistente.
Resistencia a sulfonamidas y trimetoprim
Estos dos antimicrobianos ejercen su acción bloqueando la formación de purinas y
pirimidinas (elementos esenciales en la construcción de los ácidos nucleicos) interfiriendo en
la síntesis de su precursor: el ácido fólico. Por un lado, las sulfonamidas compiten con el ácido
para-aminobenzoico por el sitio activo de la enzima dihidropteroato-sintetasa, lo que impide
trimetoprim actúa de manera similar pero en un paso posterior de la ruta, inhibiendo a la
dihidrofolato-reductasa, enzima que cataliza el paso de ácido dihidrofólico a ácido
tetrahidrofólico (Mirelis y Gurguí, 2006). El resultado de la acción de cualquiera de estos
antimicrobianos es el bloqueo de la síntesis de los ácidos nucleicos.
Los mecanismos de resistencia desarrollados contra estos agentes antibacterianos
implican la formación de enzimas diana alternativas. Así, en el caso de las sulfonamidas, los
genes responsables de su resistencia codifican enzimas dihidropteroato-sintetasas a las que
no se une el antimicrobiano. Se conocen tres genes distintos implicados en este mecanismo
de resistencia: sul1, sul2 y sul3, siendo los dos primeros los más frecuentemente encontrados
en aislamientos clínicos Gram negativos (Sköld, 2001; Michael et al., 2006). La resistencia al
trimetoprim está mediada por genes que codifican una enzima dihidrofolato-reductasa
alternativa a la de la bacteria y que es resistente a la acción del agente. Actualmente hay
más de 30 genes diferentes identificados, que se dividen en dos tipos: dfrA y dfrB, en función
de su estructura (Sköld, 2001; Michael et al., 2006).
Resistencia a rifamicinas
Las rifamicinas son compuestos macrocíclicos bactericidas que actúan inhibiendo la
transcripción a nivel de la ARN-polimerasa ADN-dependiente. El principal representante de
este grupo es la rifampicina, un derivado semisintético de la rifamicina B, que es activo frente
a micobacterias, así como otras Gram positivas y frente a Gram negativas.
El mecanismo de resistencia más frecuente que se observa en los aislamientos clínicos
consiste en la alteración de la subunidad β de la ARN polimerasa, que se produce a través
de mutaciones puntuales en el gen que la codifica (rpoB). Las sustituciones en un limitado
número de aminoácidos altamente conservados de dicha subunidad son suficientes para
generar altos niveles de resistencia a rifampicina (Telenti et al., 1993; Mariam et al., 2004;
Mirelis y Gurguí, 2006).
Resistencia a quinolonas y fluoroquinolonas: modificación de la diana celular
Las quinolonas son agentes bacteriostáticos sintéticos que interrumpen la replicación
del ADN mediante la inhibición de las topoisomerasas de tipo II (ADN girasa) y de tipo IV.
Estas enzimas son estructuras tetraméricas compuestas por dos copias de dos subunidades,
que en el caso de la ADN girasa se denominan GyrA y GyrB, y en la topoisomerasa IV, ParC y
Dentro de las quinolonas, el primer componente sintetizado fue el ácido nalidíxico,
cuya estructura es 1,8-naftiridina. Posteriormente se sintetizaron las fluoroquinolonas mediante
la adición de un átomo de flúor a su estructura, lo que aumentó su actividad y espectro de
acción. Entre ellas destacamos: ciprofloxacina, norfloxacina, levofloxacina, moxifloxacina y
trovafloxacina (Andriole, 2005; Mirelis y Gurguí, 2006). El mecanismo de resistencia a estos
compuestos más importante a nivel clínico es el que se basa en la modificación de las
proteínas diana, mediante la aparición de mutaciones puntuales en los genes que las
codifican. Generalmente, en las bacterias Gram negativas la diana principal de las
quinolonas es la ADN girasa, y las mutaciones que confieren resistencia suelen aparecer en el
gen gyrA. Así, una única sustitución en este gen (por ejemplo del aminoácido Ser83 a Phe, Tyr
o Ala; o Asp87 a Gly, Asn o Tyr), puede estar asociada a altos niveles de resistencia a
quinolonas, mientras que es necesaria la acumulación de más mutaciones en gyrA o en
parC para que se desarrolle una resistencia completa a fluoroquinolonas (Vila et al., 2000).
Resistencia a quinolonas y fluoroquinolonas: protección de la diana celular
En 1998 se descubrió un nuevo mecanismo de resistencia a quinolonas en un
aislamiento clínico de Klebsiella pneumoniae que, a diferencia de los anteriormente
descritos, estaba mediado por un plásmido (Martínez-Martínez et al., 1998). El gen
responsable se denominó qnr, y era capaz de incrementar la resistencia al ácido nalidíxico,
ciprofloxacina y otras fluoroquinolonas en las cepas que lo portaban (Tran y Jacoby, 2002).
La proteína Qnr pertenece a la familia de los pentapéptidos repetidos, y su modo de acción
consiste en la unión a la ADN girasa y a la topoisomerasa IV para protegerlas de la inhibición
(Jacoby, 2005).
Resistencia a aminoglicósidos
La modificación por metilación de la diana celular es un mecanismo de resistencia a
aminoglicósidos que se ha detectado recientemente en patógenos humanos Gram
negativos (Galimand et al., 2003). El gen responsable es armA, que codifica una
metiltransferasa capaz de modificar nucleótidos altamente conservadas del ARNr 16S. De
esta manera se impide la unión entre la molécula de aminoglicósido y el ribosoma,
confiriendo altos niveles de resistencia a toda esta familia de antimicrobianos, a excepción
de la estreptomicina. Antes del descubrimiento de armA en un aislamiento clínico de K.
pneumoniae, este sistema de metilación se consideraba exclusivo de los microorganismos
Figura 1.
Puntos diana de los antimicrobianos (1-6) y mecanismos de resistencia (a-e).
-
Entre los puntos diana: (1) síntesis de la pared bacteriana; (2) desorganización de la
membrana citoplasmática; (3) metabolismo, síntesis de precursores de ácidos nucleicos; (4)
replicación del ADN; (5) transcripción; (6) traducción.
-
Mecanismos de resistencia: (a) enzimas inactivadoras y modificadoras del antibiótico; (b)
superproducción de la diana; (c) modificación de la diana celular; (d) expulsión activa del
antibiótico; (e) alteración de la permeabilidad por cambios en las porinas.
1.c.
BASES GENÉTICAS DE LA RESISTENCIA
Detrás de estas estrategias bioquímicas hay un soporte genético que contiene las
claves, no sólo de las funciones de resistencia en sí mismas, sino también de la complejidad
estructural de la multirresistencia y de la amplia dispersión que este fenómeno tiene en la
actualidad. En este contexto debemos comenzar por diferenciar entre resistencia innata y
resistencia adquirida.
Así, por un lado, existen mecanismos intrínsecos (resistencia innata) por los que
determinados microorganismos son resistentes a una o más familias de antimicrobianos. En
2
6
5
1
3
4
a
b
c
d
este caso el fenotipo se debe a una resistencia intrínseca al mecanismo de acción del
antibacteriano o a la impermeabilidad de la célula frente al mismo. Por ejemplo, las
bacterias anaerobias carecen del sistema de transporte oxígeno-dependiente necesario
para que los aminoglicósidos penetren en la célula bacteriana, por lo que son resistentes de
forma innata a estos compuestos. Sin embargo, es la resistencia adquirida la que juega un
papel más relevante, y se consigue mediante dos vías: la mutación y la transferencia génica
horizontal (Cantón et al., 2003; Livermore, 2003; Tenover, 2006; Alekshun y Levy, 2007).
1.d. MUTACIÓN
La mutación puede modificar genes bacterianos o sus reguladores dando lugar a
genes de resistencia (genes-R) y, como consecuencia, originar organismos resistentes
(Cantón et al., 2003; Livermore, 2003). En estas condiciones, la presencia de antibacterianos
en el medio actuando como agentes selectivos, conforma el siguiente paso necesario para
el mantenimiento de las variantes resistentes ya formadas. En la mayoría de los casos, las
mutaciones afectan sólo a una familia de antimicrobianos, pero pueden darse efectos
pleiotrópicos si la permeabilidad o los sistemas de flujo se ven dañados, lo que conlleva la
resistencia a múltiples tipos o familias. Por otro lado, por acumulación de diferentes
mutaciones se puede llegar a conseguir un fenotipo multirresistente. En este caso, el
fenómeno se ve acentuado en cepas en las que los sistemas de reparación del ADN
funcionan incorrectamente, favoreciéndose la acumulación de errores en el genoma
bacteriano.
En los primeros años de la quimioterapia se atribuía a la mutación cromosómica el
principal papel en la emergencia de resistencias. Sin embargo, desde hace décadas
sabemos que la aparición de cepas multirresistentes no puede imputarse única, ni
principalmente a los mecanismos de mutación (Rowe-Magnus y Mazel, 1999; Rowe-Magnus y
Mazel, 2002). En la actualidad se considera que es la adquisición de genes-R la solución más
frecuentemente adoptada por las bacterias para eludir la actividad antimicrobiana (Cantón
et al., 2003; Rowe-Magnus y Mazel, 2002).
1.e. TRANSFERENCIA GÉNICA HORIZONTAL
Un gen mutado no sólo persiste en una población bacteriana mediante transferencia
vertical, sino que también puede diseminarse a otras bacterias con las que esté
génica horizontal. Este mecanismo supone una ruta altamente eficaz en la dispersión de
resistencias y para su funcionamiento se sirve de estructuras genéticas autotransmisibles o
movilizables, como plásmidos, bacteriófagos, transposones e integrones (Cantón et al., 2003).
Además de los elementos que controlan la transferencia física del ADN (a través de
transformación, conjugación o transducción), en la transferencia génica horizontal hay que
tener en cuenta también la capacidad de las células para replicar y expresar el ADN
exógeno. Pero a pesar de estas limitaciones hoy en día se sabe que la transferencia génica
horizontal tiene una prevalencia mucho mayor de lo que se pensaba y se han descrito
eventos de este tipo entre organismos evolutivamente distantes (Courvalin, 1994; Alekshun y
Levy, 2007). Así, la disposición de los genes-R en diferentes elementos genéticos móviles y la
inclusión de éstos en otros de mayor complejidad facilitan y aseguran, en gran medida, la
rápida dispersión de los determinantes-R entre las poblaciones bacterianas.
1.e.1. CASETES GÉNICAS
Las casetes génicas son unidades genéticas móviles muy sencillas que se componen
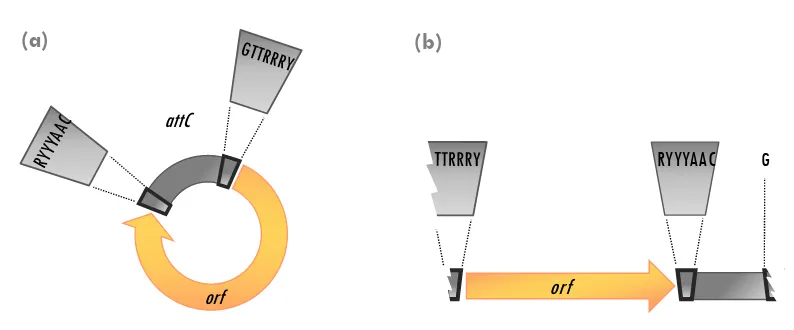
únicamente de una pauta abierta de lectura (‘open reading frame’, orf) y un sitio
secundario de recombinación. La orf comprende la mayoría de la longitud de la casete,
suele carecer de promotor y codifica una función adaptativa. Hacia el extremo 3’ de la orf
se encuentra el sitio secundario de recombinación, denominado attC o elemento de 59
bases (59-be), cuya secuencia y longitud varía considerablemente entre las diferentes
casetes génicas (Recchia y Hall, 1995).
Pese a la heterogeneidad de los sitios attC, en todos ellos se pueden identificar dos
regiones de unas 20 pb localizadas en los extremos, que son repeticiones imperfectas
invertidas. Dentro de éstas se encuentran dos secuencias de 7 pb cada una, altamente
conservadas, denominadas sitio ‘core’ inverso (cercano al extremo 3’ de la orf), con la
secuencia consenso RYYYAAC, y el sitio ‘core’ (próximo al extremo 5') que responde a la
secuencia GTTRRRY (donde Y= C+T, y R=A+G; Figura 2) (Recchia y Hall, 1995; Stokes et al.,
1997). La región central es de longitud y secuencia variable, mostrando normalmente una
estructura de repetición invertida.
Antes de su inserción, estas casetes existen en forma libre como moléculas circulares
covalentemente cerradas (Figura 2). En esta situación no son genes funcionales debido a la
ausencia de promotor, y para poder expresarse deben ser captados por sistemas de
Figura 2.
Componentes de una casete génica. Estructura de la casete génica y el sitio attC en
su forma circular covalentemente cerrada, indicando la posición del sitio attC (a) y en su forma
lineal después de la inserción (b).
1.e.2. INTEGRONES
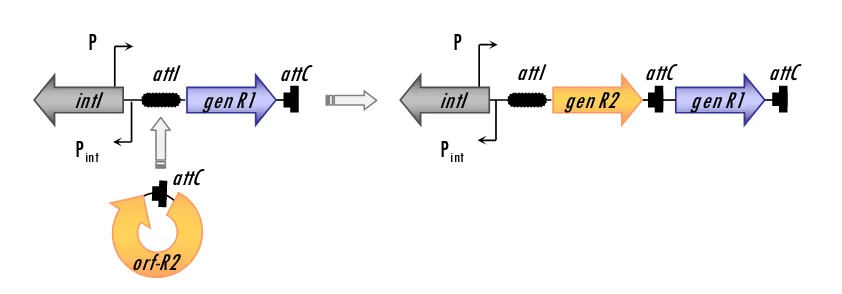
Los integrones son sistemas naturales de clonación y expresión capaces de incorporar
orfs mediante una recombinación específica de sitio, y convertirlas así en genes funcionales.
Los componentes básicos de un integrón son tres: el gen de la integrasa (intI) y el sitio
primario de recombinación (attI), responsables de la integración de casetes génicas; y el
promotor (o promotores), que permite(n) la expresión de las casetes contenidas en el
integrón (Stokes y Hall, 1989; Figura 3). Curiosamente, la estructura del sitio primario de
recombinación attI es muy diferente a la del sitio attC de las casetes, ya que sólo comparten
las 7 pb del sitio ‘core’ (GTTRRRY), careciendo attI del ‘core’ inverso y de las repeticiones
invertidas típicas de los attC (Stokes et al., 1997).
En el proceso de integración, el sitio attC es reconocido por la integrasa IntI, que es
capaz de catalizar una recombinación específica de sitio entre los elementos attC de la
casete génica y attI del integrón. En esta reacción el ‘core’ del attC participa activamente,
dado que la recombinación tiene lugar entre las bases G y T del triplete conservado GTT.
Para que se pueda producir la correcta expresión de la orf y su transformación en un gen
funcional, ésta tiene que insertarse en la orientación adecuada detrás del promotor(es) del
integrón. Es en este punto donde las diferencias en las secuencias de los dos extremos del
attC juegan un papel importante, asegurando la inserción de la casete génica en la
orientación correcta (Stokes et al., 1997; Figura 3).
orf
attC
RYYYAAC
Gorf
Además, la integrasa puede promover de forma consecutiva, la adquisición de más
de una casete génica (Figura 3), la mayoría de las cuales codifican resistencia a
antimicrobianos.
[image:35.595.97.518.143.290.2]
Figura 3.
Inserción de casetes en un integrón (Carattoli, 2001)
Las integrasas codificadas por los integrones pertenecen a una familia muy diversa de
tirosina-recombinasas y se caracterizan fundamentalmente por poseer cinco regiones
conservadas, dos de ellas de mayor tamaño y que contienen a su vez cuatro residuos
altamente conservados (Nunes-Düby et al., 1998). En base a la homología de las integrasas
(que oscila entre 41-57%) hay establecidas en la actualidad hasta nueve clases de
integrones diferentes (Nield et al., 2001; Hochhut et al., 2001). Algunos autores además, han
englobado los diferentes tipos de integrones en dos grandes grupos denominados integrones
de resistencia y superintegrones (Mazel et al., 1998; Rowe-Magnus y Mazel, 2002). Los
primeros se caracterizarían principalmente por contener casetes génicas que codifican en su
mayoría resistencia a antimicrobianos y los segundos por tener insertado un gran número de
casetes con funciones diversas. Sin embargo esta nomenclatura no está actualmente
aceptada, dado que la definición de integrón no incluye el contenido en casetes. De hecho,
éstas son consideradas como unidades móviles independientes, que sólo forman parte del
integrón al ser captadas por éste. Así, un integrón se define en base a sus tres componentes
conservados (el gen intI, el sitio attI y el promotor para la expresión de las casetes), pudiendo
englobar a todas las clases de integrones, tengan o no insertadas casetes génicas y sean
cuales sean las funciones que éstas codifiquen (Hall y Stokes, 2004; Hall et al., 2007; Nemergut
et al., 2008).
P
intattI
P
attC
gen R2
intI
attC
gen R1
P
intattI
P
attC
gen R1
intI
attC
Integrones de clase 1
Los integrones de clase 1 fueron los primeros en describirse, son los mejor estudiados y
los más ampliamente distribuidos entre los diferentes géneros bacterianos, jugando un papel
muy importante en la dispersión de resistencias, tanto en bacterias Gram negativas como
Gram positivas (Fluit y Schmitz, 2004). Se han encontrado asociados a docenas de casetes
génicas de resistencia y su frecuencia es especialmente relevante en bacterias patógenas
Gram negativas que infectan al hombre y los animales, incluyendo diferentes serotipos de
Salmonella enterica (Guerra et al., 2000a; Carattoli, 2001; Fluit y Schmitz, 2004). Este tipo de
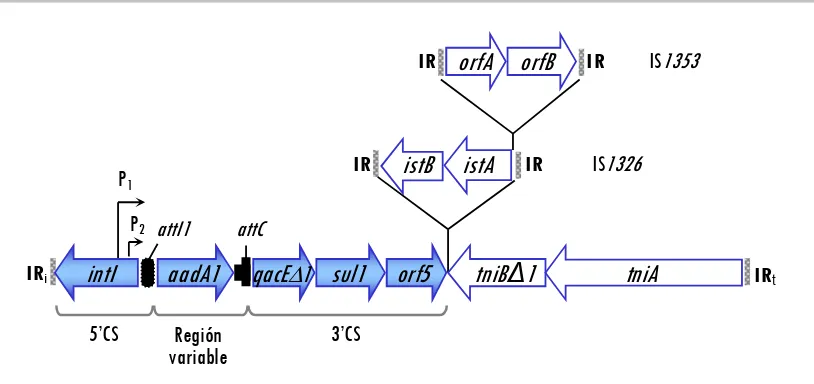
integrones se caracteriza por presentar dos segmentos conservados (5’CS y 3’CS) que
flanquean una región central variable donde pueden insertarse una o varias casetes
génicas. El segmento 5’ conservado se caracteriza por tener uno o dos promotores (para la
expresión de los genes insertados; P1 y P2, Figura 4), el gen de la integrasa intI1 y attI1. En el
extremo 3’ conservado se hallan generalmente genes que dotan a la bacteria de resistencia
a sulfonamidas (sul1) y a compuestos derivados del amonio cuaternario (qacE,
generalmente con la deleción ∆1), detrás de los cuales aparece orf5, de función
desconocida (Recchia y Hall, 1995; Carattoli, 2001).
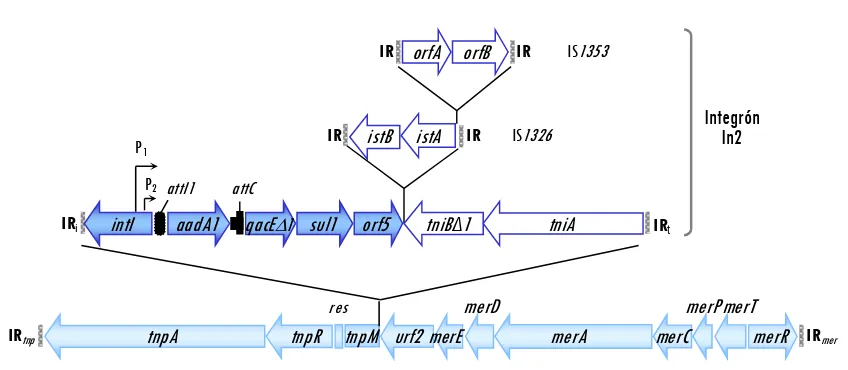
Un ejemplo de esta clase de integrones es In2, que contiene una única casete génica:
aadA1 (confiere resistencia a estreptomicina y espectinomicina), dos secuencias de inserción
denominadas IS1326 e IS1353 (ver apartado 1.e.3) y un módulo de transposición (tni)
incompleto (Figura 4). Como detallaremos más adelante (apartado 1.e.3), los integrones de
clase 1 se encuentran asociados frecuentemente a transposones de la familia de Tn21.
[image:36.595.88.495.476.668.2]
Figura 4.
Esquema del integrón In2 de clase 1 (Liebert et al., 1999).
istB
istA
orfA
orfB
IS
1353
IS
1326
IR
IR
IR
IR
tniB
Δ
1
tniA
IR
tqacE
Δ1
sul1
orf5
attC
IR
iaadA1
P
1P
2attI1
intI
5’CS
Región
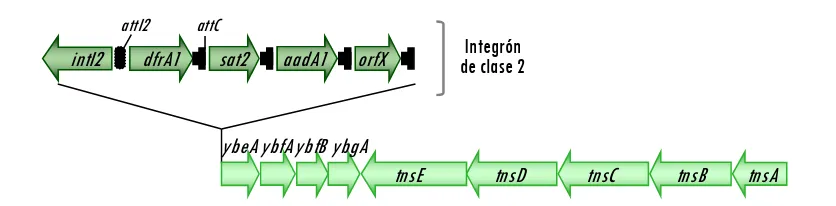
Integrones de clase 2
Los integrones de clase 2 son menos frecuentes, pero también se han descrito en
géneros de bacterias Gram negativas como Acinetobacter, Shigella (Fluit y Schmitz, 2004) y
Salmonella (Orman et al., 2002; Miko et al., 2003; Antunes et al., 2006; van Essen-Zandbergen
et al., 2007; Michael et al., 2008). Esta clase de integrones carecen del extremo 3’ descrito en
los de clase 1, y además, presentan la casete orfX que codifica una proteína de función
desconocida (Sundström y Sköld, 1990; Hansson et al., 2002). Sin embargo, comparten con los
de clase 1 la estructura del segmento 5’, ya que poseen un sitio attI2 y el gen intI2 que
codifica una integrasa no funcional por la presencia de un codón de parada (TAA) en el
interior de su secuencia nucleotídica (Figura 5). En el proceso de escisión de casetes génicas
en este tipo de integrones podría intervenir la integrasa IntI1, ya que es capaz de reconocer
casetes adyacentes a un sitio attI1 o attI2. Sin embargo existe cierta controversia respecto a
la participación de IntI1 en la recombinación integrativa. Algunos trabajos apuntan dicha
posibilidad (Hansson et al., 2002), mientras que otros autores califican de poco significativa la
frecuencia de reacciones entre attI2-attC mediadas por la integrasa de tipo 1(Collis et al.,
2002). Sea cual sea el caso de IntI2, se ha observado que el número de pseudogenes de
integrasas es mucho mayor al de otros genes, como revelan recientes análisis de genes intI
de diferentes genomas bacterianos (Nemergut et al., 2008). Entre todas las explicaciones
posibles, se apunta como más probable el impacto selectivo variable de los integrones en el
genoma bacteriano. Así, la versatilidad aportada por los integrones en la captación, escisión
y reorganización de genes exógenos podría resultar beneficiosa o perjudicial para la
bacteria, dependiendo de las condiciones medioambientales.
La configuración de casetes génicas más frecuentemente encontrada dentro de los
integrones de clase 2 es: dfrA1-sat2-aadA1, que codifican resistencia a trimetoprim,
estreptotricina y estreptomicina-espectinomicina, respectivamente (Figura 5). Como veremos
posteriormente (apartado 1.e.3) los integrones de clase 2 suelen estar asociados a
transposones de tipo Tn7.
Figura 5.
Esquema de un integrón de clase 2 basado en los datos de la secuencia del
plásmido R721de Escherichia coli (Kim y Komano, 1992; nº acceso NC_002525).
intI2
orfX
attC
attI2
aadA1
sat2
Integrones de clase 3, 4 y 5
Los integrones de clase 3 fueron identificados por primera vez en un plásmido
conjugativo de Serratia marcescens, conteniendo el gen de la integrasa intI3 (60,9% de
identidad con intI1), la casete blaIMP (gen de una beta-lactamasa que confiere resistencia a
β-lactámicos de amplio espectro) y parte del gen aacA4 (codifica una aminoglicósido acetil transferasa que confiere resistencia a aminoglicósidos del grupo de la gentamicina)
(Arakawa et al., 1995). Posteriormente se encontraron también integrones de esta clase con
una estructura similar en cepas de Ps. aeruginosa, Ps. putida, Alcaligenes xylosoxidans y K.
pneumoniae (Senda et al., 1996).
Se han identificado integrones de clase 4 en cepas de Vibrio cholerae, portando la
integrasa intI4 y estando normalmente asociados a casetes génicas que codifican para más
funciones aparte de la resistencia a antimicrobianos (Mazel et al., 1998; Shi et al., 2006). Esta
estructura fue encontrada en cepas del género Vibrio previas a la era de los antimicrobianos,
lo que sugiere que intI4 podría ser el ancestro de las demás integrasas, las cuales se pudieron
ir adaptando a la adquisición de genes-R en un pasado reciente. Esto apoya el hecho de
que los integrones jueguen un papel importante en la aceleración de la evolución
bacteriana (Mazel et al., 1998).
La quinta clase de integrón se caracteriza por la presencia de la integrasa IntI5, que
muestra una identidad del 75% con IntI4. Se ha detectado en una cepa de V. mimicus y su
secuencia se encuentra en la base de datos con el número de acceso: AF180939 (Clark et
al., 2000).
Otras clases de integrones
En un trabajo de detección de integrones en muestras ambientales de ADN, se
encontraron 3 nuevos genes codificadores de integrasas: intI6, intI7 e intI8. Las secuencias
aminoacídicas de estas integrasas mostraron identidades del 42 al 50% cuando se
compararon con las anteriormente descritas. Próximo al codón de inicio de estas integrasas
se detectó un promotor, se identificaron secuencias que sugieren la presencia de un sitio attI,
y en el caso de la 6 y la 7, también secuencias parciales de una orf insertada. Toda esta
información sugiere que las tres integrasas forman parte de integrones completos, que se
pasaron a denominar integrones de clases 6, 7 y 8 (Nield et al., 2001).
La integrasa IntI9 fue encontrada en una cepa de V. cholerae, muestra un 53% de
trimetoprim), seguida de otras cuatro casetes más, una de ellas conteniendo dos orfs en
orientaciones opuestas. Esta estructura define al integrón de clase 9 (Hochhut et al., 2001).
Finalmente indicar que existe una última clase de integrón a la que no se ha asignado
nombre, localizada en el plásmido pRVS1 de una cepa de V. salmonicida (nº de acceso:
AJ277063;Nemergut et al., 2008).
Es indudable el importante papel que juegan los integrones en la dispersión de
genes-R, siendo parte activa de la transferencia génica horizontal. Sin embargo, cabe señalar que
su participación no se limita a la dispersión de resistencias, sino también a la de otros genes
codificadores de funciones adaptativas, subrayando así el notable impacto que este flujo
horizontal de genes tiene en la evolución del genoma bacteriano (Nield et al., 2001;
Nemergut et al., 2008).
1.e.3. ELEMENTOS GENÉTICOS TRANSPONIBLES
En la dispersión de la resistencia también desempeñan un papel importante elementos
genéticos móviles capaces de desplazarse de una localización a otra del genoma. Se
denominan elementos genéticos transponibles y se caracterizan por codificar la maquinaria
enzimática necesaria para su movilidad (enzima transposasa y proteínas reguladoras), pero
no para su replicación. Desde su descripción, los elementos transponibles han despertado
mucho interés por su capacidad de promover cambios en el genoma, entre los que se
incluyen mutaciones, deleciones e inversiones. Además, pueden servir de sustrato para la
recombinación homóloga cuando se encuentran repetidos en el genoma. Por otro lado, en
su estructura suelen contener genes adicionales, la mayoría de los cuales codifican
resistencia a antimicrobianos, y unidades de captura génica como los integrones.
Dentro de los elementos genéticos transponibles destacamos los siguientes grupos en
un orden creciente de complejidad: secuencias de inserción, transposones de clase 1 o
compuestos, transposones de clase 2 o complejos y transposones de clase 4.
Secuencias de inserción
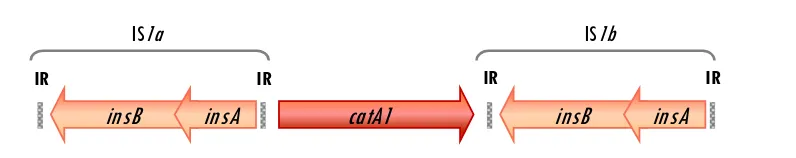
Las secuencias de inserción (IS) son los elementos transponibles más simples, son de
pequeño tamaño (normalmente menos de 2,5 kb) y poseen una organización genética
sencilla, incluyendo únicamente genes y regiones implicados en su movilidad: gen(es) que
codifica(n) una enzima transposasa, a veces también una proteína reguladora, y repeticiones
encontrar una o dos orfs ocupando casi toda la longitud del elemento transponible. Cuando
sólo hay una orf ésta codifica la enzima transposasa. Si hay dos orfs la transposasa puede
estar codificada por una de ellas o bien tratarse de una proteína de fusión formada por
ambas. Por ejemplo, IS1 es una secuencia de inserción de 768 pb que contiene dos orfs (insA
e insB, Figura 6) donde la más pequeña solapa con la mayor. El producto de insA reprime la
transcripción de las orfs, y por tanto, inhibe la transposición. Sin embargo, con una frecuencia
del 1% se dan fenómenos de cambio de pauta de lectura (‘frameshifting’) en la traducción
que permiten la expresión de la proteína de fusión InsAB’, que es la transposasa. En general
las transposasas comparten una estructura común, donde los dominios amino participan en
las funciones de unión al ADN y los extremos carboxilo son los dominios catalíticos. En cuanto
a las repeticiones terminales invertidas (IR), normalmente varían entre 10 y 40 pb de longitud,
marcan los límites de las IS y participan activamente en el proceso de transposición. Estas
secuencias terminales son reconocidas por la transposasa y actúan como sustrato en las
reacciones de corte y transferencia de hebras de ADN (Figura 6).
Por último señalar que otro rasgo muy frecuente en estos elementos es la duplicación
de un número limitado de nucleótidos presentes en el sitio de inserción. De esta manera la
mayoría de IS descritas aparecen enmarcadas por repeticiones cortas directas, cuya
longitud es específica de cada IS y suele variar entre 2 y 14 pb.
Figura 6.
Esquema de la secuencia de inserción IS1. Basado en los datos de secuencia del
plásmido R100 de Shigella flexneri (nº acceso: NC_002134).
Aunque en un principio el interés que despertaron estas secuencias estaba
relacionado con su capacidad para generar mutaciones en el genoma, pronto se descubrió
también su importante influencia en la dispersión y captación de genes bacterianos,
incluyendo aquellos que codifican resistencia a antimicrobianos. Así, por ejemplo, en la
actualidad y debido a la relevancia clínica que tiene la dispersión de beta-lactamasas de
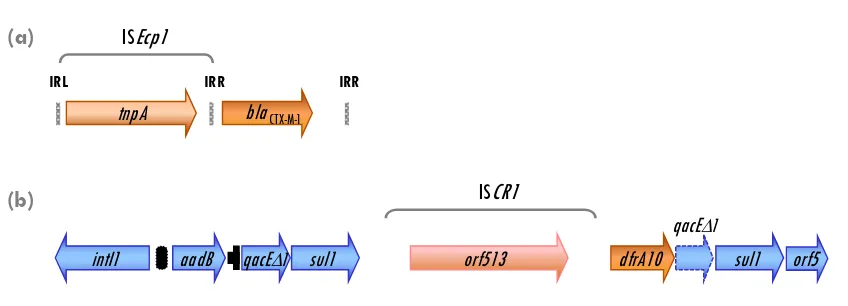
espectro extendido, son de especial interés la secuencia ISEcp1 y los elementos ISCR,
directamente relacionados con la movilización de estos determinantes-R.
La secuencia de inserción ISEcp1 pertenece a la familia IS1380 y se encuentra
frecuentemente asociada a genes de beta-lactamasas de espectro extendido de la familia