UNIVERSIDAD AUTONOMA METROPOLITANA
UNIDAD IZTAPALAPA
DIVISION CIENCIAS BIOLOGICAS Y DE LA SALUD
LICENCIATURA BlOLOGlA EXPERIMENTAL
INFORME FINAL DEL SERVICIO SOCIAL
"IDENTIFICACIÓN DE ALTERACIONES CROMOSÓMICAS
EN
PACIENTES CON LEUCEMIA
AGUDA LINFOBLÁSTICA POR EL
MÉTODO DE HIBRIDACIóN
/N
SlTU
CON FLUORESCENCIA"
PRESENTADO POR:
Título
del
proyecto
“Identificación de alteraciones cromosómicas en pacientes con
Leucemia Aguda Linfoblástica por el método de Hibridación
In
I
1
I N D I C E
1. Introducción ...
1
1 .l. Definicton . I 1 ... 1.2. Clasificaclon . I
1
...2.
3.4.
5.
6. 7. 8. . . ~, ~y.?
1.3. Análisis citogenético convencional...
.3,(
.1.4. Análisis con la metodología de hibridación in
situ
con fluorescencia...
d, (2..
>?
n
Justificaclon ... 6 ..
$2
I ? z
' >
Objetivos ... 7 *.ie
?' I , 1.- 4: U?,-
Hipótesis ... .7T
-
I :i 1 .-,r
z
, . f- Material y Metodología 8 < x 5.1. Poblaclon de estudio ... 8 F . -1o
;> , _ * -_, I I I . . ." , ' .- . ... _ . I I -,..,
5.2. Obtención de la muestra de médula ósea...
85.3. Estudio cromosómico
...
.85.3.1. Análisis de resultados del estudio cromosómico
...
95.4. Hibridación in situ con fluorescencia
(FISH)
...
95.4.1. Análisis del estudio con
FISH
...
10Resultados ...
. I 2
Discuslon..
...I 8
Concluslon . I 20
1.
INTRODUCCION
1 . l . Definición.
La leucemia es un tipo de cáncer originado por el crecimiento descontrolado de lineas
celulares inmaduras de la estirpe linfoide o mieloide, que se encuentran bloqueadas en un estado
particular de su diferenciación celular.(Pui, 1995).
1.2. Clasificación.
Las leucemias se dividen en dos grupos, con base en el grado de maduración de las
células involucradas y en su capacidad proliferativa. El primer grupo se refiere a las leucemias
agudas, que presentan cklulas inmaduras o blastos con alto grado de proliferación; en tanto que el
segundo grupo comprende a las leucemias crónicas con maduración intermedia o terminal y con
menor grado de proliferación con respecto a las anteriores.(Rivera-Luna, 1996).
Dentro de la leucemia aguda existen dos tipos principales: la leucemia aguda linfoblástica
(LAL), que afecta a las células precursoras de los linfoblastos producidos en las glándulas linfáticas
y en la médula ósea (MO); y la mieloide o no linfoblhstica (LANL), la cual muestra alteraciones
clonales de los precursores mieloides. El diagnóstico de las leucemias se basa principalmente en
el estudio citomorfológico de la MO.(Bonilla, 1993).
En este proyecto, nos concentramos en la LAL, ya que tienen una alta incidencia en la
etapa infantil y es uno de los tipos de neoplasia que con mayor frecuencia se atienden en el
Instituto Nacional de Pediatría (INP).(Compendio del Registro Histopatológico, 1996).
1.2.1. Clasificación citogenética.
Aproximadamente, entre el 70% y el 80% de los pacientes con LAL presentan lineas
celulares con alteraciones cromosómicas, tanto numéricas como estructurales, en las células de la
MO. De hecho, el analisis citogenético de estos pacientes ha llevado a la identificación de
alteraciones crornosórnicas no aleatorias que tienen importancia en la clasificaci6n diagnóstica, el
establecimiento del pronóstico, así corno gran influencia en la respuesta al tratamiento. Las
Cuadro l. Alteraciones cromosdmicas estructurales mas frecL
Tomado de Heim y Mitelman, 1992.
Ites en LAL.
!
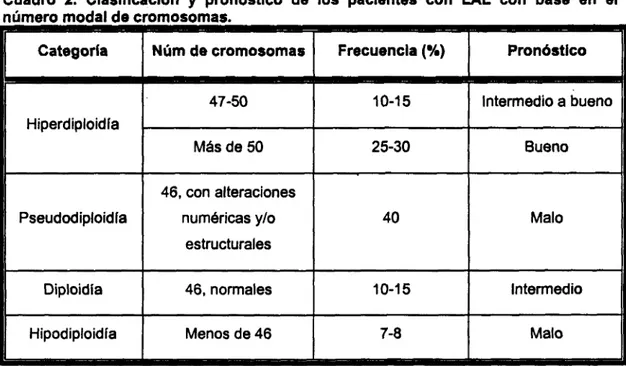
Con base en las alteraciones cromosbmicas encontradas en las celulas de los pacientes
con LAL, ha sido posible clasificarlos en subgrupos de acuerdo al número modal de cromosomas
presentes, esta clasificacibn tiene valor pronbstico (cuadro 2).
Cuadro 2. Clasificacidn y prondstico de
los
pacientes con LAL con base en el número modal de cromosomas.estructurales
Diploidia 46, normales 10-1 5 Intermedio
Hipodiploidia Menos de 46 7-8 Malo
Los pacientes con cariotipo hiperdiploide es decir, con aumento en el número
cromosdmico, presentan las tasas mas altas de remisidn o erradicaci6n de las celulas leucémicas
de la MO,
lo
cual indica que dicha alteración numérica esth asociada con buen pron6stico. Lasremisiones m& breves con sobrevida m& corta, ocurren en individuos que presentan las
translocaciones t(4;11), t(8;14), t(9;22), o hipodiploidias (disminuci6n del número normal de
cromosomas).(Salamanca, 1992).
Se ha demostrado que el aumento de cromosomas en las hiperdiploidias no ocurrre al
azar, ya que los cromosomas 21, X, 8, 10, 6, 17 y 18 se presentan con mayor frecuencia en este
tipo de alteraci6n. Se ha informado que las alteraciones numericas en la LAL tienen gran
importancia en el prondstico de
los
pacientes.(Martinet
a/, 1996)(Moorman et a/, 1996).1.3. Analisis citogen6tico convencional en MO.
Como se ha mencionado anteriormente, el estudio de
los
cambios cromos6micos en lasneoplasias ha permitido avanzar en el entendimiento del fendmeno de la transformaci6n maligna y
descubrir alteraciones citogenéticas útiles para el diagndstico y el pron6stico de las entidades
neoplásicas. Las anormalidades cromosómicas han sido eficazmente detectadas por medio de
análisis citogenético usando técnicas para obtener bandas cromos6micas. Sin embargo, este tipo
de analisis presenta algunas limitaciones:
a) El estudio se debe realizar en muestras de M 0 sin estimulaci6n mitogénica, con el objeto de
que las celulas normales no se estimulen, para evitar que se obtenga un resultado sesgado. Las
células que
se
analizan son aquellas que alcanzan espontdneamente. la metafase,desafortunadamente el número de estas es pequeno, por lo que
se
excluye del analisis un gran número de celulas que permanecen en interfase.b) Es muy difícil obtener resultados cuando las poblaciones celulares presentan bajo índice
mitótico, como es el caso de las celulas que permanecen en estado quiescente. Tampoco se
puede realizar el estudio en c6lulas totalmente diferenciadas, como son los neutr6filos.
c) Los cromosomas de M 0 de los pacientes leucemicos con frecuencia muestran mala calidad en
1.4. Análisis con la metodología de Hibridación in situ con fluorescencia.
El advenimiento de nuevos métodos de genética molecular, como la hibridación in situ con fluorescencia (FISH), han permitido la identificación de alteraciones cromosómicas en cualquier
etapa del ciclo celular. El fundamento de esta técnica radica en la complementariedad que existe
entre dos moléculas de DNA, una de las cuales es el DNA que se está analizando (DNA problema)
y la otra es una sonda de DNA conocido que se encuentra marcada.(Trask, 1993)
Las sondas son moléculas de DNA unidas covalentemente a fluorocromos o haptenos,
tales como la biotina o la digoxigenina. Los haptenos se detectan indirectamente después de la
hibridación, mediante anticuerpos conjugados con fluorocromos como son la fluoresceína, la
rodamina y el rojo Texas.(Swiger, 1996)
Para llevar a cabo la técnica de FISH, es necesario desnaturalizar tanto la sonda como el
DNA en estudio, para obtener DNA monocatenario. Posteriormente la sonda y la muestra de DNA
se someten a condiciones que promuevan la unión entre la cadena sencilla de la muestra de DNA
y la cadena de DNA de la sonda.(Trask, 1993).
Existen diversos tipos de sondas que pueden ser empleados en la detección de
anormalidades cromosómicas. Las tres clases principales de sonda son: 1) Sondas que identifican
genes, llamadas también de secuencia única; 2) Sondas a-satélite o centroméricas; y 3) Sondas
de cromosomas completos conocidas como “painting”. Con base en esto, las alteraciones
numéricas pueden ser detectadas con sondas oc-satélite dirigidas a secuencias centroméricas; las
aberraciones estructurales pueden ser identificadas mediante el uso de sondas para
los
cromosomas completos o bien, utilizando secuencias únicas que permiten detectartranslocaciones, inversiones o deleciones. Aunque este análisis proporciona sólo una cantidad
limitada de información en cuanto al cariotipo de una célula, al ser combinado con el análisis
citogenético resulta de gran valor para el diagnóstico, clasificación de la enfermedad y monitoreo
de la respuesta a la terapia aplicada a los pacientes con leucemia.(Dewald, 1995) (Anastasi, 1993).
El método de FISH ofrece varias ventajas con respecto al análisis citogenético
convencional, entre ellas:
a) Es posible obtener información tanto de células metafásicas como de núcleos en interfase.
b) No se requiere de un volumen grande de muestra celular
c ) Permite detectar pequeñas subpoblaciones, incluyendo células residuales leucémicas.
d) El método es rápido y su interpretación no requiere de un entrenamiento especial como en el
La técnica de FISH ha sido empleada en varios campos, tanto en células somáticas de
diversos tipos como en células germinales. Se ha aplicado en el análisis de dalio cromosómico
producido por agentes físicos o químicos, ubicación de genes o secuencias en los cromosomas,
diagnóstico clínico, toxicología molecular, homología cromosómica entre algunas especies y
también ha sido útil en la detección de alteraciones cromosómicas de células germinales como
los
espermatozoides.(Swiger, 1996).Las ventajas que ofrece la técnica de FISH hacen que sea una metodología elegible para ser
aplicada durante el diagnóstico y seguimiento de los pacientes con leucemia. Arkesteijn et al y Heinonen et
al,
han buscado alteraciones numéricas en pacientes con diversos tipos de leucemias agudas, con la finalidad de detectar células leucémicas durante el tratamiento delos
pacientes, basándose en las alteraciones numéricas encontradas por citogenética convencional al momentodel diagnóstico. Sus resultados han mostrado que en la mayoría de
los
casos, la presencia de alteraciones numéricas detectadas con sondas centroméricas por FISH, correlaciona con lapresencia de blastos leucémicos identificados por citornorfología. Sin embargo, en un grupo de
pacientes se encontraron discrepancias en
los
resultados, este hallazgo se atribuyó a: a) la aparición de nuevas clonas con alteraciones diferentes a las observadas al momento deldiagnóstico; b) la posibilidad de que algunos grupos de células leucémicas no tuvieran la
citomorfología clásica de un blasto; y c) el estudio citogenético en el cual se basaron, se lleva a
cabo únicamente sobre células que en ese momento se encuentren en proliferación y no toma en
cuenta a las células quiescentes, las cuales podrían portar alteraciones que no se detecten.
En
el
laboratorio en el que se efectuó este trabajo, se ha iniciado la búsqueda de alteracionesnuméricas con sondas centroméricas utilizando FISH, esto se ha llevado a cabo exclusivamente en
aquellos casos en los que no se identificaron completamente los cromosomas involucrados en la
alteración, mediante el estudio cromosómico convencional. Dentro de
los
casos estudiados se identificó a un paciente portador de un polimorfismo cromosómico para la sonda 13/21, es decir, elpaciente presentó una variante en tamaño para esta sonda; al analizar sus células de M 0 en
interfase se encontró que una señal para la sonda 13/21 pasaba desapercibida porque su tamalio
era muy pequeño.
Con base en esta observación, se plantea la posibilidad de que los resultados obtenidos con
FISH en la búsqueda de alteraciones numéricas durante el tratamiento de los pacientes, estén
influidos en parte por la presencia de polimorfismos de las sondas centroméricas, ya que se ha
2.
JUSTlFlCAClON
Las limitaciones metodológicas del estudio cromosómico, hacen que en ocasiones no se
obtengan resultados concluyentes, que éstos no reflejen el verdadero estado de las poblaciones
celulares leucémicas y por ello no se tenga la suficiente información para dar seguimiento al
paciente durante el tratamiento. En el presente trabajo, se estudió un grupo de pacientes con LAL
en el momento del diagnóstico, con cariotipos diploides e hiperdiploides, los cuales se analizaron
con dos sondas centroméricas para cromosomas que se involucran con frecuencia en alteraciones
numéricas, como
lo
son los cromosomas 1 O y 13/21.La finalidad de este trabajo fue : a) Corroborar los resultados del estudio cromosómico; b)
detectar con certeza la participación de estos cromosomas en los pacientes con hiperdiploidía, en
los cuales no se pudo llevar a cabo la identificación de cada cromosoma al aplicar la técnica de
bandas G y c) evaluar la utilidad de estos cromosomas como marcadores de neoplasia en la ,
evolución de los pacientes, con base en la probable presencia de polimorfismos centroméricos de
3.
OBJETIVOS
O Complementar el estudio citogenético convencional con el método de FISH, para identificar
alteraciones numéricas en pacientes con LAL. r
O Identificar por medio de FISH alteraciones numéricas que sirvan como marcadores de esta enfermedad y permitan el seguimiento de
los
pacientes durante su evolución.4. HIPOTESIS
O La aplicación conjunta de la citogenética convencional y el mittodo de FISH, incrementa la
información acerca de las características cromosómicas de pacientes con LAL, dado que se
combina el análisis de cklulas en metafase y de núcleos en interfase.
O La identificación de marcadores de alteraciones numéricas con FISH debe realizarse desde el
inicio de la enfermedad, ya que permite evaluar si estos son informativos cuando la leucemia está
5. MATERIAL
Y
METODOLOGIA
5.1. Población de estudio.
Se estudiaron 18 individuos en edad pediátrica considerada hasta
los
18 años de edad,captados entre noviembre de 1997 y abril de 1998, con diagnóstico de LAL basado en
los
criterios propuestos por el Grupo Francés-Américano-Británico (FAB). Los pacientes se encontrabanvírgenes de tratamiento al momento del diagnóstico, con inmunofenotipo característico de la
enfermedad y algunos de ellos con estudios cromosómicos no concluyentes, ya sea por presentar
cromosomas de mala calidad en su estructura o por tener bajo índice mitótico. Con base en el
numero modal obtenido con el estudio citogenético, la población de estudio se dividió en dos
grupos:
Nueve pacientes con cariotipo diploide (46 cromosomas)
Nueve pacientes con cariotipo hiperdiploide (más de 46 cromosomas).
5.2. Obtención de la muestra de MO.
Se obtuvo una muestra de 4 a 6 m1 de MO, de la cresta iliaca posterior por aspiración, en
una jeringa heparinizada y bajo condiciones de esterilidad.
5.3. Estudio cromosómico.
Siembra Y cosecha de MO. Se llevaron a cabo dos metodologías: 1)Técnica directa y 2)
Cultivo de 24 horas. En ambos casos: en un tubo de centrifuga se agregaron 7ml de medio de
cultivo (Amniomax, Gibco), 0.2ml de colcemida (lop1 I ml, Gibco) y de 0.5 a 1 ml de la muestra de
MO. Se incubó a 37°C por 30min. Se centrifugó por 10min a 1500rpm y al paquete celular
obtenido se le añadieron 7ml de una solución hipotónica (0.075M KCI, Merck) incubando durante
l h a 37°C. Se centrifugó nuevamente y se procedió a fijar y lavar con una solución de Carnoy
(metanol-ácido acético
3:1,
Merck).(Williams, 1986).Elaboración de las laminillas. Sobre un cubreobjetos limpio y frío se aplicaron de 4 a 6
gotas de la muestra celular, soplando fuertemente la superficie de la laminilla; finalmente se secó a
la flama. La tinción se llevó a cabo siguiendo la técnica de bandas G, en breve: las laminillas se
trataron con tripsina
(OS%,
Gibco) por lOseg, se colocaron en una solución amortiguadora deSorensen (sales de fosfato y sodio) y se tiñeron con los colorantes Wright y Giemsa (Merck), 2min
en cada uno. Se lavaron con agua corriente para eliminar el exceso y se secaron al aire.(Verma,
5.3.1. Análisis de resultados del estudio crornosórnico.
Se analizaron de 10 a 20 metafases por paciente, y con base en los criterios de la ISCN de
1995 se dio nomenclatura y se determinó si las alteraciones eran clonales.(lSCN, 1995).
5.4. Hibridaci6n in situ con fluorescencia (FISH).
La técnica de FISH se desarrolló de acuerdo con las recomendaciones del manual de la
casa comercial Oncor.
Desnaturalización de la muestra de DNA. Se hicieron las preparaciones sin secar a la
flama y se colocaron en una solución acondicionadora de citrato de sodio (2xSSC) Oncor, a 37°C y
pH 7 por 30min, se deshidrataron en etanol (Merck) al 70%, 85% y 100% a temperatura ambiente
por 2 min en cada uno y se dejaron secar al aire. Posteriormente, las laminillas se trataron con una
solución desnaturalizadora (formamida 70% / 2xSSC) Oncor, pH 7 a 72°C durante 2min deshidratando inmediatamente en etanol al 70%, 85% y
loo%,
frío (k 4"C), durante 2min en cadauno. Posteriormente las preparaciones se secaron al aire.
Sondas. Se utilizaron las sondas a-satélite (centroméricas) de Oncor para los
cromosomas 10 y 13/21 marcadas con biotina y digoxigenina respectivamente. Se descongeló la
sonda, se tomaron 1 Spl y se mezclaron con 30p1 de hybrisol VI Oncor (formamida 65% / 2xSSC).
Esta mezcla se desnaturalizó a 72°C y se colocó en un baño de hielo hasta el momento de la
hibridación.
Hibridación. Se aplicaron lop1 de la sonda sobre la laminilla y se montó con un cubreobjetos (22x22mm), sellando los bordes con goma (rubber cement) para evitar la
deshidratación de la laminilla. Se incubó en una cámara húmeda a 37°C por un período de 16h.
Se retiró el sellador con pinzas y se removió el cubreobjetos, la laminilla se colocó en una solución
0.5xSSC a 72°C por 5min y se transfirió inmediatamente a una solución de lavado (125pl tween 20
Detección
v
tinci6n. Para la detección se aplicaron sobre la laminilla60p1
de avidina ligada con rodamina (fluorocromo rojo) y 60pl de antidigoxigenina ligada con isotiocianato defluoresceina (fluorocromo verde), cubriendo rápidamente con un cubreobjetos de plástico. La
laminilla se incubó a 37°C durante 15min en una cámara húmeda. Después de remover el
cubreobjetos se enjuagó la laminilla en la solución de tween durante 2min, este proceso se repitió
tres veces. Finalmente, se aplicaron sobre la laminilla 18p1 de diamino-fenil-indol (DAPI), que es
un
contratinte fluorescente de color azul que permite observar los núcleos y metafases de lapreparación.
5.4.1. Análisis del estudio con FISH.
2 2 2 5 2 1
Por cada paciente se revisaron 1000 núcleos en interfase con un microscopio de
fluorescencia equipado con un filtro de excitación de 560-620 nm para triple banda, con este filtro
es posible observar tres fluorocromos de diferente color a la vez. Las lecturas se llevaron a cabo
con el objetivo 1 OOx. Las laminillas fueron codificadas por una persona ajena a este trabajo.
Los criterios utilizados para evaluar a los núcleos fueron los siguientes:
Con la sonda =-satélite 10, las células normales (disómicas) muestran dos señales. Las
células monosómicas muestran solamente una señal, en tanto que en las trisómicas, los núcleos
aparecen con tres señales. Las señales deberán presentarse separadas entre
sí
a una distancia de por lo menos un dominio de señal. (Fig. 1).La sonda =-satélite 13/21 marca ambos cromosomas porque no se han logrado aislar de
manera independiente, por lo tanto una célula normal presentará 4 señales. Una célula trisómica
para alguno de estos cromosomas mostrará 5 señales y en una monosdmica se observarán 3
señales.
Con la metodología de FISH se pueden encontrar valores basales de aneuploidía en
sujetos sanos, debido a efectos técnicos o bien, a errores durante la división celular que se
presentan normalmente. Los valores basales de monosomía y trisomía para las sondas utilizadas,
se calcularon a partir de las células de M0 de los pacientes con cariotipo normal. En la literatura
En donde:
-
S = Promedio obtenido de las células aneuploldes (monosómicas o trisómicas) de los pacientes
diploldes.
r; = Desviacion estandar del promedio obtenido. (tlchter. 1996)
Se calcularon de manera independiente
los
valores basales para monosomía y trisomia. ' 03
1.8 +:
.
,*'r.J 1 if * \
m .
así como para cada sonda utilizada
METAFASE
6.
RESULTADOS
Se estudiaron las muestras celulares de 18 pacientes con LAL por medio de análisis citogenético y FISH, utilizando para este último las sondas centroméricas de los cromosomas 10 y 13/21. Los resultados obtenidos se clasificaron en tres grupos:
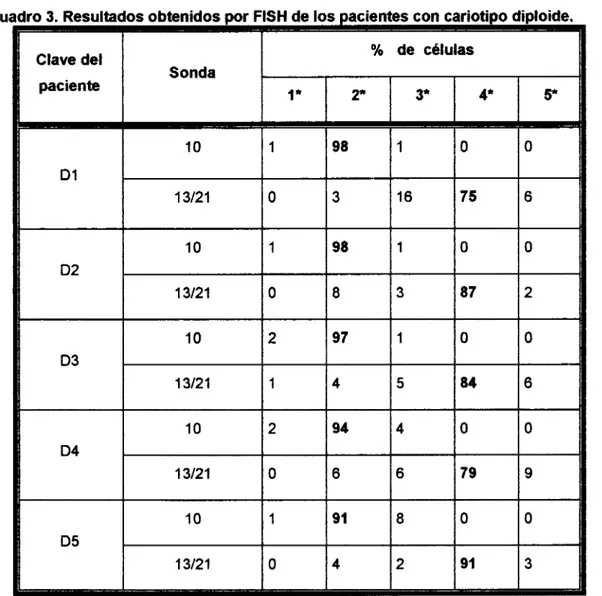
0:. Grupo l. Pacientes diploides por citogenética convencional y por FISH.(Cuadro 3).
Cinco de los 9 pacientes con cariotipo normal (Dl, D2, D3, D4 y D5), presentaron disomía para ambos cromosomas analizados por medio de FISH. Los núcleos exhibieron 4 señales correspondientes a la sonda 13/21 y dos señales de la sonda 10 en la mayor parte de las células analizadas, el porcentaje de células disómicas fue de 75 a 91% y de 91 a 98% para cada sonda respectivamente. Con estos pacientes se calcularon los valores de corte para las monosomías y trisomías (cuadro 4); se descartaron del grupo de pacientes diploides 4 casos que presentaron más del 20% de sus células con monosomía o trisomía, tomando en cuenta que el valor basal
más
alto que refiere la literatura para estas estimaciones es del 15%.(Lichter. 1996).Los pacientes que discreparon se separaron en el grupo 3 y se describirán posteriormente.
0:. Grupo 2. Pacientes hiperdiploides por citogenética convencional y FISH.(Cuadro 5).
En los 9 pacientes analizados se corroboró con base en los valores basales de aneuploidía, la presencia de líneas celulares hiperdiploides encontradas en el estudio citogenético; el análisis del FISH reveló la participación de los cromosomas 10 y/o 13/21 en el aumento del número modal. Todos los pacientes, con excepción del H1 y del H5 presentaron una línea celular trisómica para el cromosoma 10, en 15% a 45% de las células examinadas. Las muestras celulares de los pacientes H1, H2, H3, y H5, presentaron células con 5 señales con la sonda 13/21 en 20% a 36% de los núcleos revisados. En dos pacientes, H2 y H3, ambas sondas revelaron la presencia de trisomía; la sonda de los cromosomas 13/21 mostró trisomía en 4 casos en tanto que la del cromosoma 10 fue trisómica en 6 casos.
.. .. .
*:
* Grupo 3. Pacientes normales por citogenética convencional y aneuploides por FISH.
Como se mencionó anteriormente, en cuatro casos clasificados como diploides por citogenética convencional, los resultados del FISH no coincidieron con
los
obtenidos mediante elanálisis cromosómico (cuadro 6). En estos pacientes se detectaron por FISH líneas celulares monosómicas y trisómicas que no fueron evidentes por citogenética convencional.
Los casos NC2 y NC3, mostraron tres señales para el cromosoma 10 en 35% y 22% de las células, respectivamente.(fig. 2:A y B). Los casos NC1 y NC4, mostraron el 24% de sus células
con 3 señales al analizarse con la sonda 13/21. En ambos pacientes se observó que los núcleos con 4 señales, mostraban una señal de menor tamaño con respecto a las otras tres.(Fig. 2:C y D).
Cuadro 3. Resultados obtenidos por FISH de los pacientes con cariotipo diploide.
I
11
L
% de c6lulas
ll
Cuadro 4. Valores basales de monosomías y bisomias establecidos para las sondas
OC- satélite de los cromosomas 10 y 13/21. I
i
1
AneuploidíaI
Cromosoma1
Núm de sefialesI
Valor basal1
10 1 3
Monosomía i
13/21 3 22
10 3 13
I
1
TrisomíaI
I
I
I1
I
13/*1I
5
I
l3
I1
- ~. .. . . .. ~
Cuadro 6. Pacientes con LAL cuyo anhlisis cromosómico
no
concuerda con el resultadoobtenido por FISH.
Núm. modal
46
46
46
46
% de celulas
VII.
DISCUSION
El análisis citogenético es el método convencional por medio del cual se establece el cariotipo de los pacientes con LAL; sin embargo presenta limitaciones, ya que la mala calidad de
los
cromosomas analizados y el bajo índice mitótico de las células leucémicas impiden realizar un estudio resolutivo.(Anastasi, 1993).Se ha demostrado que el análisis con el método de FISH resulta de gran ayuda en estos casos, ya que se puede aplicar sobre células en interfase; por consiguiente el número de células analizadas es mayor, con
lo
cual se incrementa la posibilidad de detectar subpoblaciones celulares con alteraciones cromosómicas.(Romana, 1993).En este trabajo, las muestras de M 0 de los pacientes estudiados se hibridaron con las sondas =-satélite de los cromosomas 10 y 13/21. Las sondas se eligieron porque estos cromosomas se ven con frecuencia involucrados en hiperdiploidías, principalmente el cromosoma 21. Desafortunadamente, la secuencia centromérica del cromosoma 21 no se ha logrado aislar del cromosoma 13, por lo que al utilizarla ocurre una hibridación a z a d a produciéndose 4 señales. (Bossuyt, 1995). Por otro lado, la deteccción de la trisomia del cromosoma 10 es importante
porque se le atribuye un valor de buen pronóstico para el paciente (Martin, 1996)(Moorman, 1996).
En el presente estudio, en 14 de los 18 pacientes hubo concordancia entre los resultados obtenidos por medio del análisis citogenético y lo observado con el método de FISH. En todos
los
pacientes hiperdiploides, se confirmó mediante FISH, la presencia de líneas celulares trisómicas para el cromosoma 10 y/o para el cromosoma 21. En el grupo de pacientes estudiados, el cromosoma 10 fue más informativo en cuanto a la detección de trisomías, ya que identificó 7 casos, en comparación con la sonda 13/21 con la cual únicamente se detectaron 4 casos trisómicos. Aunque la muestra es pequeña, cabe mencionar que este dato difiere de lo descrito en la literatura, en donde se refiere que el cromosorna 21 participa en la hiperdiploidia con mayor frecuencia que el cromosoma 10. (Moorman, 1996).
Hubo discrepancia en el estudio cromosómico y de FISH en 4 pacientes en los que se había establecido un cariotipo normal.
En
el paciente NC2, el cromosoma 10 reveló una línea celular con trisomía, este hallazgo se apoyó en un estudio colateral de citometría de flujo (CF) que se lleva a cabo como parte de un proyecto principal sobre LAL. La CF permite cuantificar el contenido de DNA de miles de células en interfase y en este caso mostró una línea celular hiperdiploide lo cual coincide con lo obtenido por el estudio de FISH.El caso NC3, mostró trisomía del cromosoma 10, en este paciente también se tiene el
trisomía 10 no detectado por CF ni por citogenética clásica, o que la hiperdiploidía sea discreta en cuanto a número, de tal forma que no sea posible encontrar diferencia en cuanto al contenido de DNA entre la línea normal y la trisómica. Como una tercera posibilidad, se puede plantear la presencia de hibridación cruzada con otro cromosoma, éste evento es poco frecuente, pero se ha referido que ocurre entre los cromosomas 21 y 22.(Verlinsky, 1995).
En los pacientes NC1 y NC4, la sonda 13/21 mostró una línea celular con tres señales positivas es decir, con monosomía para uno de estos cromosomas. Particularmente, en el caso NC1 el estudio por CF muestra una línea hiperdiploide lo cual no coincide con lo obtenido por
FISH. En el caso NC4 no se contó con el estudio de CF que apoyara este resultado. Para ambos
casos, se requiere descartar o apoyar el hallazgo de la monosomía usando una sonda de
secuencia específica para el cromosoma 21; y para el paciente NC1 es necesario ensayar otras sondas con el objeto de detectar la hiperdiploidía observada por CF, ya que la sonda del
cromosoma 1 O mostró disomía.
Se ha informado recientemente de individuos con variantes polimórficas para la región oc-
satélite de los cromosomas 13/21 y 18, estos polimorfismos se reflejan en un aumento o disminución del tamaño de la región correspondiente. Una señal polimórfica pequeña al ser observada con FISH en interfase, puede pasar desapercibida.(Vedinsky, 1995) (Verma, 1997). Este hallazgo puede explicar
los
resultados no concordantes de los pacientes NC1 y NC4, de hecho en estos pacientes se identificó una señal de menor tamaño en algunos de sus núcleos.Como se refirió anteriormente, en el laboratorio donde se llevó a cabo el presente estudio, se tiene el antecedente de un paciente con LAL que fue analizado con la sonda centromérica
13/21. El análisis reveló una monosomía para alguno de estos cromosomas, pero no coincidía con el resultado del estudio cromosómico, en el cual se mostraba una línea celular hipotriploide con un cromosoma G supernumerario, esta información sugería la existencia de un polimorfismo en la
región m-satélite del cromosoma 21, lo cual se comprobó al utilizar una sonda de tinción completa específica para dicho cromosoma, con la cual se hizo evidente una línea celular disómica y otra trisómica en el mismo paciente, descartándose así la monosomía.
Para el cromosoma 10, no se ha referido especialmente que los eventos polimórficos sean frecuentes, no obstante, no se puede descartar la posibilidad de su presencia. La mayoría de los trabajos referidos con FISH sobre células en interfase son de diagnóstico prenatal, en los cuales se buscan aneuploidías de los cromosomas 13, 18,21, X y Y , ésta puede ser la causa por la cual aún no se han detectado polimorfismos en otros cromosomas del complemento humano.
células quiescentes pueda llevar a un diagnóstico equivocado y como consecuencia a un tratamiento inadecuado que dificultará el restablecimiento de los pacientes. En cuanto a las variaciones de la región =-satélite de los cromosomas, la presencia de polimorfismos puede llevar a confusión y a una estimación equivocada de las poblaciones celulares leucémicas durante el seguimiento de los pacientes. La identificación temprana de estos marcadores cromosómicos pueden ser de gran ayuda para el seguimiento del paciente leucémico durante la evolución de su padecimiento.
8.
CONCLUSION
>
La información proporcionada por la técnica de FISH complementa en gran medida elanálisis citogenético convencional en el estudio de pacientes con LAL, de tal manera que la aplicación conjunta de estas metodologías proporciona una mayor información que
conduce a un diagnóstico
más
acertado, y consecuentemente a un mejor tratamiento para estos pacientes.>
La hibridación in situ con fluorescencia, es una técnica simple y efectiva que permitedetectar anormalidades cromosómicas relevantes en el diagnóstico y pronóstico de los pacientes con IAL, principalmente en aquellos casos en
los
que el análisis citogenético no es satisfactorio. Este método, es considerablemente útil en la identificación de marcadores cromosómicos de valor pronóstico para el seguimiento delos
pacientes.3 La identificación temprana de polimorfismos de las secuencias centroméricas utilizadas en la búsqueda de aneuploidías es muy importante, ya que estas variaciones pueden dar
9.
LITERATURA CITADA
Anastasi J. Fluorescence in sito hibridization in leukemia. Applications in diagnosis. Annals of the
New York Academy of Science Clinical. 1993;667:214-224.
2 2 2 5 2 %
Arkesteijn G, Erpelinck S , Martens A, Hagemeijer A , Hagenbeek A. The use of FISH with
Chromosome-specific repetitive DNA probes for the follow-up of leukemia patients. Cancer Genetic
Citogenetic. 1996;88:69-75.
Bonilla M, Rivera R, Meza C. Clasificación de las leucemias. Criterios Pediátricos. Instituto Nacional
de Pediatría. 1993;9:65-68.
i
d
o
6 . 0
Bossuyt P, Van Tienen M, Gruyter L, Smets V, Dumon J , Wanters J. Incidence of low-fluorescence c:,
,-:
o
a
7 ,-1 =-satellite region on chromosome 21 escaping detection of aneuploidy at interphase by FISH. r + Z
5 "
Cytogenetics Cell Genetic. 1995;68:203-206.
$ Q
2
Clare N, Hansen K. Cytogenetics in the diagnosis of hematologic malignancies. Hematology/
:.
8
Oncology Clinics of North America.1994; 8:785-807..? ;D
'A
77..:
'51
Compendio del Registro Histopatológico de Neoplasias Malignas en México. Morbilidad y i5
Mortalidad BIENIO 1993-1994. i a edición. Secretaría de Salud. 1996; 67-70. $7
.a r
c!
."
. -
,
' m ."f >
Chromosome in situ hibridizafion manual. 1996. Oncor, Inc.
Dewald
G ,
Wright P. Chromosome abnormalities in the myeloproliferative disorders. Seminars inOncology. 1995; 22:341-354.
Heim S , Mitelman F. Cytogenetics analysis in the diagnosis of acute leukemia. Cancer
Supplement.1992; 70:1701-1769.
Heinonen K, Mahlamaki E. Detection of numerical chromosome abnormalities by FISH in childhood
acute lymphoblastic leukemia. Cancer Genetic Cytdgenetic. 1&3;87:123-126.
Lichter P, Fischer K,
Joos
S . Fink T. Baudis M. Potkul R, Oh1 S , Solinas-Toldo S , Weber R.Stilgenbauer S , Bentz M, Dohner H. Efficdcy of current molecular cytogenetic protocols for the
Martin P, Look T, Schnell S, Hams M, Pullen J, Shuster J, Carro1 A, Pettenati M, Rao N.
Comparison of fluorescence in situ hibridization, cytogenetic analysis, and DNA index analysis to
detect chromosomes 4 and 10 aneuploidy in pediatric acute lymphoblastic leukemia: a pediatric
oncology group study. Journal of Pediatric Hematology / Oncology. 1996;18(2):113-121.
ISCN 1995: An international system for human cytogenetic nomenclature.(l995). Ed. Karger &
Cytogenetics and Cell Genetics.
Moorman A , Clark R, Farrell D, Hawkins J, Martineau M, Secker-Walker L. Probes for hidden
hyperdiploidy in acute lymphoblastic leukaemia. Genes, Chromosomes & Cancer. 1996; 16:40-45.
Pui C-H. Chilhood leukemias. The New Eng Journal of Medecin. 1995;1618-1630.
Raimondi
S,
Roberson P, Pui C-H, Behm F, Rivera G. Hyperploid (47-50) Acute lymphoblasticleukemia in children. Blood.1992; 79:3245-3252.
Rivera-Luna R, Meza-Coria C, Cárdenas-Cardós R, Martínez-Avalos A, Leal-Leal C. Navarro-
Alegría Y. Inmunophenotypes in mexican childrens with acute lymphoblastic leukemia: an
experience from a single institution. International Journal of Pediatric Hematology / Oncology. 1996;
3157-61.
Romana S, Cherif D, Le Coniat M, Derré J, Flexor M, Berger R. In situ hibridization to interphase
nuclei in acute leukemia. Genes, Chromosomes & Cancer.1993; 8:98-103.
Salamanca F. Citogenética y cáncer. Genética y Cáncer. Gaceta Medica de México.
1992;128(2):110-117.
Swiger R, Tucker J. Fluorescence In Situ Hibridization: A Brief Review. Enviromental and
Molecular Mutagenesis.1996; 27:245-254.
Trask 6, Allen
S,
Massa H, Fertitta A , Sachs R, Van Den Engh G, Wu M. Studies of metaphase andinterphase chromosomes using fluorescence
in
situ hibridization. Cold Spring Harbor Symposiumon Quantitative Biology. 1993; LVlll:767-775.
Veriinsky Y, Ginsberg N, Chmura M, Freidine
M,
White M, Strom C, Kuliev A. Cross-hibridization ofthe chromosome 13/21 alpha satellite DNA probe to chromosome 22 in the prenatal screening of
Verma R , Babu A. Human chromosomes. Manual of basic techniques. Pergamon Press. 1989;47-
i s .
Verma R , Batish S , Gogineni S, Kleyman S, Stetka D. Centromeric alphoid DNA heteromorphisms
of chromosome 21 revealed by FISH-technique. Clinical Genetics. 1997;51:91-93.
Williams DL, Harber J, Murphy SB, Look AT, Kalwinsky DK, Rivera G, Melvin SL, Stass S, Dah1 GV.