CARACTERIZAÇÃO MOLECULAR DA RESISTÊNCIA À CLINDAMICINA EM ISOLADOS CLÍNICOS DE STAPHYLOCOCCUS AUREUS
Texto completo
(2) Palavras-chave: Staphylococcus aureus, Clindamycin, Inducible Resistance, erm genes.. Modalidade de Participação: Pesquisador. CARACTERIZAÇÃO MOLECULAR DA RESISTÊNCIA À CLINDAMICINA EM ISOLADOS CLÍNICOS DE STAPHYLOCOCCUS AUREUS 1 Aluno de pós-graduação. angelitabotega@yahoo.com.br. Autor principal 2 Aluno de pós-graduação. marissabolson@gmail.com. Co-autor 3 Outro. mo17abreu@gmail.com. Co-autor 4 Aluno de pós-graduação. feaguirre90@gmail.com. Co-autor 5 Docente. rosmari.ufsm@gmail.com. Co-orientador. Anais do 9º SALÃO INTERNACIONAL DE ENSINO, PESQUISA E EXTENSÃO - SIEPE.
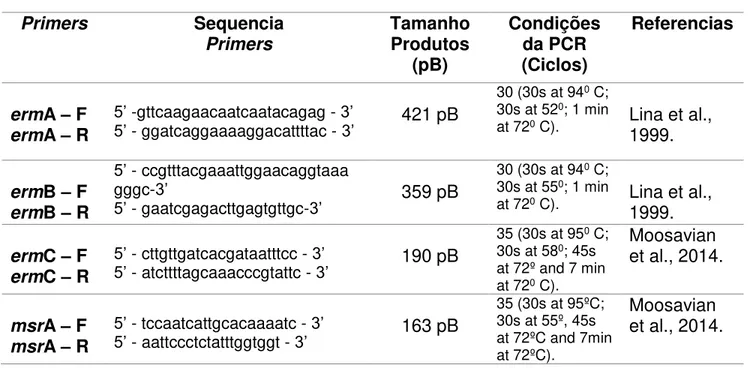
(3) CARACTERIZAÇÃO MOLECULAR DA RESISTÊNCIA À CLINDAMICINA EM ISOLADOS CLÍNICOS DE STAPHYLOCOCCUS AUREUS 1. INTRODUÇÃO A resistência do Staphylococcus aureus à classe dos macrolídeos, lincosamidas e estreptograminas do tipo B, caracteriza a chamada resistência MLSB. Sua expressão pode ser constitutiva (cMLSB) ou induzível (iMLSB) e é codificada principalmente pela presença do genes erm (erytromycin robossomal methylase) (MAHESH et al., 2013). No fenótipo cMLSB, os genes erm estão expressos e a RNA metilase é produzida mesmo na ausência de um agente indutor. Microrganismos com este fenótipo demonstram, in vitro, resistência à eritromicina, clindamicina e aos demais antimicrobianos MLSB, sendo facilmente detectada no antibiograma. Já no fenótipo iMLSB, a bactéria produz RNAm inativo, incapaz de codificar metilases. Este, tornase ativo, apenas na presença de um agente indutor como um macrolídeo, que leva a rearranjos do RNAm, permitindo que os ribossomas traduzam a sequência codificadora de metilases e o microrganismo expresse seu verdadeiro mecanismo de resistência. O tratamento das infecções com esse mecanismo, é um desafio, uma vez que podem ocorrer falhas terapêuticas (ALEKSANDRA et al., 2014; CHAUDHARY et al., 2015). Estudos de vigilância local relacionados à resistência MLSB podem contribuir para o monitoramento do uso de antimicrobianos, possibilitando a escolha da terapia empírica mais efetiva ao paciente, além de auxiliar na implementação de medidas para o controle de infecções no ambiente hospitalar. Dessa forma, este estudo investigou molecularmente a presença dos genes erm e msrA em isolados clínicos de S. aureus, obtidos de pacientes admitidos em um Hospital Terciário do sul do Brasil no período compreendido entre abril a dezembro de 2011, armazenados na bacterioteca do Departamento de Análises Clínicas e Toxicológicas da Universidade Federal de Santa Maria, os quais apresentaram no teste fenotípico (D-teste) previamente realizado (BOTTEGA et al., 2014), resistência a classe MLSB. 2. METODOLOGIA A utilização das amostras para pesquisa clínica, foi aprovada pelo Comitê de Ética e Pesquisa (CEP) da Universidade Federal de Santa Maria (UFSM), sob o número: 0117.0.243.000-08. Dos 140 isolados de S. aureus obtidos no período, foram selecionadas para realização da pesquisa molecular, apenas as 36 cepas que expressaram no teste fenotípico resistência aos MLSB. Destes, 25 apresentaram expressão constitutiva e 11 a induzível. A caracterização molecular dos genes de interesse (ermA, ermB, ermC e msrA), foi realizada através da técnica de reação em cadeia da polimerase (PCR). Os pares de primers utilizados, as condições de ciclagem e as referências utilizadas, encontram-se descritas na Tabela 1..
(4) Tabela 1: Primers usados na execução da PCR. Sequencia Primers. Primers. ermA ± F ermA ± R ermB ± F ermB ± R. ¶ -gttcaagaacaatcaatacagag - ¶ ¶ - ggatcaggaaaaggacattttac - ¶ ¶ - ccgtttacgaaattggaacaggtaaa gggc- ¶ ¶ - gaatcgagacttgagtgttgc- ¶. Tamanho Produtos (pB). Condições da PCR (Ciclos). 421 pB. 30 (30s at 940 C; 30s at 520; 1 min at 720 C).. 359 pB. 30 (30s at 940 C; 30s at 550; 1 min at 720 C).. ermC ± F ermC ± R. ¶ - cttgttgatcacgataatttcc - ¶ ¶ - atcttttagcaaacccgtattc - ¶. 190 pB. msrA ± F msrA ± R. ¶ - tccaatcattgcacaaaatc - ¶ ¶ - aattccctctatttggtggt - ¶. 163 pB. Referencias. 35 (30s at 950 C; 30s at 580; 45s at 72º and 7 min at 720 C). 35 (30s at 95ºC; 30s at 55º, 45s at 72ºC and 7min at 72ºC).. Lina et al., 1999. Lina et al., 1999. Moosavian et al., 2014. Moosavian et al., 2014.. 3. RESULTADOS e DISCUSSÃO O resultado da pesquisa dos genes que conferem resistência MLS B (erm e msrA) nos 36 isolados de S. aureus analisados, encontram-se descritos na Tabela 2. Tabela 2: Genes de resistência encontrados nos 36 isolados de S. aureus fenótipo MLSB. MLSB CONSTITUTIVO MLSB INDUZIVEL (n=11) (n=25) Genes MRSA MSSA MRSA MSSA Total de Pesquisados (n= 20) (n=5) (n= 3) (n=8) Amplificações ermA ermB ermC msrA ermA + ermB ermA + ermC ermA + msrA ermB + msrA ermC + msrA Negativo genes pesquisados TOTAL. 2 (10,0%) 1 (5,0%) 4 (20,0%) 3 (15,0%) 1 (5,0%) 3 (15,0%) 2 (10,0%) 4 (20,0%). 1 (20,0%) 1 (20,0%) 2 (40,0%) 1 (20,0%). 1 (33,34%) 1 (33,33%) 1 (33,33%) -. 1 (12,5%) 2 (25,0%) 1 (12,5%) 4 (50,0%) -. 4 2 8 5 1 3 3 1 4 5. 20 (100%). 5 (100%). 3 (100%). 8 (100%). 36 (100%). Considerando as associações onde houve amplificação para dois genes de resistência concomitantemente, pode-se afirmar que das 20 cepas com fenótipo cMLSB, resistentes à meticilina (MRSA), 8(40%) apresentaram gene ermA e 7(35%) ermC. A amplificação do ermB ocorreu em apenas 2(10%) dos isolados com este fenótipo de expressão. De acordo com Chavez et al. (2005), a associação entre.
(5) genes erm (ermA, ermB e ermC), assim como a associação destes com o msrA pode ocorrer, uma vez que um microrganismo pode apresentar mais de um gene de resistência. Essa informação corroborou com nossos resultados, onde observamos nas 25 cepas MRSA com expressão constitutiva, que 1(5,0%) isolado apresentou concomitantemente ermA e ermB e 3(15,0%) ermA e ermC. A associação entre ermA e msrA, também foi constatada em 2(10%) dos 20 isolados MRSA com este fenótipo de expressão. A associação entre genes erm e msrA em aureus, também foi relatada por Aleksandra et al. (2014) na Sérvia, em 5% dos 20 isolados analisados, e por Goudarzi et al.(2016) no Iran, em 17% dos 51 isolados estudados. Já nas 5 cepas cMLSB, sensíveis à meticilina (MSSA), não ocorreu amplificação do ermA, entretanto, ermB (1(20,0%)) e ermC (1(20,0%)), foram encontrados em isolados distintos. Dos 25 isolados cMLSB, 5(20%) não amplificaram para nenhum dos genes pesquisados. Possivelmente, este fato tenha ocorrido devido à presença de outros genes erm de resistência aos MLSB (ermD, ermF, ermY), não pesquisados neste estudo. Este fato também ocorreu no estudo de Pereira et al., (2016), em Pernambuco, onde ao submeterem 44 cepas de S. aureus a PCR, 22(50%) isolados não amplificaram para os genes erm pesquisados pelos autores. Das 11 cepas com fenótipo induzível, 3 eram MRSA e 8 MSSA. A presença do gene ermA foi evidenciada em 2(66,7%) dos isolados MRSA, sendo que destes, 1 (33,33%) apresentava tanto ermA como msrA. Não ocorreu amplificação para o ermB, enquanto o ermC amplificou em apenas 1(33,3%) dos isolados com este mecanismo de resistência. Nas 8 cepas iMLSB, sensíveis à meticilina (MSSA), ermA foi encontrado em apenas 1(12,5%) dos isolados. O gene ermC amplificou isoladamente em 2(25,0%) isolados enquanto 4(50,0%) carreavam tanto o ermC como o msrA. Já o gene ermB foi identificado em 1(12,5%) dos isolados com este fenótipo de expressão, em associação ao gene msrA. Entre os genes erm pesquisados em nosso estudo, ermC e ermA foram os prevalentes, sendo que das 36 cepas, 15(41,7%) e 11(30,5%) apresentavam estes genes, respectivamente. No estudo de Saderi et al. (2011) com 126 isolados de aureus obtidos de pacientes atendidos em 4 hospitais universitários de Teerã, os pesquisadores também encontraram prevalência desses genes, sendo que ermA e ermC amplificaram em 15(11,9%) e 8(6,4%) isolados, respectivamente. Além disso, esses autores relataram que 61(48,4%) dos isolados analisados, apresentaram concomitantemente, ambos os genes. De acordo com Fiebelkorn et al., 2003 e Vallianou et al., 2014, os determinantes ermA e ermC são os predominantes nos estafilococos. Os genes ermA são mais difundidos em MRSA por serem transportados por transposons relacionados com TN554, que codifica resistência à eritromicina e à estreptomicina, enquanto que os genes ermC são responsáveis principalmente pela resistência à eritromicina em MSSA e são transportados por plasmídeos. O gene ermB amplificou em 4(11,1%) das 36 amostras desse estudo. A relativa baixa frequência do ermB, pode ter ocorrido pelo fato deste gene estar prevalentemente associado a Staphylococcus spp., de procedência animal (Díaz et al., 2007). No estudo de Coutinho et al. (2010), a prevalência desse gene, também foi baixa, sendo que dos 152 Staphylococcus spp. analisados, apenas 3(1.9%) amplificaram para ermB. Entretanto, no estudo de Chavez et al.(2005), no Texas, com 67 isolados de aureus de origem pediátrica, 31(46,3%) carreavam este gene, presença significativamente maior da encontrada em nosso estudo..
(6) Quanto a pesquisa do gene msrA, a presença do gene foi encontrada em 13(36,1%) das 36 cepas analisadas. Destas, 7 apresentavam fenótipo cMLS B e 6 fenótipo iMLSB. Nas amostras com expressão constitutiva, 5 apresentaram este gene isoladamente, sendo 3(15%) MRSA e 2(40%) MSSA. Os outros 2(10%) isolados, carreavam tanto msrA como ermA. Já nas cepas induzíveis, essa expressão aconteceu associada aos genes ermB e ermC presentes em 1(12,5%) e 4(50%) dos isolados respectivamente. No estudo de Zmantar e colaboradores (2008) na Tunísia com 35 S. aureus, a presença do gene msrA foi encontrada em 10(28%) dos isolados pesquisados, assemelhando-se aos resultados obtidos em nosso estudo. Já no de Cetin et al. (2010), dos 47 S. aureus analisados, 13(27%) carreavam o gene msrA, sendo que desses, 6 estavam associados aos genes erm. Diferenças na caracterização molecular no que se refere à prevalência dos genes erm (ermA, ermB e ermC) e msrA com demais estudos da literatura (COUTINHO et al., 2008; SADERI et al., 2011; SEIFI et al., 2012), podem ser explicadas, já que estas taxas podem variar entre diferentes hospitais conforme a região geográfica, o que salienta a importância da determinação dessas frequências em um local específico. 4. CONSIDERAÇÕES FINAIS Esses resultados reforçam a idéia de que existem diferenças geográficas na prevalência de mecanismos de resistência aos MLS B entre os Staphylococcus aureus, e que a realização de estudos de vigilância locais podem ser uma ferramenta importante para guiar a terapêutica do paciente e promover o uso racional de antimicrobianos. 5. REFERÊNCIAS ALEKSANDRA, A. D.; MISIC, M. S.; MIRA, Z. V. et al. Prevalence of inducible clindamycin resistance among community-associated staphylococcal isolates in central Serbia. Indian Journal of Medical Microbiology, v. 32, p.49-52, 2014. BOTTEGA, A.; RODRIGUES, M. A.; CARVALHO, F. A. et al. Evaluation of constitutive and inducible resistance to clindamycin in clinical samples of Staphylococcus aureus from a tertiary hospital. Revista da Sociedade Brasileira de Medicina Tropical, v. 47, p. 589-592, 2014. CETIN, E. S.; GUNES, H.; KAYA, S. et al. Distribution of genes encoding resistance to macrolides, lincosamides and streptogramins among clinical Staphylococcal isolates in a Turkish university hospital. Journal of Microbiology, Immunology and Infection, v. 43, p. 524-529, 2010. CHAUDHARY, A. B.; SHAH, K. K.; PARMAR, R. J. et al. Inducible clindamycin resistance in Staphylococcus aureus isolated from clinical samples. International Journal of Applied & Basic Medical Research, v. 9, p. 922-924, 2015. CHAVEZ, B. S.; BOZDOGAN, B.; KATZ, K. et al. Inducible clindamycin resistance and molecular epidemiologic trends of pediatric community-acquired methicillinresistant Staphylococcus aureus in Dallas, Texas. Antimicrobial Agents and Chemotherapy, v. 49, p. 2283-2288, 2005..
(7) COUTINHO, V.L.; PAIVA, R.M.; REITER, K.C, et al. Distribution of erm genes and low prevalence of inducible resistance to clindamycin among staphylococci isolates. The Brazilian of Journal Infectious Diseases, v. 14, p. 564-568, 2010. DÍAZ, L. M.; LA CASA, A. C.; SÁNCHEZ, M. J. T. et al. Detección de resistência inducible a clindamicina en aislados cutáneos de Staphylococcus spp. por métodos fenotípicos y genotípicos. Enfermedades Infecciosas y Microbiología Clínica, v. 25, p. 77-81, 2007. FIEBELKORN, K. R.; CRAWFORD, S. A.; MCELMEEL, M. L. et al. Practical disk diffusion method for detection of inducible clindamycin resistance in Staphylococcus aureus and coagulase-negative staphylococci. Journal of Clinical Microbiology, v. 41, p. 4740-4744, 2003. GOURDARZI, G.; TAHMASBI, F.; ANBARI, K. Distribution of Genes Encoding Resistance to Macrolides Among Staphylococci Isolated From the Nasal Cavity of Hospital Employees in Khorramabad, Iran. Iranian Red Crescent Medical Journal, v.18, p.1-8, 2016. LINA, G.; QUAGLIA, A.; REVERDY, M. E. et al. Distribution of genes encoding resistance to macrolides, lincosamides, and streptogramins among staphylococci. Antimicrobial Agents and Chemotherapy, v. 43, p.1062-1066, 1999. MAHESH, C. B.; KULKARNI, R. B.; SATARADDI, J. The prevalence of inducible and constitutive clindamycin resistance among the nasal isolates of staphylococci. Journal of Clinical and Diagnostic Research, v. 7, p. 1620, 2013. MOOSAVIAN, M.; SHOJA, S.; ROSTAMI, S. et al. Inducible clindamycin resistance in clinical isolates of Staphylococcus aureus due to erm genes, Iran. Iranian Journal of Microbiology, v. 6, p. 421-427, 2014. PEREIRA, J. N.; RABELO, M. A.; LIMA, J. L. et al. Phenotypic and molecular characterization of resistance to macrolides, lincosamides and type B streptogramin of clinical isolates of Staphylococcus spp. of a university hospital in Recife, Pernambuco, Brazil. Brazilian Journal Infectious Diseases, v. 20, p. 276-281, 2016. SADERI, H.; EMADI, B.; OWLIA, P. Phenotypic and genotypic study of macrolide, lincosamide and streptogramin B (MLSB) resistance in clinical isolates of Staphylococcus aureus in Tehran, Iran. Medical Science Monitor, v.17,p.48-53, 2011. VALLIANOU, N.; EVANGELOPOULOS, A.; HADJISOTERIOU, M. et al. Prevalence of macrolide, lincosamide, and streptogramin resistance among staphylococci in a tertiary care hospital in Athens, Greece. Journal of Chemotherapy, v. 27, p. 319-323, 2014. ZMANTAR, T.; CHAIEB, K.; BEN ABDALLAH, F. et al. Multiplex PCR detection of the antibiotic resistance genes in Staphylococcus aureus strains isolated from auricular infections. Folia Microbiologica, v. 53, p. 357-362, 2008..
(8)
Figure
Documento similar
A gravidade da emergência causada pela pandemia do coronavírus (COVID-19) exige das autoridades brasileiras, em todos os níveis de governo, a efetivação concreta da proteção
No que tange a educação em saúde enquanto prática de promoção da saúde todos os estudos analisados referem que o ACS desempenha papel fundamental, em especial à
O entendimento do termo Infecções Hospitalares (IH) vem sendo substituído nos últimos anos pelo Ministério da Saúde e Agência Nacional de Vigilância Sanitária pelo termo
Portadores asintomáticos de Staphylococcus aureus meticilino resistentes (MRSA) en pescadores y horticultores de Guerrero, México Jeiry Toribio Jiménez, Yad Ciri Ojendiz Mata,
TRATAMIENTO DE LAS LESIONES DEL PIE INFECTADAS POR STAPHYLOCOCCUS AUREUS RESISTENTES A METICILINA (SARM) EN PACIENTES CON DIABETES.. AUTOR: Luchoro Pérez, Marga Nº
A nossa abordagem se coloca ao lado da diretriz interpretativa indígena, isto é, um esforço para trazer à cena a resistência guaranítica contra o massacre dirigido pelas
Referente à primeira pergunta da entrevista, pode-se dizer que houve uma preponderância de concepções associadas ao que há de mais recente em termos de teoria sobre
Com o exercício da transcrição, foi possível redesenhar uma nova série de caminhos, que partindo das memórias da comunidade, ofereciam a oportunidade à equipa do projeto