1
Evaluación preliminar de la diversidad genética de morfotipos de ibia (Oxalis
tuberosa Mol.) en municipios de Ventaquemada y Turmequé (departamento de
Boyacá) por medio de marcadores ISSR
Lucia Carolina Uribe Montes
TRABAJO DE GRADO
Presentado como requisito parcial para optar al título de
Bióloga
PONTIFICIA UNIVERSIDAD JAVERIANA
FACULTAD DE CIENCIAS
DEPARTAMENTO DE BIOLOGÍA
2
Evaluación preliminar de la diversidad genética de morfotipos de ibia (Oxalis tuberosa
Mol.) en municipios de Ventaquemada y Turmequé (departamento de Boyacá) por medio de marcadores ISSR
Lucia Carolina Uribe Montes
Directora
María del Pilar Márquez Cardona M.Sc.
Pontificia Universidad Javeriana
Facultad de Ciencias
Departamento de Biología
3
Evaluación preliminar de la diversidad genética de morfotipos de ibia (Oxalis tuberosa
Mol.) en municipios de Ventaquemada y Turmequé (departamento de Boyacá) por medio de marcadores ISSR
Lucia Carolina Uribe Montes
APROBADO
4
Evaluación preliminar de la diversidad genética de morfotipos de ibia (Oxalis tuberosa
Mol.) en municipios de Ventaquemada y Turmequé (departamento de Boyacá) por medio de marcadores ISSR
Lucia Carolina Uribe Montes
APROBADO
_________________________
María del Pilar Márquez M.Sc
Directora
________________________
Neidy Clavijo Ponce M.Sc
5
NOTA DE ADVERTENCIA
Artículo 23 de la Resolución N° 13 Julio de 1946
6
Agradecimientos
A mis padres y mi hermana por el inmenso amor que me brindan cada minuto del día y por hacerme feliz.
A mi directora de trabajo de grado María del Pilar Márquez por su confianza depositada en mí, su ayuda, enseñanzas y su compañía durante este proceso.
A Neidy Clavijo por ser un ejemplo para mí, a través de su trabajo en el campo con las comunidades y por sus correcciones y enseñanzas en salida de campo.
A los agricultores de los municipios de Turmequé y Ventaquemada por abrirnos las puertas de sus hogares y compartir su sabiduría con nosotros.
A mis amigos y compañeros del laboratorio de Biologia Molecular Vegetal por su gran colaboración, amabilidad, y compañía.
7
Tabla de contenido
Resumen ... 8
Introducción ... 8
Justificación y planteamiento del problema ... 10
Marco teórico ... 12
Los tubérculos andinos... 12
Pérdida de diversidad genética y estrategias de conservación ... 13
Oxalis tuberosa Molina ... 15
El estudio de la diversidad genética ... 17
a. Cómo se estudia la diversidad genética ... 17
b. PCR ... 18
c. Marcadores ISSR ... 19
d. Extracción de ADN ... 20
e. Estudios de diversidad de ibia por medio de ISSR ... 22
Pregunta de investigación ... 24
Objetivos ... 24
Objetivo general ... 24
Objetivos específicos ... 24
Metodología ... 24
Fase de campo: ... 25
Localización y material vegetal ... 25
Fase de laboratorio ... 27
Extracción de ADN ... 27
Métodos de análisis del ADN ... 29
PCR ... 29
Análisis de Diversidad Genética ... 31
Resultados ... 32
Extracción de ADN ... 32
Condiciones de PCR ... 34
Polimorfismo ... 35
Evaluación de la diversidad ... 36
Discusión ... 39
Extracción de ADN ... 39
8
Evaluación de la diversidad de muestras de ibia ... 42
Conclusiones ... 46
Recomendaciones ... 47
Bibliografía ... 48
Anexo 1 formato de datos para colecta ... 52
Anexo 2. Ejemplo de formato de etiquetas para herbario ... 53
Anexo 3. Lista de accesiones incluidas en estudio de diversidad. ... 54
Anexo 4 Individuos de Oxalis tuberosa utilizados para pruebas de extracción ... 55
Anexo 5. Gráficos de medias para parámetros evaluados durante la extracción de ADN ... 56
Anexo 6 Prueba de amplificación con primers RAM ... 57
Anexo 7. Protocolo y concentraciones para primers RAM ... 58
Anexo 8. Evidencia para condiciones adecuadas de PCR ... 60
Anexo 9. Protocolo Pissard ... 61
Anexo 10 Matriz binaria ... 62
Anexo 12.Tabla de colores de tallos ... 69
Anexo 13 Extracción de 29 muestras de ibia por medio de protocolo CTAB1 ... 70
Resumen
La región andina ha sido un centro de domesticación de cultivos entre los que se encuentran tres especies de tubérculos andinos consideradas especies infrautilizadas o marginadas. Las especies conocidas en Colombia como Ruba, Cubio e Ibia se encuentran fuertemente ligadas a prácticas de manejo de cultivos y usos tradicionales, que conforman una identidad en las comunidades de indígenas y pequeños productores. En estas especies se ha reconocido un posible proceso de erosión genética, lo que indica la necesidad de caracterizar la variabilidad genética en regiones consideradas microcentros de diversidad, lo cual permite establecer estrategias de conservación. Uno de los posibles microcentros de diversidad en Colombia corresponde a los municipios de Ventaquemada y Turmequé en el departamento de Boyacá. Con base en caracterizaciones previas de los sistemas productivos de estos cultivos en la región y la determinación de algunos morfotipos, se realizó una caracterización molecular de la variabilidad genética intraespecífica de la especie Oxalis tuberosa M. (ibia). Para llevar a
9
la extracción resulta exitosa para las muestras de ibia con un protocolo reportado en estudios previos de O. tuberosa. En cuanto a las condiciones de PCR se determinó que el patrón de
bandeo más claro resulta de aumentar las concentraciones de los reactivos reportadas en trabajos previos de diversidad de tubérculos andinos. El estudio de diversidad exhibe que en general las muestras se agrupan según su morfotipo, sin embargo se hace necesario corroborar esta evaluación preliminar con datos que permitan obtener análisis más robustos.
Introducción
Considerando que el número de alimentos que conforman la dieta básica de la población mundial se restringe principalmente a maíz, arroz, trigo y papa, es fundamental indagar en las posibilidades de diversificación de la dieta. Esta vulnerabilidad actual del sistema alimentario mundial exige emprender esfuerzos por superarla, tales como la investigación en torno a cultivos que se han denominado marginados o infrautilizados (Galluzzi et al.,
2013). La contribución de estas investigaciones busca comprender el potencial de estas especies y su valor cultural, nutricional y económico.
Entre estos cultivos marginados se encuentran tres especies de tubérculos andinos Ruba (Ullucus tuberosus Caldas), Cubio (Tropaeolum tuberosum Ruiz y Pavón) e Ibia (Oxalis tuberosa Molina) que han hecho parte de la dieta de comunidades indígenas y campesinas de
los países andinos por cientos de años. Hasta el momento se han adelantado pocos estudios relacionados con estos cultivos, los cuales provienen principalmente de entidades nacionales e internacionales en Perú, Ecuador y Bolivia. En el caso de Colombia existen algunas investigaciones relacionadas con etnobotánica, estudio de las prácticas de manejo, fisiología vegetal y características morfológicas de los tubérculos. Sin embargo estos estudios no han tenido una alta divulgación e impacto, a pesar de que son un punto de partida para reconocer la importancia nutricional, cultural y otros usos potenciales de estas especies (Clavijo et al.,
2014).
10
En el presente trabajo se implementan los marcadores moleculares denominados ISSR (Inter- secuencias simples repetidas) para evaluar la diversidad intra específica en las ibias (Oxalis tuberosa). Para efectuar el estudio se realizó un muestreo en fincas de pequeños agricultores
en los municipios de Turmequé y Ventaquemada (Boyacá), colectando material vegetal de diferentes morfotipos identificados por los agricultores con base en las características externas del tubérculo (color). Los estudios previos en la zona señalan la existencia de 5 morfotipos para esta región que corresponden a ibias amarillas, blancas, rosadas, rojas y amarillas con rayas rojas (Clavijo et al., 2014).
Junto con un estudio de diversidad de la especie Tropaeolum tuberosum (Cubio) desarrollado
a la par, la presente investigación de variabilidad intraespecífica con la ibia se enmarca dentro de un macro-proyecto interdisciplinar denominado “Memoria histórica, caracterización genética y aprovechamiento alimentario de tubérculos andinos marginados en las provincias de Centro y Márquez (Boyacá)”. Este busca realizar caracterizaciones de la zona y de los cultivos existentes desde tres enfoques, que además de la caracterización genética incluyen una caracterización bromatológica y nutricional y finalmente una enfoque histórico, que estudia los cambios sociales, culturales y agroecológicos de los agroecosistemas y su entorno a través del tiempo.
El estudio de la diversidad se lleva a cabo por medio de marcadores ISSR y busca evaluar la posibilidad de considerar estos morfotipos como variedades genéticas. Para lograr un resultado final es necesario procesar las muestras colectadas en el laboratorio y someterlas a un proceso previo de extracción de ADN y hallar condiciones adecuadas de la técnica de PCR. A continuación se presentan los resultados de este proceso, que significarán un aporte hacia la comprensión de las dinámicas y la importancia de estos cultivos, buscando lograr el uso sostenible y la conservación de la agrobiodiversidad1 de nuestro país.
Justificación y planteamiento del problema
Por miles de años los países andinos han sido centros fundamentales de diversidad y domesticación de cultivos que han desarrollado adaptaciones para estas regiones específicas. De los cultivos domesticados existen tres especies de tubérculos de diferentes familias
11
botánicas poco conocidas internacional e incluso localmente, que guardan similitudes morfológicas entre ellos: la ruba, el cubio y la ibia (Clavijo, 2014).
Estas especies han sido consideradas especies marginadas o infrautilizadas por asociarse a prácticas de manejo tradicionales, condiciones agroecológicas poco favorables y baja competitividad en el mercado, distribuyéndose principalmente en mercados locales (Galluzzi
et al, 2013). Sin embargo especies marginadas como los tubérculos andinos tienen una gran
importancia en la búsqueda de la diversificación de la dieta global.
Estos recursos se han mantenido hasta la actualidad gracias a comunidades indígenas y campesinas, sin embargo, la pérdida progresiva de conocimiento tradicional y la presión del mercado ha ocasionado un proceso de erosión genética, generando la disminución de recursos que tienen un uso y valor potencial que aún no se ha estudiado ampliamente. Esto se encuentra ligado a la pérdida de prácticas de manejo de cultivos y usos tradicionales que forjan la identidad de las comunidades andinas (FAO, 2012; Galluzzi et al., 2013; Clavijo et al., 2014;).
Para evitar esta pérdida y establecer métodos de conservación apropiados para los cultivos, es necesario el estudio de la diversidad genética en zonas estratégicas. Hasta el momento estos estudios de diversidad en Colombia han sido muy reducidos, reportándose algunos esfuerzos de caracterización de los sistemas de producción en el departamento de Nariño (Rosero, 2010) y Boyacá (Clavijo et al., 2011; Aguirre et al., 2012).
Con el fin de construir estrategias de conservación se debe tener en cuenta que la distribución de la variabilidad se da en forma desigual, concentrándose en zonas denominadas “microcentros”, los cuales cuentan con condiciones favorables para el establecimiento de estos cultivos (García & Cadima, 2003). Al respecto Clavijo y Pérez (2014) indican la posible existencia de un microcentro de diversidad en los municipios de Turmequé y Ventaquemada (Boyacá), identificando presencia de ibias, cubios y rubas a diferentes escalas de siembra en la región. Estos cultivos representan la base principal de la alimentación familiar (Clavijo & Pérez, 2014).
12
2014). Para contribuir al reconocimiento de los posibles efectos de la erosión genética en estos cultivos y establecer la posibilidad de reconocer esta región como un microcentro de diversidad, la caracterización de la variabilidad genética de las diferentes especies es indispensable (FAO, 2010).
La ibia, Oxalis tuberosa M. es una de las especies de tubérculos cultivados en esta región
de la cual se han identificado hasta el momento en las fincas de los municipios Turmequé y Ventaquemada, cinco morfotipos (rosados, amarillos, rojos, blancos y amarillos con rayas rojas) (Clavijo, 2014). En el presente estudio se plantea como objetivo general, la evaluación molecular de la diversidad existente entre estos morfotipos.
La caracterización molecular debe incluir una fase inicial de estandarización para lograr finalmente evaluar la diversidad entre la totalidad de las muestras por medio de los marcadores moleculares ISSR (Weising et al., 2005). Se espera contribuir al conocimiento
de los patrones de diversidad de esta especie, que puedan incluso servir como modelos de otras especies con condiciones agroecológicas similares y lograr un aprovechamiento sostenible de la biodiversidad que ofrecen los suelos colombianos.
Marco teórico
Los tubérculos andinos
Las rubas, cubios e ibias han evolucionado en ambientes inusuales que se caracterizan por alturas entre 2400 y 4000 msnm, climas fríos y niveles de precipitación anuales entre los rangos de 700 – 800 mm para rubas, 700-1600mm para los cubios y 570-2500mm para las ibias. Estas especies de tubérculos poseen una alta tolerancia a condiciones de erosión, fluctuaciones en temperaturas y lluvias, suelos empobrecidos y alta radiación UV (Flores et al., 2003).
13
La investigación alrededor de estos cultivos se empieza a desarrollar a partir de 1979 (Clavijo
et al., 2004). Los estudios se han establecido en torno a temas de conservación, diversidad
genética y sistemas de producción (Malice & Baudoin, 2009). Estos se han desarrollado generalmente en Ecuador, Bolivia y Perú, donde ha habido mayor disponibilidad de recursos para colecta, conservación y utilización de estas plantas productoras de tubérculos, ya que históricamente se ha considerado que estos países poseen la mayor diversidad de especies (Rosero, 2010).
En Colombia la producción de tubérculos se ubica en los departamentos de Nariño, Cauca, Cundinamarca y Boyacá. Estos cultivos han complementado la dieta de comunidades indígenas y campesinas en ecosistemas altoandinos y paramunos de Colombia desde la época prehispánica hasta la actualidad (Pérez, 2009; Rosero, 2010).
Estas especies tienen una importancia fundamental en la seguridad alimentaria ya que ofrecen una alternativa para diversificar la dieta en la población general y en particular en comunidades rurales e indígenas (Clavijo et al., 2011). Por esta razón es crucial evitar la
pérdida de sistemas de producción locales y a pequeña escala, lo cual puede lograrse a través de investigaciones enfocadas en disminuir eventos de “cuello de botella” y en sistemas de conservación que complementen los métodos in situ y ex situ (Galluzzi et al., 2013),
incrementando posibilidades de uso de los recursos genéticos de tubérculos de forma sostenible (García & Cadima, 2003).
Pérdida de diversidad genética y estrategias de conservación
Los tubérculos andinos han atravesado un proceso progresivo de deterioro que ha conducido a la pérdida de diversidad genética2 (Clavijo et al., 2011). Esta pérdida de diversidad, llamada
erosión genética se ha atribuido a factores asociados a presiones del mercado, dificultades en comercialización, pérdida de conocimiento tradicional o la asociación del producto a un alimento propio de poblaciones de bajos recursos. Adicionalmente contribuyen aspectos
14
como la degradación de la tierra o factores bióticos como enfermedades (García & Cadima, 2003; Malice & Baudoin, 2009).
Con el fin de conservar la diversidad genética existen dos métodos fundamentales conocidos como in situ y ex situ.
El enfoque in situ consiste en la conservación de poblaciones y ecosistemas en sus hábitats
de origen. Se busca que este tipo de conservación se constituya en zonas de alta diversidad genética de especies tanto cultivadas como silvestres, favoreciendo los procesos evolutivos naturales (García & Cadima, 2003). La influencia humana juega un papel fundamental en la conservación in situ, ya que el uso y manejo de estas especies puede alterar los patrones de
diversidad genética en estas (Rosero, 2010).
La variabilidad de las especies no se distribuye de igual forma en toda la franja andina, por lo que se debe priorizar que la conservación in situ se realice en zonas denominadas “microcentros” que cuenten con características ambientales y socioculturales favorables (García & Cadima, 2003).
Este enfoque beneficia también a agricultores y comunidades asociadas, constituyendo una base de seguridad alimentaria. De igual forma se debe considerar que forman parte del equilibrio de los ecosistemas, puede asociarse a la conservación de parientes silvestres y permite perpetuar procesos de evolución natural (García & Cadima, 2003).
La conservación ex situ consiste fundamentalmente en bancos de germoplasma que
mantienen muestras de poblaciones en lugares físicos con condiciones apropiadas para conservar los recursos genéticos. Si bien esto puede implicar que se obstaculicen los procesos evolutivos naturales, puede por otro lado permitir conservar la biodiversidad que se encuentra amenazada en condiciones naturales por diversos factores (García & Cadima, 2003).
Considerando tanto ventajas como desventajas de cada método o enfoque, las actuales estrategias de conservación buscan constituirse logrando complementar ex situ e in situ para
15
Oxalis tuberosa Molina
Oxalis tuberosa Molina. Pertenece a la familia botánica Oxalidaceae, orden Geraniales; sus
nombres comunes en español incluyen ibia (Colombia); oca, quiba, ciuba, ciuva, huisisai, (Otras partes de Sur América) y papa roja (México) (National Research Council, 1989).
Además de cultivarse en la región andina con mayor abundancia en Ecuador, Perú y Bolivia, donde, después de la papa, constituye el cultivo de tubérculo más importante (Flores et al.,
2003), la ibia ha sido cultivada en México y Nueva Zelanda (National Research Council, 1989). La domesticación de esta especie ocurrió probablemente en la región central de Perú y en el norte de Bolivia donde se presenta la mayor diversidad de formas cultivadas (Pissard
et al, 2006).
Generalmente la propagación de las especies de tubérculos andinos que se han mencionado, incluyendo la ibia, es vegetativa a través de los tubérculos (Malice & Baudoin, 2009). O. tuberosa es una hierba anual generalmente de 20 a 30 cm de alto con tallos cilíndricos cuya
variación de color puede ser de verde a rojo (King, 1987). Los tallos normalmente se alzan desde la base de la planta y son pubescentes. En días largos, los estolones crecen como tallos aéreos y en días cortos, penetran en el suelo formando tubérculos (National Research Council, 1989). Los tubérculos exhiben una amplia variación de color y tamaño y poseen niveles variables de ácido oxálico; las variedades con altos niveles se consideran amargas y se procesan de diversas formas (King, 1987).
Las hojas son alternas, trifoliadas, pinnadas o palmaticompuestas y pubescentes, los foliolos son de 1 a 4 cm con color verde oscuro en el haz y purpura o verde en el envés. Las inflorescencias son axilares con cuatro o cinco flores hermafroditas. El cáliz está conformado por cinco sépalos y la corola por cinco pétalos unidos en la base, de color amarillo o anaranjado con bordes trilobados y tres nervios principales de color negro. Los estambres se encuentran dispuestos en dos verticilos pentámeros de diferente longitud; cuentan con un ovario pentacarpelar con carpelos separados y cinco estilos libres; El fruto consiste en una capsula (dehiscencia explosiva) de cinco lóculos de pared membranosa, cada lóculo con una a tres semillas (Barrera et al, 2004). El ciclo de la planta puede durar entre 7 y 10 meses
16
Generalmente se afirma que es una especie octoploide 2n= 8x= 64, sin embargo Rosero (2010) indica que la naturaleza de la oca no es clara ya que diversas investigaciones presentan resultados inconsistentes (Rosero, 2010). Posee un sistema trimórfico de incompatibilidad genética lo que impide el autocruzamiento y conlleva a la polinización cruzada (Pissard et al, 2006).
O. tuberosa presenta gran variación en los niveles nutricionales y se puede considerar que
su valor nutricional es igual o mejor que la papa. En promedio contienen de 70 a 80% de humedad, 11-22% de carbohidratos y alrededor de 1% de grasa y fibra. En cuanto a los niveles de proteína se presenta una gran diferenciación entre variedades, algunos tubérculos con alta concentración de proteína pueden contener más del 9 % de su peso seco en proteína de alta calidad con un buen balance de aminoácidos esenciales (National Research Council , 1989) .
En un estudio realizado por Flores et al. (2002) se presenta una caracterización de la proteína
soluble más importante del tubérculo, denominada Ocatina (constituye del 40% al 60% de la proteína soluble total) y demostró que posee actividades antibacterianas y antifúngicas contra diversos microorganismos del suelo, por lo que consideran su función como una proteína de almacenamiento que puede desempeñar un papel en la resistencia natural a patógenos (Flores et al., 2002) También se ha determinado que el tubérculo posee un
potencial para producción de almidón, alcohol y alimento de ganado (National Research Council , 1989).
El germoplasma de ibiaex situ se ha distribuido en diversos bancos de germoplasma en
Suramérica, ubicados principalmente en Perú con el mayor número de accesiones (1696) en el instituto nacional de investigación agropecuaria (INIA) (Flores et al., 2003).
La caracterización y diversidad de Oxalis tuberosa no ha sido suficientemente explorada en
Colombia y no se registran bancos de germoplasma que conserven una muestra representativa (Rosero, 2010). Los estudios realizados parecen reflejar una menor importancia (en cuanto a producción) de este cultivo con respecto a Tropaeolum tuberosus
y Ullucus tuberosus (a diferencia de la tendencia que se presenta en países como Perú y
17
El estudio de la diversidad genética
a. Cómo se estudia la diversidad genética
Los organismos se diferencian por variaciones en las secuencias en el ADN y por alteraciones ocasionadas por el ambiente. El estudio de estas diferencias es fundamental para establecer estrategias de conservación y esto se ha realizado a través del desarrollo de herramientas moleculares que han permitido, entre otras utilidades, la identificación de polimorfismos causantes de la variación genética (FAO, 2010).
Los denominados marcadores genéticos permiten indicar el genotipo de un individuo y de uno o varios loci relacionados con el marcador, proporcionando herramientas para la identificación de plantas y el fitomejoramiento (Henry, 2013)
Para determinar un marcador genético ideal se toma en consideración que este sea polimórfico (con capacidad de revelar variaciones genéticas individuales), multialélico, codominante (haciendo posible distinción entre homocigotos y heterocigotos), no epistáticos (independencia entre locus), neutral (lo que implica que las sustituciones de alelos en el locus del marcador no inciden en el fenotipo o proceso de selección) y que no sean susceptibles a cambios ambientales. Los marcadores que mejor aplican en la actualidad para estas condiciones han sido los marcadores de ADN que se han impuesto ante los marcadores morfológicos o bioquímicos (Vienne, 2003). Esto se debe a que los métodos basados en ADN permiten un análisis directo en el genoma en lugar de inferencias basadas en el análisis de la expresión de los genes (Henry, 2013). De igual forma, estos análisis son independientes del estado de desarrollo o del tejido analizado y son casi infinitos en número, permitiendo aplicaciones adicionales en biología molecular (Vienne, 2003).
Los marcadores de ADN han sido utilizados ampliamente en análisis de diversidad, caracterización y mapeo genético (Meena et al., 2015). Los primeros métodos desarrollados
18
Esta técnica fue ampliamente utilizada hasta la aparición de métodos basados en la reacción en cadena de la polimerasa o PCR (Joshi et al., 1999). Además de requerir menores
cantidades de ADN, la implementación de los métodos basados en PCR aporta una mayor sensibilidad, especificidad, poder de resolución y facilidad en la ejecución del procedimiento, lo que se traduce en una mayor confiabilidad de los datos obtenidos en los análisis (Henry, 2013).
b. PCR
La técnica de PCR desarrollada por Mullis y colaboradores en 1983 permite la amplificación
in vitro de secuencias de ADN de interés. Desde el momento en el que hubo disponibilidad
de la enzima polimerasa termoestable, esta técnica ha reemplazado ampliamente previas metodologías de la biología molecular por su mayor sensibilidad y conveniencia (Weising
et al., 2005). La reacción en cadena de la polimerasa se basa en la acción de dos primers
(secuencias de oligonucleótidos o cadenas cortas de ADN o ARN), cada uno complementario a ambas cadenas opuestas de una sección de ADN. La síntesis de cada nueva cadena debe direccionarse de tal forma que su extensión se dirija una hacia la otra obteniendo como producto final la síntesis de una región específica limitada por los dos primers (Mcpherson et al., 1996).
Para que se lleve a cabo la reacción de amplificación debe prepararse un mix que contenga: una polimerasa termoestable que adicionará nucleótidos a partir del extremo 3´de los
primers; un buffer que generalmente contiene Tris-HCl, KCl y MgCl; deoxinucleótidos o
dNTPs (dATP, dCTP, dGTP, dTTP); Primers y el ADN que se pretende amplificar. Durante
el primer ciclo, en una primera fase a 94°C, ocurre la denaturación, permitiendo la separación de la doble cadena de ADN. Posteriormente ocurre una disminución de la temperatura con el fin de que los primers se hibriden a las secuencias blanco en la cadena; la siguiente etapa
19
Con el fin de optimizar las condiciones para la amplificación es posible modificar ciertos aspectos del proceso de PCR. Determinar las concentraciones apropiadas para reactivos como magnesio, dNTPs o la polimerasa puede significar una mejora en los resultados del proceso. Generalmente la optimización inicia con un ensayo de diferentes concentraciones de magnesio; las concentraciones óptimas reportadas varían entre 1mM a 6 mM. El magnesio es un cofactor requerido de la taq polimerasa y las variaciones en este pueden afectar el funcionamiento de la enzima. Debido a que los dNTPs corresponden a las unidades de construcción para los amplicones de PCR y que se requieren también como cofactor de la polimerasa, las variaciones en su concentración deben optimizarse para lograr un resultado exitoso. De igual forma las concentraciones y la calidad de los primers afectarán la
amplificación. Las modificaciones en el protocolo como tal consisten en cambios en temperaturas de hibridación o en los ciclos , esto puede aumentar la especificidad del producto y evitar la unión de primers en regiones inadecuadas (Kennedy & Oswald, 2011)
c. Marcadores ISSR
Los marcadores ISSR se encuentran dentro de los marcadores basados en la reacción en cadena de la polimerasa (PCR). Técnicas como ISSR, AFLP o RAPD son sistemas de marcadores dominantes y por lo tanto no permiten la detección de variabilidad en los alelos. Sin embargo, son fáciles de implementar e implican un menor costo (Vijayan, 2005), esto aplica especialmente para ISSR, por lo que se está implementando cada vez de forma más frecuente en estudios filogenéticos, de diversidad genética, mapeo genético, entre otros (Reddy et al., 2002).
La implementación de estos marcadores se basa en la amplificación de segmentos de ADN que se encuentran entre dos regiones idénticas repetidas de microsatélites (SSR) situadas en dirección opuesta (Vijayan, 2005). Los primers utilizados corresponden a microsatélites de
16 a 25 pares de bases de longitud, con repeticiones de di, tri, tetra o pentanucleotidos. Los
primers se unen a diferentes loci en el genoma y se genera la amplificación de las secuencias
intermedias entre los microsatélites de diferentes tamaños. Los primers pueden ser de dos
20
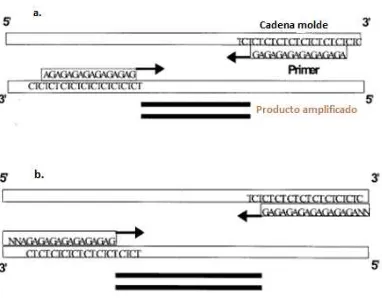
[image:20.612.216.407.72.221.2]Modificado de (Reddy et al., 2002)
Figura 1. A) Primer (AG)n no anclado que se une en cualquier parte de la región repetida de la cadena molde. B) Primer (AG)n anclado con dos nucleótidos (NN) extra en su extremo 5´. Como resultado, amplifica parte de la secuencia repetida y el producto tiene una mayor longitud en comparación con el primero
Los productos de amplificación corresponden generalmente a 200 a 2000pb y el nivel de polimorfismo que se revela puede responder a los distintos métodos de detección. Se ha reportado una mayor resolución de bandas con la técnica de electroforesis en gel de poliacrilamida (PAGE) con radioactividad, seguido por PAGE con tinción con plata y gel de agarosa. La longitud de los primers utilizados con ISSR permiten que sea una técnica
altamente reproducible (Reddy et al., 2002)
d. Extracción de ADN
Para lograr llevar a cabo el estudio de diversidad que se pretende desarrollar por medio de marcadores ISSR es indispensable la disponibilidad de ADN genómico que proceda de un método de extracción que pueda ejecutarse en el laboratorio de forma simple y rápida sin implicar un alto costo (Aubakirova et al., 2014).
La estandarización de protocolos de ADN es necesaria debido a que los procedimientos pueden ser específicos y variar incluso entre individuos de especies del mismo género. Además, ciertos componentes celulares extraídos pueden llegar a inhibir las reacciones moleculares (Aubakirova et al., 2014)
21
el ADN de endonucleasas y finalmente el aislamiento del ADN de polifenoles, proteínas, RNA y polisacáridos (Aubakirova et al., 2014).
El primer paso de la extracción corresponde a la disrupción del tejido que consiste en romper las paredes celulares para facilitar la interacción con los componentes del buffer de lisis. Este proceso puede realizarse de forma química (en presencia de componentes como detergentes y proteasas que rompen las uniones celulares) o de forma mecánica (utilizando nitrógeno líquido que congela inmediatamente la muestra evitando la degradación del ADN por DNasas) (Henry, 2008).
Para que el ADN tenga acceso al medio de extracción debe ser liberado de la membrana celular. Entre los componentes del Buffer de extracción se incluyen diferentes tipos de detergentes que permiten la liberación del ADN unido a la membrana; También se adiciona Tris (pH 7.0-8.0) que actúa como agente para controlar el pH de la solución, EDTA que actúa como agente quelante para inactivar la actividad de las nucleasas, sales para solubilizar el ADN y otras moléculas como β mercaptoetanol para prevenir actividades de la peroxidasa o polifenoloxidasa (Henry, 2008).
En el siguiente paso que corresponde a la purificación que se realiza con agentes orgánicos como mezclas de fenol, cloroformo e isoamilalcohol. Posteriormente es necesario separar la fase orgánica y la fase que contiene el ADN (sobrenadante). Luego de la extracción orgánica el ADN se precipita por acción de componentes como isopropanol. Finalmente se hidrata el ADN y se resuspende para mantenerlo en solución, para esto se puede emplear agua a un pH neutro o Buffer TE (Mezcla de Tris-HCl a 10mM y EDTA a 0.1M a un pH de 8.0) (Cornejo, Serrato, Rendón, & Rocha, 2014).
Al finalizar el procedimiento, es necesario evaluar el producto obtenido cuantificando mediante espectrofotometría. De esta forma se puede estimar la concentración y pureza (evaluada mediante proporciones de absorbancia). Adicionalmente es importante observar la integridad de la muestra mediante electroforesis (Cornejo et al., 2014)
22
e. Estudios de diversidad de ibia por medio de ISSR
Los aspectos relacionados con la diversidad genética de esta especie no han sido muy evaluados y las investigaciones reportadas se han realizado en su mayoría en países andinos distintos de Colombia como Ecuador, Perú y Bolivia, evaluando la diversidad genética principalmente a través de marcadores moleculares ISSR (inter-secuencias simples repetidas) ( Pissard et al , 2006; Malice et al., 2007; Pissard et al., 2008).
Al igual que ISSR, se ha implementado la utilización de marcadores AFLP (polimorfismos en la longitud de fragmentos amplificados) para detectar variación entre taxones cercanamente relacionados. Sin embargo se indica que los marcadores ISSR pueden tener una mayor sensibilidad para la distinción de individuos y pares clonales. (Moscoe & Emshwiller, 2015).
Entre los estudios principales se encuentran los desarrollados por Pissard et al. (2006). En
una primera aproximación a la diversidad de Oxalis tuberosa, Pissard y colaboradores
evaluaron la diversidad entre accesiones de distintos países (Argentina, Bolivia, Perú y Chile), provenientes de la colección de germoplasma del CIP (Centro internacional de la papa en Lima, Perú). Los resultados arrojados agrupan las accesiones en conjuntos o clusters de manera que sugieren que los marcadores ISSR permiten distinguir genotipos cultivados de acuerdo al sitio de colección. Esto podría indicar que es posible que la estructura genética de la ibia se encuentre influenciada por la distancia geográfica entre áreas de cultivo (Pissard et al. 2006).
En un estudio posterior Pissard y colaboradores (2008) evalúan la congruencia entre marcadores moleculares y morfológicos en la diversidad de accesiones agrupadas en cinco morfotipos de diferentes regiones de Perú. En los resultados las accesiones correspondientes al mismo sitio de colección tendieron a agruparse (patrón similar al observado en el anterior estudio). Sin embargo a pesar de que se presentó una alta correlación entre marcadores morfológicos y moleculares, el autor expone que esto puede atribuirse a una relación cercana entre las accesiones y cabe la posibilidad de que no se presente la misma congruencia con un rango mayor de muestras incluidas en un estudio (Pissard et al. 2008).
23
morfotipos de ibia (denominados Señora Oca, K´ellu Qayara y Puca kamusa) cuyas muestras provienen de orígenes de conservación in situ y ex situ en Bolivia. Los análisis moleculares
arrojan que no fue posible diferenciar dos de los tres morfotipos como variedades independientes. La totalidad de los grupos reconocidos como morfotipos presentaron una variabilidad genética dentro de los morfotipos. Los autores determinaron una mayor variación molecular entre las accesiones ex situ que las in situ (Pissard et al., 2008). Por otro
lado, Malice y colaboradores (2007) evaluaron seis morfotipos y obtuvieron una variación mayor en las muestras conservadas in situ en comparación con las ex situ. En general, como
en el estudio de Pissard, se evidenció diferenciación genética entre los morfotipos al igual que un nivel de heterogeneidad dentro de estas (Malice et al., 2007).
Si bien estos trabajos proveen una primera perspectiva de la diversidad de la ibia, la disponibilidad de información es limitada y los tamaños de las muestras incluidos no parecen ser lo suficientes para proveer un análisis más robusto.
Hasta el momento los esfuerzos de caracterización de diversidad de la ibia en Colombia se han llevado a cabo a través de marcadores morfológicos. Rosero (2010) realizó un trabajo de investigación en seis territorios indígenas en el departamento de Nariño en el cual por medio de descriptores distinguió ocho grupos entre los que se distribuyen las variedades conocidas comúnmente por su color. A partir de esta trabajo se conformó el Centro de Conservación de raíces y tubérculos andinos del Suroccidente Colombiano ubicado en el resguardo indígena el Gran Cumbal (Nariño) que cuenta con 32 accesiones, este se estableció como una estrategia de conservación y caracterización, pero sin embargo no constituye una muestra significativa (Rosero, 2010).
Con la necesidad de fortalecer las investigaciones de esta especie en Colombia para lograr implementar estrategias sostenibles de conservación, es necesario complementar los estudios de diversidad incluyendo caracterizaciones genéticas con métodos moleculares.
24
Pregunta de investigación
Teniendo en cuenta que para la zona de los municipios de Turmequé y Ventaquemada se han reportado cinco morfotipos de ibia que corresponden a los colores del tubérculo blanco, amarillo, rojo, rosado y amarillo con rayas rojas, ¿Existe una correspondencia entre la diversidad de morfotipos de Oxalis tuberosa M. identificados por medio de características
morfológicas en los municipios de Ventaquemada y Turmequé (Boyacá) y la caracterización molecular de la diversidad genética realizada por medio de marcadores moleculares ISSR?
Objetivos
Objetivo general
Evaluar la diversidad genética de los morfotipos colectados de Oxalis tuberosa M. existentes
en los municipios de Ventaquemada y Turmequé por medio de marcadores moleculares ISSR.
Objetivos específicos
- Estandarizar el protocolo de extracción de ADN adecuado para muestras de Oxalis
tuberosa provenientes de municipios de Ventaquemada y Turmequé (Boyacá)
- Estandarizar un protocolo de amplificación por PCR de muestras de Oxalis tuberosa provenientes de municipios de Ventaquemada y Turmequé (Boyacá) con marcadores moleculares ISSR
- Realizar la evaluación de la diversidad genética de las muestras de Oxalis tuberosa colectadas en municipios de Ventaquemada y Turmequé (Boyacá) por medio de marcadores moleculares ISSR
Metodología
El presente trabajo consiste en una primera aproximación al análisis de diversidad de Oxalis tuberosa M. en la región de Turmequé y Ventaquemada en el departamento de Boyacá por
medio de marcadores moleculares ISSR. El enfoque metodológico de esta investigación se compone de una fase de campo y una de laboratorio. Dentro de esta última se describe a su vez tres etapas acordes con los objetivos específicos formulados. El trabajo se desarrolló en conjunto con otro estudio de investigación de diversidad genética de cubios (Tropaeolum tuberosum). Se busca articular los resultados de estos trabajos junto con otras investigaciones
25
efectos que han tenido influencia en los patrones de diversidad e identificar propiedades de estos tubérculos que exalten las bondades de su consumo.
Fase de campo:
Localización y material vegetal
El estudio se llevó a cabo en diferentes agroecosistemas ubicados en los municipios de Turmequé (ubicado en provincia de Márquez) y Ventaquemada (ubicado en Provincia Centro), ambos localizados en el departamento de Boyacá. Este departamento, de gran importancia agrícola ubicado en el centro del país, abastece a las ciudades más importantes de Cundinamarca, Boyacá e incluso otras regiones de Colombia. Estos municipios se caracterizan por un legado que conserva aún en la actualidad tradiciones de comunidades indígenas y el consumo de especies vegetales nativas entre las que se incluyen los tubérculos andinos hace parte de las tradiciones culturales de esta región (Clavijo et al., 2014).
Turmequé se encuentra a 73° 30´ al oeste de Greenwich a una altura de 2389 msnm y Ventaquemada se encuentra a 73° 32´al oeste de Greenwich a 2630 msnm (Clavijo, 2014).
La colecta de material se efectuó en diferentes fincas de pequeños productores en ambos municipios, considerados centros de conservación in situ. Con la colaboración de los
[image:25.612.85.525.637.714.2]cultivadores se realizó un reconocimiento de la zona, las parcelas y los morfotipos cultivados. Se registraron las coordenadas geográficas de cada sitio de colecta y datos relacionados con características particulares de los cultivos, usos o prácticas de manejo, los cuales fueron consignados en un formato (Anexo 1) diligenciado para cada accesión. Estos datos son fundamentales en el proceso de análisis, para lograr relacionar los resultados arrojados por el análisis de diversidad con los factores que han ejercido una influencia sobre los agroecosistemas y la siembra de estos tubérculos de estudio, en este caso particular, de la ibia. La tabla a continuación resume las características biofísicas básicas registradas en campo.
Tabla 1. Resumen de características reportadas en campo
Numero de Finca
Accesiones colectadas
Municipio Coordenadas Altura msnm Observaciones adicionales
1 020
022 032 034 035
Ventaquemada N 05°22´39,1”
26
se siembran los cultivos se encuentra en una ladera.
2 036
037 038 040 041 043 044
Ventaquemada N 05°25´47,1”
W073°30´41,0” 3027 Parcela en terreno plano. Gran densidad de vegetación, cultivo de ibia alternado aleatoriamente con cultivos de cubios, rubas, habas, maíz y otros.
3 051
052 053 054 055 056
Ventaquemada N 05°22´57,5”
W073°30´21,6” 2735 Cultivos situados en surcos, de forma más homogénea en el terreno, es posible identificar más fácilmente individuos de plantas aislados.
4 064 Turmequé N 05°19´15,2”
W073°29´59,2” 2387 Asociación de varios cultivos diferentes
5 068
069 071 074
Turmequé N 05°19´18,7”
W073°30´35” 2368 Plantas de ibia asociados a otros cultivos.
6 075 Turmequé N 05°18´58,9”
W073°31´38,8” 2374 Asociación de varios cultivos diferentes
7 079
080 081 082
Ventaquemada N 05°23´54,6”
W073°29´09.1” 2848 Plantas de ibia asociados a otros cultivos. Gran densidad de vegetación.
Se tomaron muestras de tejido foliar correspondientes a los diferentes morfotipos de tubérculos de ibia existentes, para el posterior análisis molecular en el laboratorio. Estas muestras se almacenaron inmediatamente en bolsas plásticas que contenían gel de sílice para absorber la humedad y evitar la rápida oxidación de los tejidos. Posteriormente para conservar las muestras en el laboratorio, fueron almacenadas en nevera a -80°C.
27
Las colecciones de herbario representan fuentes de información fundamentales en cualquier estudio relacionado con plantas y su conservación, por esta razón se considera una herramienta importante para incluir en este trabajo ya que será un complemento del proceso de investigación realizado (Vera et al., 2005). Las muestras botánicas incluyendo los
tubérculos estarán disponibles como elementos de referencia en el herbario, y los datos asociados representarán también un registro importante en cuanto a la especie, la región y ecosistema. De esta forma estos registros pueden ser utilizados como referencias para futuras investigaciones en esta y otras regiones del país.
Fase de laboratorio Extracción de ADN
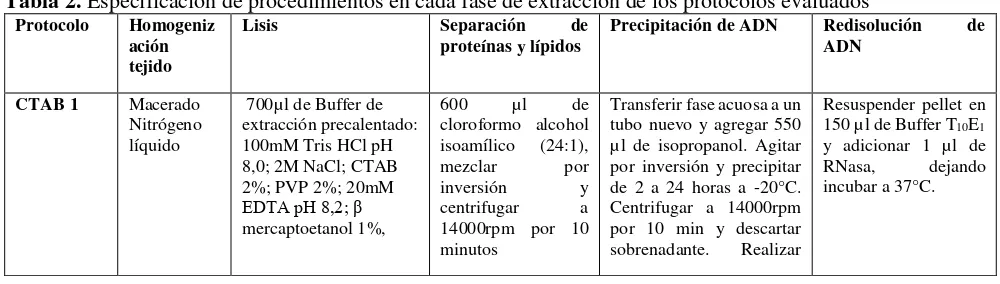
Para el procedimiento de extracción de ADN se evaluaron cinco protocolos, cuya prueba tuvo dos repeticiones.
CTAB1 ( Pissard et al., 2006), SDS (Dellaporta et al, 1983 modificado), CTAB 2 (Doyle &
Doyle, 1990 modificado), Stefenon (Stefenon et al., 2004) y CTAB 3 (Doyle & Doyle, 1990
modificado) (Tabla 2).
[image:27.612.83.583.583.725.2]En todos los casos, se realizó una maceración del material vegetal colectado con nitrógeno líquido empleando un mortero. A la totalidad de los protocolos se agregaron aproximadamente 0,2g de material vegetal macerado a cada buffer de extracción específico en tubos de centrifuga de 0,5 - 2ml. En los pasos finales de cada protocolo se realizó un lavado con etanol al 70 % que puede permitir la remoción de reactivos que inhiben el proceso de PCR, se descartó el sobrenadante y como paso final, el pellet se resuspendió con buffer T10E1 y se trató con RNasa.
Tabla 2. Especificación de procedimientos en cada fase de extracción de los protocolos evaluados
Protocolo Homogeniz ación tejido
Lisis Separación de
proteínas y lípidos
Precipitación de ADN Redisolución de ADN
CTAB 1 Macerado Nitrógeno líquido
700µl de Buffer de extracción precalentado: 100mM Tris HCl pH 8,0; 2M NaCl; CTAB 2%; PVP 2%; 20mM
EDTA pH 8,2; β
mercaptoetanol 1%,
600 µl de cloroformo alcohol isoamílico (24:1), mezclar por inversión y centrifugar a 14000rpm por 10 minutos
Transferir fase acuosa a un tubo nuevo y agregar 550 µl de isopropanol. Agitar por inversión y precipitar de 2 a 24 horas a -20°C. Centrifugar a 14000rpm por 10 min y descartar sobrenadante. Realizar
28
mezclar con agitador Vortex e incubar 65°C por 40 min
lavado con 500 µl etanol al 70%, centrifugando esta vez por 5min a 14000rpm.
SDS Macerado
Nitrógeno líquido
700µl de Buffer de extracción precalentado: 100mM Tris HCl pH 8,0; 5M NaCl; SDS 2%;
50mM EDTA pH 8,2; β
mercaptoetanol 10mM mezclar con agitador Vortex e incubar 65°C por 30 min mezclando por inversión cada 5min
500 µl de cloroformo alcohol isoamílico (24:1), mezclar por inversión 5min y centrifugar a 14000rpm por 10 minutos
Transferir sobrenadande a un tubo nuevo y adicionar 1/10 volumen de Acetato de Sodio 3M pH5,3 y 6/10 volumen de isopropanol, mezclando por inversión y dejando incubar a -20°C
por 30 min.
Posteriormente
centrifugar a 14000rpm por 15 min, descartar el sobrenadante dejar secar precipitado a temperatura ambiente. Lavar el precipitado con 500 µl de etanol a 70 %, y centrifugar por 5 min a 14000rpm
Resuspender pellet en 100 µl de Buffer T10E1 y adicionar 1 µl de RNasa, dejando incubar a 37°C.
CTAB 2 Macerado Nitrógeno líquido
600µl de Buffer de extracción precalentado: 100mM Tris HCl pH 8,0; 1,4M NaCl; CTAB 3%; PVP 1%; 20mM
EDTA pH 8,2; β
mercaptoetanol 0,2% mezclar con agitador Vortex e incubar 65°C por 20 min mezclando por inversión cada 5min
600 µl de cloroformo alcohol isoamilico (24:1), mezclar por inversión 5min y centrifugar a 13000rpm por 10 minutos
Transferir sobrenadande a un tubo nuevo y realizar un lavado con etanol absoluto (2.5 volumenes) más acetato de amonio 1/10 de volumen mezclando de nuevo por inversión. Incubar por 2 horas a -20°C. Posteriormente se centrifugar a 13000rpm por 5 min, descartar el sobrenadante y realizar lavado con 500 µl etanol al 70%. Centrifugar por 5min a 13000rpm
Resuspender pellet en 100 µl de Buffer T10E1 y adicionar 1 µl de RNasa, dejando incubar a 37°C.
Stefenon
et al 2004
Macerado Nitrógeno líquido
1500µl de Buffer de extracción precalentado: 100mM Tris HCl pH 8,0; 1,5M NaCl; CTAB 2%; PVP 2%; 20mM
EDTA pH 8,2; β
mercaptoetanol 1% mezclar con agitador Vortex e incubar 65°C por 20 min mezclando por inversión cada 15min
Adicionar 600 µl de cloroformo alcohol isoamilico (24:1), mezclar por inversión y centrifugar a 13000rpm por 5 minutos.
Transferir fase acuosa a tubo nuevo y adicionar 5µl de RNasa. Incubar a 34°C por 40 min. Posteriormente adicionar 600µl de cloroformo alcohol isoamilico.
Centrifugar 5min a 13000rpm y transperir
sobrenadante. Adicionar solución 1 (CTAB 10% y
Agregar 2/3 de volumen de isopropanol y dejar 30 min a -20°C. Se centrifuga a 4000 rpm por 5 min y se descarta el sobrenadante. Lavar el pellet con etanol al 75% e incubar a -20°C por 30 min. Centrifugar a 13000rpm por 5 min y descartar sobrenadante.
29
NaCl 1,4M). Adicionar 600µl de cloroformo y centrifugar
nuevamente 5min a 13000.
CTAB 3 Macerado
Nitrógeno líquido
600µl de Buffer de extracción
precalentado: 100mM Tris HCl pH 8,0; 1,4M NaCl; CTAB 3%; PVP 1%; 20mM EDTA pH 8,2; β mercaptoetanol 0,2% mezclar con agitador Vortex e incubar 65°C por 20 min mezclando por inversión cada 5min
Adicionar 600 µl de cloroformo alcohol isoamílico (24:1), mezclar por inversión 5min y centrifugar a 13000rpm por 5 minutos
Transferir sobrenadande a un tubo nuevo y realizar un lavado con etanol absoluto (2.5 volumenes) más acetato de amonio 1/10 de volumen mezclando de nuevo por inversión. Incubar por 2 horas a -20°C.
Posteriormente se centrifugar a 13000rpm por 5 min, descartar el sobrenadante y realizar lavado con 500 µl etanol al 70%. Centrifugar por 5min a 13000rpm
Resuspender pellet en 150 µl de Buffer T10E1 y adicionar 1 µl de RNasa, dejando incubar a 37°C.
Métodos de análisis del ADN
Por medio de los parámetros de calidad (pureza e integridad) y concentración, se evaluaron los resultados obtenidos en las pruebas para lograr discernir entre los diferentes protocolos y elegir el más adecuado para las muestras de O. tuberosa.
La cuantificación de ADN se realizó utilizando un espectrofotómetro NanoDrop™ Thermo scientific. La valoración de la pureza se realizó a través de la proporción de absorbancias A 260 nm/ 280nm el valor ideal en el que se considera ADN puro corresponde a un rango entre 1,8 y 2,0. Valores menores a este, pueden indicar contaminación de la muestra con proteínas. Una segunda proporción utilizada para determinar la pureza corresponde a A 260/230, donde los valores apropiados se consideran entre el rango 2.0 - 2.2 y un valor menor puede reflejar contaminación por carbohidratos o fenoles (Cornejo et al., 2014).
Para determinar la integridad de las muestras se realizaron ensayos de electroforesis en gel de agarosa, utilizando el colorante fluorescente SYBR® Safe. (Weising et al., 2015).
PCR
Para el análisis molecular se emplearon primers ISSR reportados como polimórficos por
Pissard et al. (2006) (tabla 3) y se evaluó el protocolo también reportado por este autor en
30
Tabla 3. Primers ISSR y su temperatura óptima de anillamiento (tomado de Pissard et al 2008)
En primera instancia se realizaron pruebas preliminares con primers de marcadores
moleculares RAM (Random Amplified Microsatellite) para tener una primera aproximación a la estimación de la calidad de la amplificación y determinar si el ADN extraído posee la calidad apropiada para llevar a cabo técnicas moleculares como la PCR. Estas pruebas se efectuaron con 5 primers (figura 5) preparando reacciones con un volumen de 25 µL y
utilizando concentraciones de 50ng del ADN genómico. La concentración de componentes en cada reacción preparada equivale a 1x de Buffer; 2,5mM de MgCl2; 0,25mM de dNTPs; 4mM de Primer y 1U de la enzima Taq polimerasa. Finalmente se evaluaron los productos
por medio de electroforesis en gel de agarosa al 2% a un voltaje constante de 65V.
Para los primers ISSR se probó el protocolo reportado por Pissard (2006) para la PCR a
unvolumen final de 25 µL (volumen modificado del protocolo original) que contenía 10ng de ADN, 0,4 µmol/l de primer, 80 µmol/l cada dNTP, 1U de Taq polimerasa y 1x de Buffer
. Para los primers 1-6 (tabla 3), en primer lugar se realizó una denaturación por 10 min a
95°C (35 ciclos) y la elongación final se efectuó por 5 min a 72°C. Para los primers restantes,
el primer paso de denaturación es de 5 min a 94° (35 ciclos) y un paso final de elongación de 4 min a 72 °C. Los productos de PCR se analizaron por electroforesis con un gel de agarosa al 2% a un voltaje constante de 70 V.
31
restringió el gradiente a valores cercanos al rango entre 1.5 a 3 reportado por Pissard et al.
(2006). Las mejores condiciones se determinaron de acuerdo con la intensidad y claridad de las bandas en la electroforesis.
Análisis de Diversidad Genética
Para la evaluación de la diversidad de morfotipos de ibia, se analizaron los fragmentos de ADN amplificados de cada una de las 29 accesiones (Anexo 3) con los primers ISSR
escogidos. Los fragmentos se consideran como un carácter unitario y se evidencian por medio de bandas que se visualizan en un gel de electroforesis. El análisis inicia asignando un valor binario para la presencia (1) y la ausencia (0) de las bandas. Teniendo en cuenta estudios reportados que utilizan ISSR, es recomendable tomar en cuenta para la puntuación aquellas bandas con una separación de aproximadamente 0,5mm y cuya banda se pueda evidenciar fácilmente (evitar fragmentos con aplificación tenue) debido a que estas condiciones aumentan las posibilidades de reproducibilidad de los ensayos (Joshi et al.,2000; Chia, 2009).
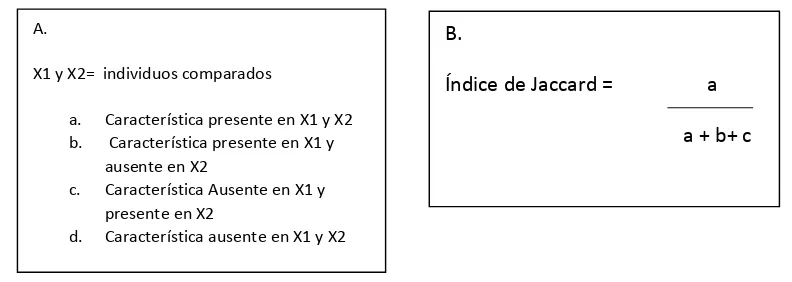
[image:31.612.91.487.525.666.2]Con los valores obtenidos de presencias y ausencias se diseñó una matriz en donde se enfrentaron las accesiones evaluadas y los loci o caracteres tenidos en cuenta. Posteriormente se realizó una matriz de similitud, producto del cálculo de un coeficiente de similitud para los datos, utilizando el software estadístico PAST. Los datos se obtuvieron en principio empleando el coeficiente de Jaccard. Este coeficiente adquiere valores de 0 a 1 y se basa en la relación de las presencias y ausencias de una característica, en este caso, en dos muestras o accesiones comparadas. El coeficiente se define a continuación (Figura 2) (Rodríguez et al., 2001).
Figura 2. A) Combinaciones para condiciones de presencia y ausencia de una característica con dos muestras
comparadas. B) Fórmulas para cálculo de índice de Jaccard . B.
Índice de Jaccard = a a + b+ c A.
X1 y X2= individuos comparados
a. Característica presente en X1 y X2 b. Característica presente en X1 y
ausente en X2
c. Característica Ausente en X1 y presente en X2
32
Con esta matriz se realizó un agrupamiento con el algoritmo UPGMA (por sus siglas en inglés: Unweighted Pair Group Method using Arithmetic averages) el cual permitió agrupar los individuos de estudio con base en las distancias genéticas. El resultado de la aplicación de este algoritmo utilizando el software PAST fue un dendrograma o árbol fenético donde se evidenció de forma gráfica las distancias y el agrupamiento. De igual forma se obtuvieron representaciones de los agrupamientos en dos dimensiones por medio de un análisis de coordenadas principales, que permitió observar gráficamente la diversidad reflejando la relación entre individuos (Barrera et al., 2004).
Resultados
Extracción de ADN
Como resultado de una primera experiencia en campo se realizó la colecta de 4 individuos (accesiones) de Oxalis tuberosa (Anexo 4), meterial que se procesó para llevar a cabo el
ensayo de comparación de protocolos en el laboratorio de Biologia Molecular Vegetal de la Pontificia Universidad Javeriana. El formato de datos de campo de la colecta se incluye en el Anexo 1.
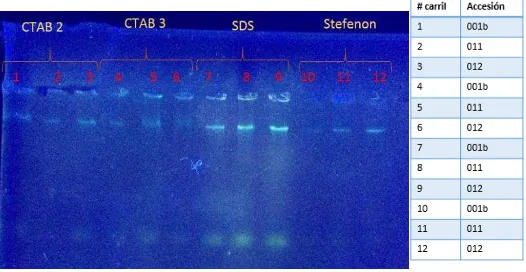
[image:32.612.122.492.526.697.2]En la fase extracción de ADN en el laboratorio se pusieron a prueba los protocolos especificados en la metodología (CTAB 1, SDS, CTAB2, Stefenon y CTAB 3) con las muestras colectadas.
Tabla 4. Datos de concentración, pureza e integridad de los protocolos evaluados con muestras de ibia.
# de accesión
ng/ul a 260/280 a 260/230 presencia de bandas
CTAB 1 005 379,79 2,26 0,99 +
011 275,13 1,89 0,97 +
012 434,28 2,23 1 +
SDS 001b 1022,74 1,93 0,88 +
011 976,63 1,72 0,86 +
33
CTAB 2 001b 202,77 2,34 0,73 +
011 107,89 2,08 0,91 +
012 281,81 2,11 0,93 +
CTAB 3 001b 210,89 2,33 0,73 +
011 531,22 2,12 1,06 +
012 374,43 2,13 0,94 +
STEFENON MODIFICADO
001b 61,03 2,54 0,36 +
011 51,5 2,54 0,45 +
012 32,77 2,6 0,41 +
Los resultados de concentración arrojados por espectrofotometría indican una mayor concentración en el caso del protocolo SDS, seguido de CTAB 3, CTAB 1 (con un valor muy cercano a CTAB 3), CTAB 2 y finalmente Stefenon que registró valores mucho menores en comparación con los otros protocolos (el gráfico de medias de concentración se incluye en el anexo 5)
En cuanto a los resultados de estimación de la pureza, teniendo en cuenta que se considera un ADN puro aquel cuya proporción de absorbancia 260 /280 arroje un valor mayor a 1,8, la totalidad de los protocolos presentan valores óptimos. Si bien los valores no difieren en gran proporción entre los protocolos, Stefenon modificado exhibe un valor mayor, seguido por CTAB 3, CTAB 2, CTAB 1 (con un valor muy cercano a CTAB 2) y SDS. La gráfica de medias de valores de proporciones de absorbancia (anexo 5) permite corroborar estos resultados demostrando la cercanía entre las medias de los valores. Por otro lado, la proporción 260 /230 utilizada como indicador de contaminación por compuestos como carbohidratos y fenoles, no alcanza en ningún resultado el rango óptimo de pureza considerado entre 2.0 y 2.2. El valor más alto se registra con CTAB 1.
34
[image:34.612.175.438.124.260.2]bandeo de mayor claridad para CTAB 1, con bandas más definidas cercanas a los pozos de siembra
Figura 3. Electroforesis en gel de agarosa 0,8% con muestras de ibia y los resultados de la extracción de ADN
con los protocolos CTAB 2, CTAB 3, SDS y Stefenon
Finalmente, se escogió el protocolo CTAB 1, que ofrece un buen equilibrio entre los criterios analizados. El resultado de electroforesis de la extracción con el protocolo CTAB1 con la totalidad de las muestras colectadas se incluye en el anexo 13. Para evaluar la calidad del ADN se probó la amplificación por medio de primers de marcadores RAM (Microsatélites
Amplificados al Azar), la cual fue exitosa (Anexo 6) (el protocolo de PCR se encuentra en el anexo 7).
Condiciones de PCR
Luego de la extracción de ADN se efectuaron las pruebas para observar la amplificación con los 9 primers de ISSR elegidos según estudios previos de Pissard et al. (2006).
Con el fin de elegir las condiciones más adecuadas para la amplificación, las reacciones se prepararon según las especificaciones establecidas por Pissard y colaboradores (2006) comparándolas con las condiciones utilizadas previamente para la amplificación con primers
de los marcadores RAM. Los mejores resultados de amplificación se obtuvieron con las condiciones evaluadas para los primers de RAM (Anexo 8).
Para optimizar los resultados de amplificación se realizaron modificaciones fundamentalmente en las concentraciones de magnesio. Los resultados obtenidos indican una mejor definición de bandas incorporando a la reacción una concentración de MgCl2 de 3mM
35
Bajo estas condiciones se efectuaron pruebas con los primers ISSR restantes para verificar
su amplificación. La totalidad de los primers amplificaron para el ADN de O. tuberosa
extraído, sin embargo, los controles negativos3 permitieron identificar contaminación en
cuatro primers (DHB (CGA)5; VHV (GT)7G; (AG)8T; DDC (CAC) 5), los cuales se
excluyeron de las pruebas posteriores de diversidad. Dos de estos primers (DHB (CGA)5 y
VHV (GT)7G) fueron reemplazados por primers RAM (GT y CGA) que tienen la misma
secuencia microsatélite. Sin embargo, con el primer CGA no se obtuvo amplificación del
ADN de las muestras.
En general comparando los diferentes primers, aquellos que permiten distinguir bandas con
mayor definición son los que incluyen repeticiones de nucleótidos AG, GA y AC. Finalmente, considerando los resultados de las pruebas, las condiciones elegidas para realizar la PCR fueron: ADN a 25ng; Buffer 1x, MgCl2 a 3mM, dNTPs 0.25mM, primers 4µM y
1U de Taq polimerasa, en un volumen final de 20ul. Las temperaturas de anillamiento y los protocolos de PCR usados fueron los propuestos por Pissard et al. (2006) (Anexo 9).
Polimorfismo
En la amplificación de la totalidad de las muestras con los primers elegidos se detectaron
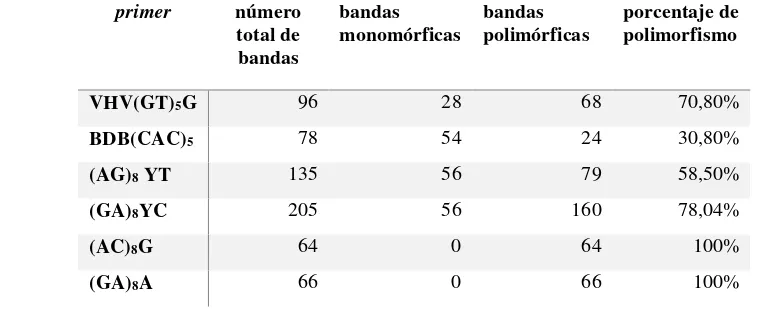
[image:35.612.99.482.501.665.2]tamaños de fragmentos desde 150 hasta 1700 pb. A través de la matriz binaria generada se calcularon los porcentajes de polimorfismo de cada uno de los 6 primers utilizados (tabla 5).
Tabla 5. Porcentajes de polimorfismo para primers ISSR seleccionados
primer número
total de bandas
bandas monomórficas
bandas polimórficas
porcentaje de polimorfismo
VHV(GT)5G 96 28 68 70,80%
BDB(CAC)5 78 54 24 30,80%
(AG)8 YT 135 56 79 58,50%
(GA)8YC 205 56 160 78,04%
(AC)8G 64 0 64 100%
(GA)8A 66 0 66 100%
3Los controles negativos se incluyen en los experimentos para verificar si hay presencia de alguna contaminación. Estos
36
El menor grado de polimorfismo equivale a un 30% y se obtuvo con el primer BDB (CAC)5.
Los primers más polimórficos fueron (AC)8G y (GA)8A reportando un 100% de
polimorfismo.
Evaluación de la diversidad
Utilizando los 6 primers (tabla 5) que resultaron libres de contaminación y arrojaron una
buena amplificación, se obtuvieron resultados para las accesiones colectadas. La información proveniente de las bandas evidenciadas en las electroforesis se procesó para obtener una matriz binaria donde se registró la presencia (1) y la ausencia (0) de cada banda o locus en los individuos evaluados (Anexo 10 )
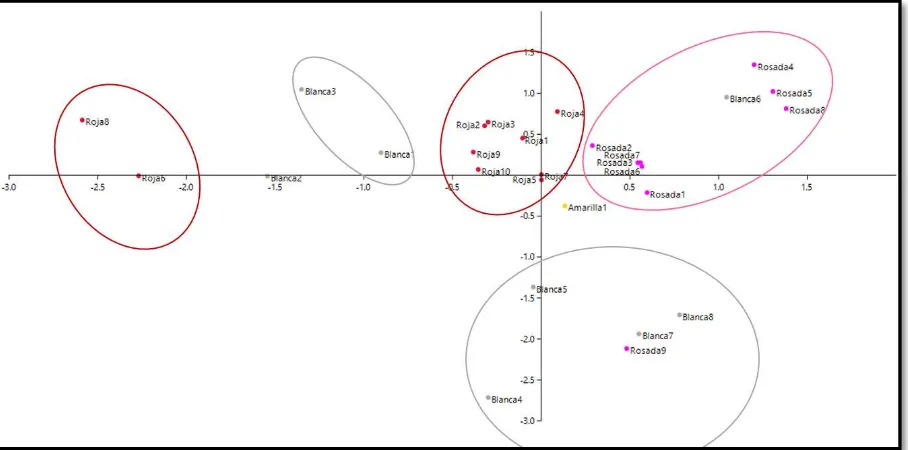
Con el análisis de diversidad con el coeficiente de Jaccard se obtuvieron distancias que oscilaron entre 0.296 y 0.909 lo cual evidencia una alta variabilidad intraespecífica entre los morfotipos evaluados.
El dendrograma obtenido por medio del software estadístico PAST utilizando el coeficiente de Jaccard se resuelve en un punto intermedio de similitud entre los valores 0.6 y 0.66 (Figura 5). A este nivel se definen 5 clusters o agrupaciones y una accesión aislada. De estos, los clusters 2, 3 y 4 agrupan la mayor cantidad de muestras y en general corresponden a los morfotipos identificados por colores (rojo, rosado y blanco respectivamente). El cluster 1 contiene dos accesiones del morfotipo blanco que se separan de las demás de su tipo, al igual que dos accesiones rojas (roja 6 y 8) que se agrupan en el cluster 5. Por último, se muestra una accesión aislada que corresponde a la única muestra del morfotipo amarillo (Figura 5).
37
Figura 4. Análisis de coordenadas principales realizado con coeficiente de Jaccard para 29 muestras de ibia
38
Figura 5. Dendrograma realizado por el método de agrupamiento UPGMA utilizando el coeficiente de
39
Discusión
Extracción de ADN
Con un proceso de extracción exitoso, es posible el desarrollo de investigaciones con técnicas moleculares, logrando obtener resultados confiables y reproducibles.
La principal limitación de la extracción ADN en plantas se atribuye al contenido de proteínas, polisacáridos (que pueden encontrarse en la pared celular o en acumulaciones como gránulos de almidón), taninos (u otros compuestos fenólicos), pigmentos y RNA que obstaculizan el aislamiento y la purificación de la molécula (Bryant, 1997; Puchooa, 2004). Durante la extracción los metabolitos secundarios pueden liberarse y co-precipitarse con el ADN. Estos contaminantes son evidenciables durante la extracción e impiden la manipulación de la molécula de ADN en procesos como el pipeteo y la resuspensión final del pellet ( Porebski
et al., 1997; Puchooa, 2004; Aubakirova et al., 2014)
Las etapas generales de los protocolos manuales de extracción comprenden: la homogenización o ruptura de uniones celulares del tejido vegetal; etapa de lisis celular por medio de un Buffer de lisis que incluye detergentes ; separación de proteínas y lípidos; precipitación y finalmente resuspender el ADN resultante (Cornejo et al., 2014). En la fase de colecta de material previa a este protocolo es importante buscar en lo posible obtener muestras frescas y en caso de ser tejido foliar, preferiblemente evitar colectar hojas maduras con mayor contenido de metabolitos secundarios (Porebski et al., 1997; Wangsomnuk et al.,2014).
Dentro del Buffer de lisis puede haber diversos tipos de detergentes que permiten la liberación del ADN. Entre los más comunes se encuentra el Bromuro de cetiltrimetilamonio (CTAB) y el dodecilsulfato sódico (SDS).
40
un protocolo que incluya CTAB es más apropiado para la extracción de ADN de muestras de ibia.
Estos resultados son consistentes con reportes en la literatura en los que extracción con SDS en especies con altos contenidos de polifenoles resulta en una baja concentración y pureza (Minas et al., 2011; Puchooa, 2004). Li et al. (2007) muestran resultados de electroforesis
con bandas degradadas utilizando un protocolo SDS para extracción de ADN de girasol (Helianthus annuus L.).
Los demás protocolos evaluados (CTAB 2, Stefenon y CTAB 3), además de contener CTAB, poseen otras sustancias adicionales como NaCl, PVP (polivinilpirrolidona), EDTA y β- mercaptoetanol. Estos componentes evitan la degradación del ADN, actúan como antioxidantes y contribuyen a la eliminación de polifenoles y polisacáridos ( Porebski et al.,
1997; Cornejo et al., 2014)
Para lograr separar proteínas y lípidos se utilizan solventes orgánicos, en este caso Cloroformo alcohol isoamilico (24:1) (Cornejo et al., 2014). Los protocolos que incluyen
solventes orgánicos generan un mejor resultado que protocolos que no utilizan estos solventes, reportando una gran efectividad (mayor y mejor purificación) en combinar altas concentraciones de CTAB, NaCl y posteriormente realizar dos lavados con cloroformo- alcohol isoamílico (Minas et al., 2011; Sá et al.2011). Los protocolos CTAB 1, 2, 3 y SDS
concuerdan con estas condiciones (doble lavado con cloroformo alcohol isoamílico) en su fase de separación de lípidos y proteínas. A esto podría atribuirse los resultados del índice 260/280 que arrojan valores apropiados de pureza para los protocolos.
Con los resultados obtenidos se escogió el protocolo CTAB 1 que arrojó resultados adecuados para los criterios evaluados. En promedio se obtuvo una concentración de 363,07 ng/ul. Este protocolo fue reportado por Pissard et al. (2006) para Oxalis tuberosa y se incluye
en reportes de estudios de diversidad con tubérculos andinos ( Pissard et al., 2006; Malice et al., 2007; Pissard et al., 2008 .
La amplificación exitosa con primers RAM indica que el ADN obtenido se encuentra libre