Tecnologías para el almacenamiento de datos usados en la visualización científica
Texto completo
(2) Hago constar que el presente trabajo fue realizado en la Universidad Central Marta Abreu de Las Villas como parte de la culminación de los estudios de la especialidad de Ciencias de la Computación, autorizando a que el mismo sea utilizado por la institución, para los fines que estime conveniente, tanto de forma parcial como total y que además no podrá ser presentado en eventos ni publicado sin la autorización de la Universidad.. Firma del autor. Los abajo firmantes, certificamos que el presente trabajo ha sido realizado según acuerdos de la dirección de nuestro centro y el mismo cumple con los requisitos que debe tener un trabajo de esta envergadura referido a la temática señalada.. Firma del tutor. Firma del jefe del Seminario.
(3) D E. D I. C. A T O R I. A. DEDICATORIA A mis extraordinarios padres, de lo cuales me siento orgulloso y que tanto se han sacrificado por mí. A mi hermano que ha sido excepcional. A mi querida novia por su ayuda, su preocupación y su amor. Reinaldo. Este trabajo, que hasta ahora es el mayor logro profesional que he alcanzado en mi vida, se lo dedico a las personas que más quiero en este mundo: A mi mamá, por su sacrificio sin límites, por su empeño, por todo su amor. A mi papá, por toda su ayuda, y el ejemplo a seguir que siempre ha sido para mí. Yoany.
(4) A G R. A D E. C I M I E. N T O S. AGRADECIMIENTOS En general queremos agradecerles a todas las personas que de una forma u otra han contribuido con esta investigación. No podemos dejar de mencionar a: Nuestros tutores el Dr. Carlos Pérez Risquet y el Msc. Alberto Morell por el tiempo que han dedicado a la terminación de la misma. A nuestros compañeros de estudio Ernesto Sarduy, Yoandry Vázquez y Alejandro por su asesoramiento en la Base de Datos, también Alain Cárdenas Castillo por su contribución con las herramientas de traducción. A los fabricantes del Internet Download Manager, por haber creado un acelerador de descarga tan bueno.. Y en particular:. A mi vecina Ana Ramos por su ayuda en el montaje e impresión del documento. Reinaldo. Quiero agradecer principalmente a Dunia por el gran apoyo que me ha dado, por su preocupación constante, y la seguridad que siempre tuvo en mí, pues sin su ayuda y dedicación, no hubiera sido posible alcanzar este resultado que hoy, es también de ella. A mi hermana por toda su confianza y cariño. A mis amigos por estar siempre pendientes de mi carrera. Yoany.
(5) R E S. U M. E N. RESUMEN En los últimos años ha crecido exponencialmente el volumen de datos producidos y almacenados, gracias al desarrollo acelerado de las tecnologías de la información. Explorar y analizar esta gran cantidad de información es una tarea muy compleja. La visualización científica constituye una solución al problema de tratar con estos grandes volúmenes de datos. Al ser esta un área poco explorada en nuestro país, el presente trabajo intenta un acercamiento a la misma. Con este objetivo en el presente trabajo se hace un estudio de los tipos de datos usados en la visualización científica para su clasificación, de manera que se reflejen las características del espacio de observación y de los datos, dimensionalidad y radio de acción, entre otros. Se estudian, además, las tecnologías de almacenamiento de los datos científicos más utilizadas en la actualidad, como son netCDF, CDF, HDF y DX. También se estudiaron las técnicas de visualización, según la clasificación en cuanto al área de impacto de los datos. Por último, se estudió el uso del paquete de visualización OpenDX, y varias bibliotecas para el trabajo con datos científicos..
(6) A B S. T R. A C T. ABSTRACT In the lasts years, produced and stored data volume has grown exponentially, as a consequence of the quick development of information technologies. To explore and analyze this huge quantity of information is an extremely complex task. Scientific Visualization constitutes a solution to the problem of dealing with these big volumes of data. This is almost an unexplored area in our country, so the present investigation attempts an approach to the same one. With this objective a study of the different data types used in scientific visualization for its classification is made in the current work, to reflect observation space and data characteristics, data dimensionality and data impact area, between others. Are studied, also, the more used storage technologies of scientific data nowadays, like netCDF, CDF, HDF and DX. Visualization techniques were also studied, according to the classification as for the impact area of data. Finally, it was studied the use of OpenDX visualization package and several libraries for the management of scientific data..
(7) Í. N D I. C E. ÍNDICE 1. INTRODUCCIÓN .................................................................................................. 1 1.1. Formatos de almacenamiento de datos, técnicas de visualización y. aplicaciones más comúnmente utilizadas .............................................................. 4 1.2. Objetivo general: ........................................................................................ 9. 1.3. Objetivos específicos: ................................................................................ 9. 2. MODELACIÓN DE DATOS PARA LA VISUALIZACIÓN CIENTÍFICA ................. 10 2.1 Modelos de Datos para la Visualización Científica......................................... 10 2.2 Formatos de Datos utilizados en la Visualización Científica .......................... 19 2.2.1 HDF ........................................................................................................ 19 2.2.2 CDF ........................................................................................................ 24 2.2.3 netCDF ................................................................................................... 30 2.2.4 DX .......................................................................................................... 34 2.3 Uso de bases de datos relacionales para el almacenamiento de datos científicos ............................................................................................................ 40 2.4 Comparación de los diferentes formatos de datos ......................................... 42 3. TÉCNICAS DE VISUALIZACIÓN ........................................................................ 49 3.1 Métodos elementales o puntuales ................................................................. 49 3.2 Métodos locales............................................................................................. 56 3.3 Métodos globales .......................................................................................... 59 4. CONCLUSIONES Y RECOMENDACIONES ....................................................... 70 REFERENCIAS BIBLIOGRÁFICAS ........................................................................ 72 ANEXOS ................................................................................................................. 74.
(8) C. A P. Í. T U L. O. 1. 1. INTRODUCCIÓN En la actualidad existen estaciones de medición de alta precisión, satélites o supercomputadoras que generan diariamente volúmenes de datos tan grandes y complejos, que no pueden ser analizados completamente en forma numérica. En general se asume que de todos los datos generados sólo una cuarta parte se almacena, y de ellos a su vez, sólo una cuarta parte realmente se analiza. Esto trae como consecuencia que se pierdan datos valiosos y sea utilizado solamente un pequeño por ciento de informaciones importantes. Entonces surge la necesidad de nuevos métodos de análisis para examinar estos datos de forma efectiva y extraer de ellos información y conocimientos. Precisamente las técnicas y herramientas de Visualización Científica constituyen el marco apropiado para lograr un mejor análisis de los datos.. Existe una frase popular que justifica el uso de la visualización: "Una imagen vale más que mil palabras". Se estima que 50% de las neuronas está dedicado a la visión, además, la densidad de información por unidad de área es notablemente mayor a la de un texto. Por otro lado, la visualización nos permite ver lo que no es posible ver, es posible reconocer patrones de comportamiento de los datos, ver en una sola imagen o en una secuencia de imágenes (animación) una gran cantidad de datos y nos facilita la comprensión de algunos conceptos, sobre todo de tipo abstracto.. La visualización científica se ha desarrollado en los últimos años como un área de investigación bien definida, que proporciona una alternativa eficaz de solución a problemas que difícilmente pueden enfrentarse con las técnicas tradicionales existentes.. Algunas técnicas de visualización son más apropiadas para un tipo de dato específico que para otros, de ahí la importancia de disponer de una descripción precisa de los datos como punto de partida para una visualización efectiva. Alrededor de esta problemática se han analizado las posibilidades y fronteras de los diferentes métodos de visualización y sus aplicaciones en problemas específicos [1], 1.
(9) C. A P. Í. T U L. O. 1. [2], [5], [6] en una serie de investigaciones, artículos y libros. Sin embargo, existen hasta ahora pocos trabajos que describan esta temática de forma sistémica y uniforme, sin concentrarse en un área de aplicación especial o una clase específica de algoritmos.. Las Técnicas de Visualización Científica posibilitan realizar un análisis rápido y eficiente, de la gran cantidad de información que es generada por satélites, sensores, radares, estaciones meteorológicas, etc., evitando que datos importantes sean omitidos por equivocación, facilitando la comprensión de conceptos complicados, indicando cuáles datos son necesarios por su importancia y cuáles no lo son, y facilitando la comunicación entre científicos. El problema más grave de la visualización es la complejidad —el número de dimensiones— de los datos. Si, por ejemplo, se quiere visualizar al mismo tiempo la posición, la velocidad, la dirección, la energía y la carga eléctrica de una partícula nuclear, al menos se tienen que visualizar cinco dimensiones, mientras que una imagen sólo tiene dos dimensiones. El problema radica en cómo reducir la complejidad a un grado que no sobreexige al expectador y la vez no omite datos importantes.. Como se mencionó anteriormente los datos a visualizar provienen de diferentes fuentes, las cuales se describen a continuación: Mundo real: Datos generados por instrumentos de medición, tales como tomografías computarizadas o imágenes de satélite. Mundo teórico: Cómputos basados en modelos matemáticos, tales como modelación de moléculas y meteorología. Mundo Artificial: Datos creados por los humanos, por ejemplo en el arte, televisión, filmes.. Dentro de estas fuentes se encuentran ramas específicas, cuyo volumen de información generada varía en el orden de MegaBytes, GigaBytes y TeraBytes como puede apreciarse en la Tabla 1.. 2.
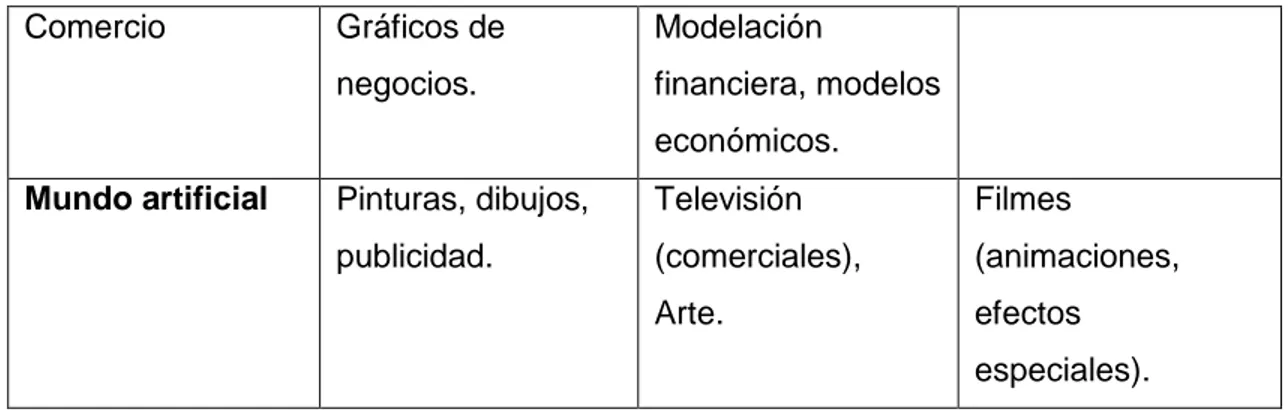
(10) C. A P. Í. T U L. O. 1. La visualización científica ha sido aplicada para resolver diversos problemas provenientes de las fuentes de datos descritas anteriormente. Por ejemplo, la representación de datos multivaluados o la graficación de datos volumétricos son aplicaciones de gran utilidad dentro de la investigación científica en temas tan diversos como la matemática, medicina, ciencias naturales e ingeniería, y se utilizan para representar datos que pueden provenir de sensores, como es el caso de tomógrafos y satélites, o bien de tareas computacionales anteriores, como simulaciones o análisis por elemento finito. Al mismo tiempo, los resultados de la visualización de estos datos no son meramente una representación cuantitativa de los mismos, es decir, no se busca necesariamente la presentación fiel de valores sino un entendimiento global de determinadas propiedades del modelo o de la simulación que produjo los datos. Estos objetivos son sumamente exigentes en términos de tecnología, tanto de hardware como de software. Tabla 1. Clasificación de áreas de aplicaciones.. Mundo real. Megabyte. Gigabyte. Terabyte. Medicina, sistemas. Datos sísmicos,. Física de alta. de información. cristalografía.. energía,. geográfica,. astronomía,. microscopios. aplicaciones. electrónicos.. militares.. Mundo Teórico Ciencia. Movimiento de. Diseño de. moléculas, química. moléculas,. cuántica,. meteorología,. matemáticas.. Simulación de fluidos (fluid simulations CFD).. Ingeniería. Navegación,. Diseño. arquitectura y. (automóviles).. construcción.. 3.
(11) C. Comercio. A P. Gráficos de. Modelación. negocios.. financiera, modelos. Í. T U L. O. 1. económicos. Mundo artificial. Pinturas, dibujos,. Televisión. Filmes. publicidad.. (comerciales),. (animaciones,. Arte.. efectos especiales).. 1.1. Formatos de almacenamiento de datos, técnicas de visualización y aplicaciones más comúnmente utilizadas. Hoy en día el uso de la Visualización Científica se ha extendido a muchos campos de la ciencia, cada uno con sus requerimientos específicos, por lo que se han creado formatos de datos para el almacenamiento de la información y se han desarrollado técnicas y aplicaciones con el objetivo de visualizar sus datos.. Los paquetes de visualización y bibliotecas utilizadas para almacenar y visualizar datos científicos poseen sus propios formatos de datos, con una estructura que le permite acceder a los datos en el fichero, de una forma rápida eficiente. En la Tabla 2 se muestra un resumen de algunas de las características de los formatos más importantes, en los cuales se ha basado la presente investigación. Tabla 2. Formatos de datos más utilizados en la visualización científica.. Extensión. Nombre. Fabricante. Breve Descripción. cdf. Common. NASA/Goddard. Es una abstracción de datos para. Data Format. Space Center. Flight almacenar, manipular y acceder a conjuntos. de. datos. multidimensionales. El componente básico de CDF es una interfaz de programación que tiene una vista del modelo de datos independiente del dispositivo.. 4.
(12) C. HDF, h5. A P. Í. T U L. O. 1. Hierarchical. National Center Es una biblioteca y un formato de. Data Format. for. fichero. multiobjeto. para. la. Supercomputing transferencia de datos numéricos y Applications. gráficos entre máquinas.. (NCSA) nc. Network. Unidata. netCDF (Network Common Data. Common. Program Center. Format) es una interfaz para acceso. Data Format. a datos y una biblioteca que provee una implementación de la interfaz. La biblioteca netCDF también define un formato independiente de la máquina para la representación de datos científicos. La interfaz junto con la biblioteca y el formato soporta la creación, acceso y compartición de datos científicos.. dx, cm. Data Explorer IBM. El. Explorador. de. Datos. para. Format. Visualización de IBM es un sistema de flujo de datos cliente-servidor para la visualización, el mismo está construido sobre un modelo que soporta representaciones generales de campos con una API, lenguaje de alto. nivel,. acceso. a. programas. visuales. Incluye además, soporte para mallas curvilíneas e irregulares y. jerarquías. (ejemplo,. árboles,. series, datos compuestos), datos vector y tensor, etc.. En cuanto a las técnicas de visualización, se puede afirmar que no existe una técnica de visualización que cubra toda la gran variedad de posibles datos científicos. Estas técnicas se pueden clasificar de diferentes formas según sea el. 5.
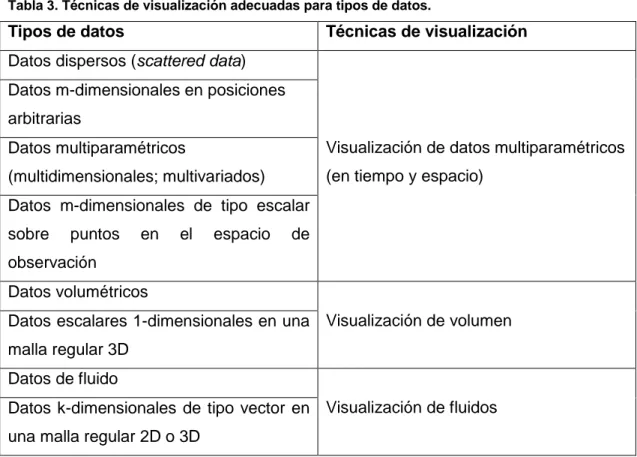
(13) C. A P. Í. T U L. O. 1. tipo de datos, el enfoque y el alcance (puntuales, locales, globales). Existen, sin embargo, técnicas específicas que resultan más adecuadas para clases especiales de datos1, las cuales se relacionan en la Tabla 3. Tabla 3. Técnicas de visualización adecuadas para tipos de datos.. Tipos de datos. Técnicas de visualización. Datos dispersos (scattered data) Datos m-dimensionales en posiciones arbitrarias Datos multiparamétricos. Visualización de datos multiparamétricos. (multidimensionales; multivariados). (en tiempo y espacio). Datos m-dimensionales de tipo escalar sobre. puntos. en. el. espacio. de. observación Datos volumétricos Datos escalares 1-dimensionales en una Visualización de volumen malla regular 3D Datos de fluido Datos k-dimensionales de tipo vector en Visualización de fluidos una malla regular 2D o 3D. Con el objetivo de representar, almacenar y procesar esta información surgen una serie. de. aplicaciones. para facilitar. su. manipulación.. Estas. aplicaciones,. desarrolladas en su mayoría por empresas y universidades, permiten diversas representaciones para muchos tipos de datos, acceso rápido y eficiente a estos y proveen herramientas para la visualización de los mismos. Las aplicaciones que se usan con mayor frecuencia son varias. Dentro de ellas podemos mencionar las siguientes:. 1. Véase el epígrafe 2.1 para una descripción detallada de los tipos de datos empleados en la. visualización científica.. 6.
(14) C. A P. Í. T U L. O. 1. a) Bibliotecas para la creación y manipulación de ficheros de datos científicos para visualización (CDF, netCDF, HDF), las mismas soportan una serie de APIs para el trabajo con este tipo de ficheros. Ver epígrafe 2.2. b) Paquetes de visualización para el trabajo con ficheros de datos científicos para visualización (OpenDX, IRIS Explorer, AVS, otros. Ver Anexos), que soportan trabajo con ficheros de este tipo y además tienen implementadas diferentes técnicas de visualización que pueden ser utilizadas. Ver Tabla 4. Tabla 4. Paquetes de visualización.. Nombre. Contacto. URL. AVS. AVS. http://www.avs.com/. Data Explorer. IBM. http://www.almaden.ibm.com/dx. FAST. Centro de. http://science.nas.nasa.gov/Software/FAST/. Investigaciones NASA/Ames IRIS. Explorer NAG. http://www.nag.co.uk/Welcome_IEC.html. Khoros. Universidad de. http://www.khoros.unm.edu/. Nuevo México Scian. Universidad del. http://www.scri.fsu.edu/~lyons/scian/. estado de Florida VIS-5D. Universidad de. http://www.ssec.wisc.edu/~billh/vis5d.html. Wisconsin. Lo anteriormente expuesto demuestra que en la actualidad existe una gran diversidad de tecnologías de almacenamiento de datos, paquetes y técnicas de visualización empleados en la visualización científica. A partir de aquí se proponen para el presente trabajo las siguientes preguntas de investigación.. 1. ¿Es posible clasificar los datos que se utilizan en la visualización científica de manera, que a partir de esta clasificación pueda seleccionarse una tecnología de almacenamiento adecuada para un tipo de dato y una aplicación específicos?. 7.
(15) C. A P. Í. T U L. O. 1. 2. ¿Es posible realizar una comparación entre los diferentes métodos de almacenamiento. de. datos. utilizados. en. la. visualización. científica,. independientemente del tipo de dato y el área de aplicación específica a la que pertenezcan tales datos? 3. ¿Resulta conveniente almacenar los datos empleados en la visualización científica en una base de datos relacional? 4. ¿Cuáles son las ventajas del uso del paquete OpenDX para la visualización científica?. Resultan entonces las siguientes hipótesis de investigación para este trabajo.. 1. A partir de un estudio y caracterización de las diferentes clasificaciones de los datos que se emplean en la visualización científica, es posible seleccionar la tecnología que resulta más apropiada para almacenar un tipo de datos y una aplicación específicos. 2. Debido a que existen varias tecnologías de almacenamiento y organización de los datos científicos se puede establecer una comparación entre los diferentes métodos de almacenamiento de datos sin tener en cuenta el tipo de dato y el área de aplicación a la cual pertenece. 3. A pesar de que las bases de datos relacionales son ampliamente utilizadas en el proceso de almacenamiento y manipulación de información debido a sus potencialidades de organización e indexado, estas no son apropiadas para almacenar y procesar datos científicos. 4. El paquete de visualización OpenDX es una interfaz gráfica para el trabajo con datos científicos, que permite crear programas visuales utilizando módulos internos, los cuales tienen implementadas técnicas de visualización que pueden ser utilizadas sin necesidad de programarlas; sin embrago, el mismo. provee. una. interfaz. interna. de. programación. para. hacer. modificaciones que no estén disponibles a nivel de módulos, brinda la posibilidad de controlar la apariencia de un objeto, teniendo como uno de sus rasgos más significativos la posibilidad de importar diferentes formatos de datos científicos (HDF, CDF, netCDF) y ficheros binarios y ASCII con diferentes estructuras internas.. 8.
(16) C. 1.2. A P. Í. T U L. O. 1. Objetivo general:. Realizar un estudio comparativo de las diferentes tecnologías que se utilizan en la visualización científica para el almacenamiento de datos y las técnicas de visualización que resultan adecuadas para cada tipo de dato. 1.3. Objetivos específicos: 1. Estudiar las diferentes clasificaciones de tipos de datos científicos existentes y seleccionar una como base para la realización de una valoración crítica de los métodos de almacenamiento de datos y las técnicas de visualización empleados en la visualización científica. 2. Valorar los diferentes métodos de almacenamiento de datos usados en la visualización científica. 3. Estudiar y clasificar las técnicas actuales de visualización empleadas en la visualización científica. 4. Valorar la conveniencia del uso de bases de datos relacionales para el almacenamiento de los datos que se utilizan en la visualización científica. 5. Estudiar el paquete de visualización “OpenDX” y describir las ventajas y desventajas de su uso para la visualización científica.. 9.
(17) C. 2.. MODELACIÓN. DE. DATOS. A P. Í. T U L. PARA. O. 2. LA. VISUALIZACIÓN CIENTÍFICA En este capítulo se hace un análisis de la modelación de los datos para la visualización, proceso que permite identificar un conjunto de datos de otro y darle una estructura adecuada a los datos a visualizar.. Además se muestra una descripción de los diferentes formatos de almacenamiento de datos utilizados en la visualización científica y de sus interfaces. También se realiza un análisis del uso de bases de datos relacionales para almacenar estos datos.. Por último se hace una comparación entre los formatos de almacenamiento de datos en cuanto a una serie de parámetros con el objetivo de mostrar las potencialidades que caracterizan a cada uno independientemente.. 2.1 Modelos de Datos para la Visualización Científica La modelación de datos constituye un aspecto sumamente importante dentro de la visualización para lograr construir un conjunto de datos bien organizado, que pueda brindar mediante su visualización la mejor calidad de información. El objetivo de la especificación de datos es proporcionar las propiedades de un conjunto de datos para los diferentes procesos que intervienen en la visualización. Existen diferentes categorías de propiedades pertenecientes a los datos (metadatos), las cuales son interesantes para el proceso de visualización [4], [2]. Una de las más importantes para este proceso es la de “Metadatos-Atributos”. Esta categoría caracteriza los datos y dentro de ella se encuentran las características que describen el conjunto de datos y el espacio de definición.. Uno de los primeros enfoques para una especificación general de datos, es en el que se consideran los datos de aplicación como elementos m-dimensionales definidos en una malla k-dimensional, y utilizan para ello la notación: 10.
(18) C. A P. Í. T U L. O. 2. Lkm . Esta notación proporciona una clasificación tangible de los datos y es fácil de manipular. Algunas clases de datos comunes se pueden describir como sigue: – Multiparamétricos:. Lkm , para m ≥ 2.. – No estructurados:. L0m , para m ≥ 1.. – Volumétricos: – Fluidos:. L13 , donde cada elemento es un escalar.. L1k , para k ≤ 3, donde cada elemento es un vector.. Sin embargo, esta notación no tiene en cuenta algunas propiedades importantes del conjunto de datos. Por ejemplo, no se considera el tipo de los elementos. Así, datos volumétricos y de fluido sobre una malla tridimensional, no son distinguibles sin información adicional. Tampoco es posible la descripción de datos heterogéneos.. Considerando estos aspectos [6] define un espacio para los datos denominado “underlying field”, de donde se extrae la entidad E a visualizar. E es una función que está especificada por su dominio e imagen. El dominio de la función está dado por las variables independientes, y la imagen abarca las variables dependientes. Las variables independientes están descritas por su dimensionalidad y radio de acción, y las dependientes por su dimensionalidad y tipo. Así, por ejemplo,. E 3V 3 representa una magnitud vectorial de tres componentes definida en un dominio tridimensional, y. E52S representa 5 magnitudes escalares definidas en regiones limitadas de un dominio bidimensional. Con esta notación se pueden identificar unívocamente los datos volumétricos y de fluido, y también se pueden describir conjuntos de datos heterogéneos, como, por ejemplo, con: 3 E23SV . 11.
(19) C. A P. Í. T U L. O. 2. representa 2 magnitudes escalares y una magnitud vectorial de tres componentes definidas en regiones limitadas de un dominio tridimensional.. En la notación, los corchetes son utilizados para indicar el radio de acción, también se utilizan paréntesis y llaves para indicar los distintos tipos de radios de acción.. En otros enfoques para la especificación de datos, se intenta considerar muchas características del conjunto de datos. En general, mientras más características de los datos se consideren en su especificación, más información estará disponible para una rápida evaluación y para la visualización. Sin embargo, una forma notacional de este tipo, rápidamente se torna confusa e inmanejable para algunas aplicaciones especiales.. Otro enfoque es el que considera que los datos del mundo real, teórico y artificial están localizados en puntos en un espacio n-dimensional llamado el espacio de observación.. Aquí debe distinguirse entre las dimensiones que definen el espacio de observación y los parámetros (atributos) que son observados/medidos en el espacio de observación. Debe distinguirse, además, entre las variables dependientes e independientes.. Para la caracterización de los puntos de observación (ejemplo, puntos en el espacio de observación donde los datos están disponibles) necesitamos conocer lo siguiente: Dimensionalidad del espacio de observación. Área de impacto en el espacio (puntual, local, global). Conectividad de los datos. Dimensionalidad del espacio de observación: Las dimensiones que definen el espacio de definición conforman las variables independientes. Las mismas pueden estar definidas de manera discreta o continua. Por ejemplo, en la literatura la clasificación “running clock” se corresponde con la. 12.
(20) C. A P. Í. T U L. O. 2. definición continua de la variable “tiempo”, y en caso contrario con la clasificación “epoch-based”.. Para más de dos dimensiones debe realizarse una proyección del espacio de definición para la visualización, en tal caso pueden ocurrir solapamientos e interpretaciones ambiguas.. Área de impacto en el espacio: Especifica la región en que tienen validez los valores tomados en un punto. Dichas regiones se clasifican en tres grupos. – Impacto puntual: Los datos tienen validez solo en el punto de observación correspondiente (ejemplo, valores de datos en un punto de una laguna). – Impacto local: Los datos tienen validez en una cierta vecindad del punto de observación correspondiente (ejemplo, datos en la parte norte de la laguna). – Impacto global: Los datos tienen validez en todo el espacio de observación (ejemplo, toda el área que abarca la laguna).. Si el impacto es solamente en unos puntos, los valores de datos deben ser asignados a sólo a esos puntos. En la Figura 1 se muestra la visualización de un conjunto de datos que tienen impacto puntual.. 13.
(21) C. A P. Í. T U L. O. 2. Figura 1 Visualización de datos con impacto puntual.. Si el impacto es global, los valores de datos en la representación gráfica son asignados a toda el área.. Si el impacto es local, se pueden inferir los datos cercanos al punto de observación desde el punto de observación.. Para definir áreas locales, sean p i los puntos de observación en el espacio de observación.. Existen dos formas de modelar el impacto local: Diagrama de Voronoi El diagrama de Voronoi (Dirichlet tesselation) es una única partición del espacio de observación donde cada región contiene un punto de observación. A cada región se le asigna el valor de dato de su punto de dato, como se muestra en la Figura 2.. 14.
(22) C. A P. Í. T U L. O. 2. Interpolación de datos dispersos Una función de interpolación con bases locales es utilizada. Esta función encierra un valor de datos para cada punto del espacio de observación. Diagrama de Voronoi Def.: El diagrama de Voronoi (Dirichlet tesselation) es una partición del espacio de observación n-dimensional en regiones G j con las siguientes propiedades: Cada punto p j está en la región G j . Además, G j consiste en todos los puntos x R n que se encuentran a menor distancia de p j que de cualquier otro punto de observación p k con j k .. G j {x R n : // x p j // // x pk //, j k}. Figura 2 Diagrama de Voronoi en 2D, Hagemann 1995.. Interpolación de datos dispersos Formulación para espacio de observación 2D.. 15.
(23) C. A P. Í. T U L. O. 2. Dado un conjunto pi ( xi , yi , f i ) con i 1,2,, n , buscamos una función bivariada. F ( x, y) con F ( xi , yi ) f i para i 1,2,, n . Los puntos de observación pueden tener cualquier distribución.. Existe una variedad de métodos de interpolación de datos dispersos. Uno de ellos es la aproximación Shepard (Shepard approach). En la Figura 3 puede apreciarse este tipo de interpolación. La misma obtiene los valores de los puntos cercanos mediante la siguiente función: n. F ( x, y ) . W k 1. k. ( x, y ) f k. n. W k 1. k. ( x, y ). La función de peso Wk ( x, y) es construida de tal forma que el impacto de un punto de observación decrece al alejarse del punto de observación, i.e.. Wk ( x, y) d k ( x xk ) 2 ( y y k ) 2. Para esta elección de Wk , todos los valores de datos tienen impacto global.. Para obtener un impacto local se puede utilizar la función de peso Franke Little mostrada a continuación.. (R d k ) Wk ( x, y ) R dk. con R d k . 2. R d k si R d k 0 en otro caso. 16.
(24) C. A P. Í. T U L. O. 2. Figura 3 Interpolación Shepard, Hagermann 1995.. Conectividad de los datos Los puntos de observación pueden estar organizados de acuerdo a diferentes criterios, especialmente si es necesario realizar cálculos, como es el caso de la visualización de fluidos. Para la descripción de esta organización se utilizan mallas, que establecen la relación entre los puntos. Cada punto de la malla se corresponde con un punto de observación.. Diferentes autores utilizan criterios diferentes para clasificar los tipos de mallas, que no siempre son uniformes. En [3] se diferencia fundamentalmente entre mallas estructuradas y no estructuradas, en dependencia de si la relación entre los puntos de la malla está dada de forma implícita o explícita. Otros utilizan criterios similares para distinguir entre mallas regulares e irregulares. Teniendo en cuenta los. 17.
(25) C. A P. Í. T U L. O. 2. conceptos empleados en los sistemas de visualización actuales, distinguimos los siguientes tipos de mallas: – Regulares: La malla está definida por líneas paralelas a los ejes de coordenadas y la distancia entre ellas es constante. – Irregulares o no estructuradas: La malla está definida por líneas de cualquier tipo con cualquier distancia entre ellas. Esto implica que tanto las coordenadas como la relación entre los puntos de la malla deben especificarse de forma explícita. – Estructuradas por bloque: Definida por líneas paralelas a los ejes de coordenadas con diferentes distancias entre ellas. – Estructuradas: Definida por líneas de cualquier tipo con puntos equidistantes. Si las líneas son curvas paramétricas, entonces la malla se denomina curvilínea. – Híbridas: Compuesta por la unión de diferentes tipos de mallas. Los distintos tipos de mallas se muestran en la Figura 4.. Figura 4 Tipos de mallas.. 18.
(26) C. A P. Í. T U L. O. 2. Los modelos descritos anteriormente para la especificación de datos no toman en cuenta cómo se almacenan físicamente los datos y sus propiedades. En la práctica este aspecto no puede obviarse. Por ello juega un papel decisivo para el almacenamiento consistente de los datos con sus metadatos y la elección de un formato uniforme.. Para estos fines, existen diferentes formatos de datos que son utilizados en la visualización científica. Los más importantes son: CDF, HDF, netCDF y DX, siendo este último, el formato propio del sistema de visualización de IBM: Data Explorer.. 2.2 Formatos de Datos utilizados en la Visualización Científica A continuación se describen los principales formatos de datos utilizados en la visualización científica. 2.2.1 HDF HDF significa Hierarchical Data Format y es desarrollado por el Centro Nacional de Apliaciones de Supercómputo (National Center for Supercomputing Applications, NCSA) en la Universidad de Illinois. Es una biblioteca y un formato de fichero multiobjeto para la transferencia de datos gráficos y numéricos entre máquinas. HDF se encuentra disponible de forma gratis. La distribución consiste en la biblioteca HDF, las utilidades de línea de comando HDF y una suite de prueba (código fuente solamente). Niveles de interacción de HDF: En la Figura 5 se muestran los niveles de interacción de HDF. En su nivel más bajo es un formato de fichero físico para el almacenamiento de datos científicos. En su nivel más alto es una colección de utilidades y aplicaciones para manipular, ver y analizar datos en los ficheros HDF. Y entre esos niveles HDF una biblioteca de programas que provee APIs de alto nivel y una interfaz de datos de bajo nivel.. 19.
(27) C. A P. Í. T U L. O. 2. Figura 5 Niveles de interacción de HDF.. Aplicaciones generales En el nivel más alto hay utilidades de línea de comandos de HDF, aplicaciones de NCSA que soportan visualización de datos y análisis, y variedad de aplicaciones de terceros desarrolladores.. Existen utilidades de línea de comandos de HDF para: . Convertir de un formato a otro (por ejemplo, desde y hacia JPEG/HDF).. . Analizar y ver ficheros HDF (siendo hdp una de las herramientas más útiles).. . Manipular los ficheros HDF.. De las utilidades HDF, la “hdp” es una de las más importantes. A continuación se ofrece una descripción de su función.. Provee información rápida sobre los contenidos y objetos de datos en un fichero HDF. Puede listar los contenidos de los ficheros HDF en varios niveles con diferentes detalles. Además, puede también vaciar los datos de uno o más ficheros en el fichero, en formato binario o ASCII.. Las otras utilidades son: . gif2HDF – convierte imágenes GIF en imágenes HDF GR.. 20.
(28) C. A P. Í. T U L. O. 2. . HDF24to8 – convierte imágenes de 24-bit a imágenes de 8-bit.. . h4cc – compila un programa HDF4 escrito en lenguaje C.. . h4fc – compila un programa HDF4 escrito en lenguaje Fortran 90.. . h4redeploy – actualiza caminos en h4cc/h4fc después que los binarios precompilados HDF4 han sido instalados en una nueva localización.. . HDF2gif – convierte imágenes HDF a imágenes GIF.. . HDF2jpeg – convierte imágenes de puntos HDF a imágenes jpeg.. . HDF8to24 – convierte imágenes de 8-bit a imágenes de 24-bit.. . HDFcomp – re-comprime un fichero de puntos HDF de 8-bit.. . HDFed – editor de ficheros HDF, requiere conocimiento avanzado de HDF.. . HDFimport – importa datos ASCII hacia HDF.. . HDFls – lista información básica acerca de un fichero HDF.. . HDFpack – comprime un fichero HDF mediante la eliminación de espacio en desuso que ha sido creado debido a las modificaciones de fichero.. . HDFtopal – extrae una paleta de un fichero HDF.. . HDFtor8 – extrae imágenes de puntos de 8-bit y paletas de un fichero HDF.. . HDFunpac – descomprime un fichero HDF mediante la exportación de elementos de datos científicos (DFTAG_SD) hacia elementos de objetos externos.. . hdiff – compara dos ficheros HDF y reporta las diferencias.. . hrepack – copia un fichero HDF hacia un nuevo fichero con/sin compresión y/o fragmentación (chunking).. . jpeg2HDF – convierte imágenes jpeg a imágenes raster HDF.. . paltoHDF – convierte una paleta en bruto (raw palette) a HDF.. . r8toHDF – convierte imágenes de puntos de 8-bit a HDF.. . ristosds – convierte una imagen a un SDS.. . vmake – crea vsets.. . vshow – extrae vsets de un archivo HDF.. Además están incluidas las utilidades de netCDF, ncdump y ncgen, que han sido compiladas con la biblioteca HDF.. 21.
(29) C. A P. Í. T U L. O. 2. Interfaces de programación de aplicaciones Estas incluyen conjuntos de rutinas para el almacenamiento y acceso a tipos de datos específicos. A pesar de que cada interfaz (Application Programming Interface o API) requiere programación, todos lo detalles de bajo nivel pueden ser ignorados. Estas se encuentran disponibles en C y Fortran. Los tipos de estructura de datos que soporta HDF son Conjuntos de datos científicos (Scientific Data Sets, SDS and DFSD APIs), Imágenes de puntos (Raster Images, General, 8-bit, 24-bit APIs), paletas de colores, entradas de texto, y Vdatas y Vgroups. La interfaz de dos niveles Esta interfaz está reservada para desarrolladores de software. Fue diseñada para E/S de flujo de datos directamente desde archivos, manipulación de errores, administración de memoria y almacenamiento físico. Es esencialmente un conjunto de herramientas de software para programadores experimentados, quienes desean que HDF haga algo más que lo que está actualmente disponible a través de la interfaz de alto nivel. Las rutinas de bajo nivel están disponibles sólo en C.. Compresión de datos HDF 4.0 (y versiones posteriores) soportan una interfaz de compresión de bajo nivel, la cual permite a que cualquier objeto de dato sea comprimido utilizando una variedad de algoritmos.. Actualmente solo tres algoritmos de compresión están soportados: Run-Length Encoding (RLE), Huffman adaptativo, y un codificador de diccionario LZ-77 (el algoritmo de decodificación de gzip). Planes para algoritmos futuros incluyen un codificador de diccionario Lempel/Ziv-78, un codificador aritmético y un algoritmo rápido de Huffman.. HDF 4.0 (y entregas posteriores) soportan compresión de n-bit para SDS, RLE (Run Length Encoding), IMCOMP, y compresión JPEG para imágenes de puntos.. 22.
(30) C. A P. Í. T U L. O. 2. Nuevo con HDF 4.1 es el soporte para fragmentación (chunking) y fragmentación con compresión. La fragmentación de datos permite a un SDS n-dimensional o imagen GR ser almacenadas como series de fragmentos n-dimensionales2.. Con HDF4.2r0, HDF soporta compresión SZIP. Para información adicional ver SZIP Compression in HDF Products.. El fichero HDF Un fichero HDF contiene un encabezamiento de fichero, al menos un bloque descriptor de datos, y cero o más elementos de datos, como se describe en la Figura 6.. Figura 6 Esquema físico de un fichero HDF que contiene un objeto de dato.. El encabezamiento de fichero identifica el fichero como un fichero HDF. Un bloque descriptor de datos contiene un número de descriptores de datos. Un descriptor de 2. Para más información véase la Guía de Usuario de HDF.. 23.
(31) C. A P. Í. T U L. O. 2. datos y un elemento de dato juntos, conforman un objeto de dato, el cual es la estructura básica de agrupamiento para el encapsulamiento de datos en un fichero HDF. Cada uno de estos términos es descrito en las secciones siguientes.. El fichero HDF posee una serie de rasgos que se mencionan a continuación. . Es versátil. HDF suporta muchos tipos diferentes de modelos de datos. Cada modelo de dato define un conjunto específico de tipo de dato y provee una API para lectura, escritura y organización de datos y metadatos del tipo correspondiente. Los modelos de datos soportados incluyen arreglos multidimensionales, imágenes de puntos, y tablas.. . Es auto-descriptivo, permitiendo una aplicación para interpretar la estructura y contenidos de un fichero sin ninguna información proveniente del exterior.. . Es flexible. Con HDF, se pueden mezclar y asociar objetos relacionados agrupados en un fichero y acceder a ellos como un grupo o como objetos individuales. Los usuarios pueden además crear sus propias estructuras de agrupamiento utilizando un rasgo de HDF llamado vgroups.. . Es extensible. Puede acomodar fácilmente nuevos modelos de datos, sin tener en cuenta si fueron adicionados por el equipo de desarrolladores de HDF o por los usuarios de HDF.. . Es portable. Los ficheros HDF pueden ser compartidos a través de la mayoría de las plataformas comunes, incluyendo muchas estaciones de trabajo y computadoras de alto desempeño. Un fichero HDF creado en una computadora puede ser leído en un sistema diferente sin modificación alguna.. 2.2.2 CDF El Common Data Format (CDF) es un formato de datos auto-descriptivo para el almacenamiento y manipulación de datos escalares y multidimensionales en una forma independiente de la plataforma y el campo de especialidad. A pesar de que CDF tiene su propio formato interno auto-descriptivo, éste consiste en algo más que sólo un formato de datos. CDF es un paquete de administración de datos científicos (conocido como la “Biblioteca CDF") la cual permite a los programadores y 24.
(32) C. A P. Í. T U L. O. 2. desarrolladores de aplicaciones administrar y manipular arreglos de datos escalares, vectoriales y multidimensionales. Contenidos de un fichero dotCDF del CDF V3.0 El fichero dotCDF3 contiene un número mágico y dos o más registros internos (RIs) que son utilizados para organizar los contenidos de un CDF. Diferentes tipos de registros internos (Tabla 5) son utilizados para almacenar información sobre varios aspectos y/u objetos en el CDF. Cada registro interno contiene dos o más campos. El primer campo (en el desplazamiento 0x0 del registro interno), referido como el campo RecordSize, es un entero sin signo de 8 bytes conteniendo el tamaño en bytes del registro interno. El segundo campo (en el desplazamiento 0x4 del registro interno), referenciado como el campo RecordType, es un entero con signo de 4 bytes conteniendo el tipo de registro interno.. Todos los ficheros dotCDF contienen un CDF Descriptor Record (CDR) y un Global Descriptor Record (GDR). Otros registros internos estarán presentes dependiendo de los contenidos del CDF. El CDR se encuentra siempre en el desplazamiento4 0x0000000000000008 del fichero, el cual sigue inmediatamente al número mágico o los números mágicos mencionados anteriormente. El desplazamiento dentro del fichero del GDR es almacenado en el CDR.. El único registro interno en una localización fija en el fichero dotCDF es el CDR. Todos los otros registros internos (incluyendo el GDR) pueden estar presentes en cualquier orden (el cual depende, generalmente, del orden en que fueron creados por la aplicación los contenidos del CDF). Los desplazamientos dentro del fichero son utilizados para “apuntar” a otros registros internos. Las listas enlazadas de registros internos son implementadas mediante el almacenamiento del desplazamiento dentro del fichero del primer. 3. dotCDF se refiere al fichero de datos .CDF. 4. El desplazamiento en (hexadecimal) bytes desde el inicio del fichero.. 25.
(33) C. A P. Í. T U L. O. 2. registro interno en la lista enlazada, habiendo almacenado ese registro interno, el desplazamiento dentro del fichero del próximo registro interno y así sucesivamente.. La Figura 7 muestra un posible orden de registros internos en un fichero dotCDF descomprimido. Note que el GDR apunta al primer zVDR el cual, a su vez apunta al próximo zVDR. Los desplazamientos dentro del fichero son utilizados para implementar esta lista enlazada. Los registros internos pueden ser ordenados en diferentes formas, dependiendo de cómo el CDF fue escrito por la aplicación. La Figura 8 muestra un posible orden de los registros internos en un fichero dotCDF, que tiene una variable comprimida. La Figura 9 muestra el orden de los registros internos en un fichero dotCDF completamente comprimido. Tabla 5 Registros internos.. Tipo de registro interno. Campo. Propósito/Contenido del valor interno. RecordType CDR. 1. CDF Descriptor Record (Información general sobre el CDF). GDR. 2. Global Descriptor Record (Información general adicional sobre el CDF). rVDR. 3. rVariable Descriptor Record (Información sobre una rVariable). ADR. 4. Attribute Descriptor Record (Información sobre un atributo). AgrEDR. 5. Attribute g/rEntry Descriptor Record (Información sobre una gEntry o rEntry de un atributo). VXR. 6. Variable Index Record (Información de indexado para una variable). VVR. 7. Variable Values Record (Uno o más registros variables). zVDR. 8. zVariable Descriptor Record (Información de una zVariable). AzEDR. 9. Attribute Entry Descriptor Record (Información sobre una sobre una zEntry de un atributo). CCR. 10. Compressed CDF Record (Información sobre un CDF comprimido o una variable comprimida). 26.
(34) C. CPR. 11. Compression. Parameters. A P. Í. T U L. Record. O. 2. (Información. sobre la compresión utilizada para un CDF o una variable) SPR. 12. Sparseness Parameters Record (Información sobre el arreglo sparseness especificado). CVVR. 13. Compressed Variable Values Record (Información para el CDF comprimido o la variable comprimida). UIR. -1. Unused Internal Record (Un registro interno que no se está utilizando actualmente). 27.
(35) C. A P. Í. T U L. O. 2. Figura 7 Ejemplo de ordenamiento para un fichero dotCDF.. 28.
(36) C. A P. Í. T U L. O. 2. Figura 8 Ejemplo de ordenamiento para un fichero dotCDF con una Variable Comprimida.. 29.
(37) C. A P. Í. T U L. O. 2. Figura 9 Ejemplo de un ordenamiento de un fichero dotCDF Completamente Comprimido.. 2.2.3 netCDF The Network Common Data Form o netCDF, es una interfaz a una biblioteca de funciones de acceso a datos diseñada para el almacenamiento y recuperación de datos en forma de arreglos, donde cada arreglo es una estructura rectangular ndimensional (con n 0,1,2 ) en la que todos sus elementos son del mismo tipo de dato (ejemplo: caracteres de 8-bit, entero de 32-bit) y un escalar (un solo valor) equivale a un arreglo 0-dimensional.. La biblioteca netCDF implementa un tipo de datos abstracto, por lo que todas las operaciones para acceder y manipular datos en un conjunto de datos de netCDF, deben usar sólo las funciones que provee la interfaz. La representación de los datos está oculta para aplicaciones que usen la interfaz, así que la forma en la cual estos se encuentran almacenados puede ser cambiada sin afectar los programas existentes. La representación física de los datos de formato netCDF esta diseñada para ser independiente de la computadora en que se almacenen.. Los datos son vistos como una colección de objetos portables y auto-descriptibles que pueden ser accedidos a través una simple interfaz. Se puede acceder directamente a sus valores, sin conocer detalles sobre la forma en que se encuentran almacenados. Las informaciones auxiliares sobre los datos, tales como, las Units que utilizaron, se pueden almacenar conjuntamente con los datos. Las Utilidades Genéricas (Generic utilities) y los programas de aplicación pueden acceder a conjuntos de datos en netCDF y transformar, combinar, analizar o mostrar. 30.
(38) C. A P. Í. T U L. O. 2. campos específicos de los datos. El desarrollo de tales aplicaciones ha dado lugar a una mejor accesibilidad de datos y una mejor reutilización de software para la administración, análisis y visualización de datos orientados a arreglos.. Unidata soporta las interfaces netCDF para C, FOTRAN 77, FORTRAN 90 y C++. La biblioteca netCDF está soportada para varios sistemas operativos UNIX y tiene también disponible un puerto de Microsoft Windows. El programa fue también portado hacia otros sistemas operativos y probado en los mismos, asistidos por usuarios con acceso a esos sistemas, antes de cualquier lanzamiento mayor.. El programa netCDF de Unidata está disponible vía FTP para alentar a su uso generalizado. (ftp://ftp.unidata.ucar.edu/pub/netCDF). Desempeño de netCDF Una de las metas de netCDF es soportar acceso eficiente a pequeños subconjuntos de grandes conjuntos de datos. Para lograr esto, netCDF utiliza accesos directos en lugar de accesos secuenciales. Esto puede ser mucho más eficiente cuando el orden en que los datos son leídos es diferente del orden en que fueron escritos, o cuando debe ser leído en diferentes órdenes para distintas aplicaciones.. La cantidad de costo para la representación externa portable depende de muchos factores, incluyendo el tipo de dato, el tipo de computadora, la granularidad del acceso a datos, y de cuan bien se ha ajustado la implementación a la computadora en la que está corriendo. Este costo es típicamente pequeño en comparación con todo el conjunto de recursos utilizados por una aplicación. De cualquier forma, el costo de la capa de representación externa, es usualmente, un precio razonable a pagar por acceso de datos portables.. A pesar de que la eficiencia en el acceso a datos ha sido un asunto importante en el diseño e implementación de netCDF, todavía es posible utilizar la interfaz netCDF para acceder a datos de formas ineficientes: por ejemplo, mediante la petición de una porción de datos que requiere un sólo valor de cada registro.. 31.
(39) C. A P. Í. T U L. O. 2. El en formato de dato HDF5 las operaciones con metadatos tienen un costo mayor que las operaciones con datos. Partes del fichero netCDF clásico Un conjunto de datos de netCDF clásico o de desplazamiento de 64 bit es almacenado como un sólo fichero que consiste en dos partes: . Un encabezado (header), conteniendo toda la información sobre las dimensiones, atributos y variables, excepto para los datos de variable.. . Una parte de datos, que comprende los datos de tamaño fijo (datos para variables que no poseen una dimensión ilimitada) y los datos de tamaño variable (datos para variables que presentan una dimensión ilimitada).. El encabezado y las partes de datos son representados en una forma independiente de la arquitectura de la máquina. Esta forma es muy similar a XDR5 (eXternal Data Representation), extendida para soportar almacenamiento eficiente de arreglos de datos no byte (non-byte data).. El encabezado, al principio del fichero contiene información sobre las dimensiones, variables, y atributos en el fichero, incluyendo sus nombres, tipos, y otras características. La información sobre cada variable incluye el desplazamiento al inicio de los datos de la variable para variables de tamaño fijo, o el desplazamiento relativo de otras variables dentro de un registro. El encabezado también contiene longitudes de dimensión e información necesaria para trasladar (map) índices multidimensionales de cada variable hacia los desplazamientos apropiados dentro del fichero.. Por defecto, este encabezado tiene poco espacio extra para uso posterior pues posee solamente el tamaño necesario para las dimensiones, variables y atributos (incluyendo todos los valores de atributos) del conjunto de datos y una pequeña. 5. XDR es una biblioteca de rutinas que permite a un usuario transferir datos entre. computadoras que tienen diferentes convenciones de almacenamiento.. 32.
(40) C. A P. Í. T U L. O. 2. cantidad de espacio extra que se obtiene de redondear el tamaño que hay hasta bloque más cercano del disco. Esto presenta la ventaja de que los ficheros netCDF son compactos, requiriendo un costo pequeño para almacenar los datos auxiliares que hacen auto-descriptivos a los conjuntos de datos. Una desventaja de esta organización es que cualquier operación que demande crecimiento en el encabezamiento (o menos probable, decrecimiento) en un conjunto de datos de netCDF (por ejemplo, adicionando nuevas dimensiones o nuevas variables) requiere de movimiento de los datos mediante copia de los mismos. Se incurre en este gasto cuando se hace una llamada a la función enddef: nc_enddef en C (ver sección “nc_enddef” en la Guía de Interfaz en C de netCDF) y NF_ENDDEF en Fortran (ver sección “NF_ENDDEF” en la Guía de la Interfaz Fortran 77 de netCDF), después de una llamada previa a la función redef: nc_redef en C (ver sección “nc_redef” en la Guía de Interfaz en C de netCDF) o NF_REDEF en Fortran (ver sección “NF_REDEF” en la Guía de la Interfaz Forran 77 de netCDF).. Si se crean todas las dimensiones necesarias, variables y atributos antes de escribir los datos, se evita adiciones posteriores y renombramientos de componentes de netCDF que requieran más espacio en la parte de encabezado del fichero, entonces puede evadirse el costo asociado al cambio posterior del encabezamiento.. Se puede usar una versión alternativa de la función enddef con dos caracteres subrayado en lugar de uno, para explícitamente, reservar espacio extra en el encabezamiento del fichero cuando es creado: en C nc__enddef (ver sección “nc__enddef” en la Guía de Interfaz en C de netCDF), en Fortran NF__ENDDEF (ver sección “NF__ENDDEF” en la Guía de la Interfaz Fortran 77 de netCDF), después de una llamada previa a la función redef. Esto permite reservar suficiente espacio extra en el encabezamiento para acomodar cambios anticipados tales como, la adición de nuevos atributos o la extensión de una cadena de atributos existente, con el objetivo de que soporte cadenas más largas y evitando así, el costo de mover todos los datos posteriormente.. Cuando el tamaño del encabezamiento es modificado, los datos en el fichero son movidos y cambia la localización de los valores de datos contenidos en él. Si otro. 33.
(41) C. A P. Í. T U L. O. 2. programa está leyendo el conjunto de datos de netCDF durante la redefinición, su vista del fichero estará basada en índices antiguos, probablemente incorrectos. Si los conjuntos de datos de netCDF están compartidos durante la redefinición, debe estar provisto algún mecanismo externo a la biblioteca netCDF que evite el acceso de los lectores y provoque que los mismos hagan una llamada a nc_sync/NF_SYNC antes de cualquier acceso posterior.. Las partes de datos con tamaño fijo que siguen al encabezamiento contienen todos los datos de variables para las variables que no presentan una dimensión ilimitada. Los datos para cada variable son almacenados contiguamente en esta parte del fichero. Si no hay variables con dimensión ilimitada, esta es la última parte del fichero netCDF.. La parte de datos de registro que le sigue a los datos de tamaño fijo y consiste en un número variable de registros de tamaño fijo, que cada uno contiene datos para todas las variables de tipo registro. Los datos registro para cada variable son almacenados contiguamente en cada registro.. El orden en que aparecen los datos de la variable en cada sección de datos, es el mismo en que fueron definidas: un orden numérico incremental por el ID de variable de netCDF. Este conocimiento puede ser utilizado a veces para mejorar el desempeño de acceso a datos, puesto que el mejor acceso a datos es actualmente logrado mediante la lectura y escritura de datos en orden secuencial.. 2.2.4 DX EL IBM Visualization Data Explorer (o simplemente DX) es un paquete de software de propósitos generales para la visualización y análisis de datos que permite la exploración visual de grandes cantidades de datos (pueden ser dependientes del tiempo) provenientes de experimentos (o simulaciones) científicos o ingenieriles.. Está diseñado para ser utilizado tanto por usuarios finales como por programadores y posee una interfaz gráfica que permite a los usuarios finales introducir datos y ajustar los valores de entrada a procesos de visualización existentes. Un usuario 34.
(42) C. A P. Í. T U L. O. 2. ligeramente sofisticado puede crear sus propios procesos de visualización construyendo una red de módulos. Y un programador experto puede crear sus propios módulos para luego introducirlos en un programa visual.. El DX posee una amplia gama de módulos que pueden ser utilizados para visualizar los datos. Estos módulos basan su interoperabilidad en el modelo de datos que se utiliza de fondo.. El DX está construido según el modelo cliente-servidor y sobre los sistemas abiertos TCP/IP, sockets, X Windows System y Motif. Por esto los usuarios programadores pueden construir módulos (en C o FORTRAN) que se ajusten a sus requerimientos e incluso, se salgan de los estándares.. El modelo cliente-servidor del DX está formado por cuatro capas (Figura 10): las capas de Ventanas e Interfaz de Usuarios conforman la parte cliente mientras que las capas Ejecutivo, Módulos y de Manejo de Datos conforman el servidor. Las capas de la parte servidor permiten trabajar de manera distribuida en varias máquinas de una red. El usuario puede interactuar con cualquiera de las cuatro capas.. Figura 10 Capas del modelo Cliente-Servidor.. 35.
(43) C. A P. Í. T U L. O. 2. Esta estructura del Data Explorer permite que se ejecuten diferentes partes del proceso de visualización en diferentes plataformas. El Data Explorer se puede ejecutar actualmente en las plataformas IBM RISC/6000, Sun y SGI, y está diseñado. para. aprovechar. al. máximo. las. capacidades. de. sistemas. multiprocesadores. Interfaz de usuario La interfaz gráfica esta construida sobre los estándares X Windows System y Motif. Esto permite el uso de múltiples ventanas para el control, y edición de la visualización. Esta interfaz tiene dos niveles de servicios: . Usuarios finales: Permite la ejecución de programas visuales previamente creados con una amplia gama de interactores como: menús, diales, botones deslizantes, etc.. . Programadores: Permite la creación y edición de programas visuales basado en el paradigma de programación visual, es decir, mediante la interconexión gráfica de módulos.. En ambos niveles de servicios se tiene acceso a la ayuda en línea, la cual provee acceso al Manual de Usuario del Data Explorer.. La interfaz gráfica del DX consta de las siguientes ventanas primarias: . Ventana del Editor de Programación Visual (VPE): Permite la creación y edición de programas visuales y macros.. . Ventana del Panel de Control: Permite ajustar y controlar las variables y parámetros de entrada de las herramientas utilizadas en el programa visual.. . Ventana de Imagen: Muestra la imagen o las imágenes generadas por el programa visual y permite la interacción con dichas imágenes.. . Ventana de Ayuda: Provee acceso al Manual de Usuario del Data Explorer. Es sensible a la posición del cursor, con lo que permite la documentación de los programas visuales.. 36.
(44) C. A P. Í. T U L. O. 2. Modelo de datos El modelo de datos que utiliza el DX esta diseñado para soportar: . Variadas topologías de mallas y tipos de elementos.. . Datos dependientes de la posición o centrados en la celda.. . Datos multidimensionales y multiparamétricos.. . Series de datos (por ejemplo con dependencia temporal).. . Estructuras jerárquicas (particionamiento para paralelismo).. Provee además, objetos de estructura auto-descriptiva. . Tipo de dato (float, byte, etc).. . Forma y rango de los datos (scalar, 2-vector, 3-vector, etc).. . Dimensionalidad de las posiciones (2D, 3D).. . Tipos primitivos (quad, tetrahedrom, etc).. . Tipo de Objeto (grupos, campos).. . Inválidos de posición o conexiones (valores perdidos).. Formato Nativo El formato de fichero nativo del Data Explorer encapsula el modelo de datos DX en disco (o en una salida estándar como resultado de un programa de conversión externa). Este formato de fichero es compresivo y flexible pues puede representar cualquier objeto creado en el Data Explorer. De esa forma, cualquiera de los objetos puede ser exportado en cualquier punto. Un fichero de datos en este formato puede ser importado hacia una sesión del Data Explorer especificando “dx” como el valor del parámetro format en el módulo Import. Ver referencia de usuario de OpenDX.. Un fichero DX consiste en una sección de encabezado, seguida por una sección de datos opcional. La sección de encabezado consiste en una descripción textual de una colección de Objetos. La sección de datos contiene los datos pertenecientes a Objetos de Arreglo en formato texto o binario y es referenciada por la sección de encabezado. Una sección de encabezado puede hacer referencias a objetos y datos en el fichero actual o en otros ficheros.. La Figura 11 muestra los datos empotrados en la sección de encabezado. Otro fichero no puede referirse a los datos en este fichero porque no hay sección de. 37.
(45) C. A P. Í. T U L. O. 2. datos especificada. Sin embargo, las secciones de encabezado en este fichero pueden referirse a secciones de datos en otros ficheros. Este método a veces es más conveniente cuando se crean ficheros de datos con programas simples.. La Figura 12 muestra una sección de encabezado con referencia a una sección de datos en otro fichero. El encabezado se refiere a los datos utilizando el nombre del fichero de datos y una localización de desplazamiento (en bytes desde el inicio de la sección de datos dentro del fichero).. La Figura 13 muestra una sección de encabezado y datos en el mismo fichero. El encabezado se refiere a la sección de datos utilizando un desplazamiento en bytes, relativo al inicio de la sección de datos.. Figura 11 Datos empotrados en la sección de encabezado.. 38.
(46) C. A P. Í. T U L. O. 2. Figura 12 Encabezamiento con referencia a datos en otro fichero.. Figura 13 Encabezamiento y datos en un mismo fichero.. Estas configuraciones pueden ser utilizadas mutuamente en conjunto. Por ejemplo, un fichero puede contener un encabezamiento y datos, y puede referirse a datos tanto en el mismo fichero como en otro fichero. Un fichero puede además, tener sólo un encabezado y referirse a datos en un fichero de sólo datos o en un fichero que contenga encabezado y datos. Esta flexibilidad permite construir un encabezado que apunte a datos en ficheros existentes y permite ver y editar la información del encabezado (si es necesario) utilizando herramientas estándares.. 39.
(47) C. A P. Í. T U L. O. 2. 2.3 Uso de bases de datos relacionales para el almacenamiento de datos científicos. En la actualidad, los Sistemas Gestores de Bases de Datos son utilizados para el almacenamiento de grandes cantidades de información. Por las ventajas que estos brindan (seguridad, acceso concurrente, rapidez de recuperación de datos, indexado de los datos, etc.) se ha extendido su uso a casi cualquier aplicación que necesite almacenar datos.. No obstante, los Sistemas Gestores de Bases de Datos existentes no son apropiados para el almacenamiento y procesamiento de tipos de datos orientados a arreglos como los que soportan las diferentes interfaces de trabajo con datos científicos (ejemplo, netCDF, CDF, HDF).. Los sistemas de bases de datos basados en el modelo relacional, no soportan objetos multidimensionales (arreglos) como una unidad básica de acceso a datos y el hecho de representar arreglos como relaciones hace complicados algunos tipos útiles de accesos a datos. Además de esto, proveen poco soporte para las abstracciones de datos multidimensionales y las herramientas que poseen para el trabajo con sistemas de coordenadas no son lo suficientemente potentes, por lo que se necesita un modelo de datos bastante diferente que pueda facilitar la recuperación, modificación, manipulación matemática y visualización de datos orientados a arreglos.. Relacionado a esto hay un segundo problema con los sistemas de bases de datos de propósito general: su pobre desempeño en arreglos grandes. Las colecciones de imágenes de satélites, salidas (información) pertenecientes a modelos científicos y observaciones a largo plazo del tiempo global, se encuentran más allá de las capacidades que poseen la mayoría de los sistemas de bases de datos de organizar e indexar la información para lograr una recuperación eficiente.. Finalmente, los sistemas de bases de datos de propósito general proveen muchas facilidades que no son necesarias para el análisis, administración y muestra de. 40.
(48) C. A P. Í. T U L. O. 2. datos orientados a arreglos agregando un costo significativo en términos de recursos y rendimiento en el acceso. Por ejemplo, las funciones de actualización, auditorías, formato de reportes y los mecanismos designados para el procesamiento de transacciones son innecesarios para la mayoría de las aplicaciones científicas.. Cualquier fichero de larga escala basado en un Sistema Gestor de Bases de Datos Relacionales (SGBDR) es costoso, requiriendo sistemas de computadoras de alta potencia y buenas conexiones de red. Los archivos creados con SGBDR no son portables (intercambiable) directamente, a menos que se utilicen en ambas partes los mismos tipos de engines de SGBDR, o sea usado un formato basado en texto llano. En general, los SGBDR crean un archivo de gran tamaño debido a la cantidad de sobrecarga que le añaden. Esto último puede apreciarse en la Tabla 6, la cual muestra los resultados en cuanto a tamaño de archivo, acceso a datos y recuperación de datos, de una prueba realizada a un conjunto de datos pertenecientes a mediciones recopiladas por sensores de una autopista de Estados Unidos. En la misma, a partir de un fichero en formato binario que contenía los datos de las mediciones, se creó un conjunto de datos utilizando la biblioteca CDF y otro utilizando un SGBDR, siendo este último casi 75 veces mayor que el segundo y 9 veces mayor que el primero. Tabla 6 Sumario de los resultados de la prueba de almacenamiento y recuperación de los datos.. Tiempo de. Binario. CDF. RDBMS. Descomprimido. (GZIP nivel 4). N/A. 5 minutos. 6 horas. 40 MB. 16.6 MB. 370 MB. N/A. 2 minutos. 2 horas. almacenamiento Tamaño del archivo Tiempo de Recuperación. Sin embargo, muchas aplicaciones generan datos que se almacenan en bases de datos relacionales por eso tiene sentido desarrollar un módulo (o varios) que. 41.
(49) C. A P. Í. T U L. O. 2. permitan trabajar con estos datos desde los sistemas de visualización científica existentes. 2.4 Comparación de los diferentes formatos de datos. Las diferencias entre los formatos CDF, HDF y netCDF están basadas en una descripción de alto nivel correspondiente a cada uno de ellos.. Esta valoración puede variar con el tiempo, dependiendo de próximos lanzamientos de los softwares.. No obstante, la mejor forma de evaluar un paquete es adquiriéndolo y probando si cumple con los requerimientos del usuario. CDF y netCDF El CDF fue diseñado y desarrollado en 1985 por el Centro de Datos de Ciencia Espacial Nacional (NSSDC) en NASA/GSFC. Al principio fue programado en Fortran y sólo estaba disponible para los ambientes VAX/VMS.. El netCDF fue desarrollado unos años más tarde por el Centro Nacional para la Investigación Atmosférica (Nacional Center for Atmospheric Researches NCAR). El modelo de netCDF estaba basado en aquel modelo conceptual CDF, pero proporcionó varios rasgos adicionales (como encuadernaciones con el lenguaje C, portabilidad hacia a varias plataformas, formato de datos independiente de la arquitectura de la máquina, etc.). Hoy ambos modelos y software existentes han madurado considerablemente y son bastante similares en la mayoría de los aspectos, a pesar de diferenciarse de los modos siguientes: . Aunque las interfaces proporcionen realmente la misma funcionalidad básica, presentan diferentes sintaxis. (Ver guías de usuarios para más detalles.). . NetCDF soporta dimensiones con nombres (i.e., TEMP[x, y, ...]) mientras que CDF utiliza el método lógico tradicional (i.e., TEMP[true, true, ...]) de indicar dimensionalidad.. 42.
Figure

Documento similar
Acorde con los criterios previamente enunciados, se propone como problema científico, el cómo utilizar varias técnicas de la visualización científica para realizar una
Se busca analizar las funciones de MongoDB por medio del diseño de un clúster para el almacenamiento de datos distribuidos para la visualización de datos y
En la caracterización realizada se incluyeron los for - matos de almacenamiento, transporte y visualización más relevantes en el dominio de los SIG, específi - camente sobre
Fue dise˜ nado para ser flexible, pudiendo trabajar sobre varios sistemas de almacenamiento (bases de datos relacionales, en memoria, en sistema de archivos, etc´etera) con
Incluye un intérprete de datos RDF, soluciones de almacenamiento en memoria, soluciones de almacenamiento para bases de datos relacionales (mediante un convertidor), así como
En este artículo comentaremos algunas técnicas de extracción de conocimiento sobre bases de datos y de su visualización, centrándonos en bases de datos médicas, debido a que este
Con este módulo de visualización científica se incrementan las potencialidades de Weka para la exploración y análisis de datos iniciales, al adicionarle las técnicas de
En este sentido, una herramienta de visualización de datos de eventos de emergencia que combine funciones de visualización de datos y componentes de captura, almacenamiento y predicción





